From Wikipedia, the free encyclopedia
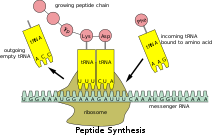
The interaction of tRNA and mRNA in protein synthesis.
| tRNA | |
|---|---|
| Identifiers | |
| Symbol | t |
| Rfam | RF00005 |
| Other data | |
| RNA type | gene, tRNA |
| PDB structures | PDBe 3icq, 1asy, 1asz, 1il2, 2tra, 3tra, 486d, 1fir, 1yfg, 3eph, 3epj, 3epk, 3epl, 1efw, 1c0a, 2ake, 2azx, 2dr2, 1f7u, 1f7v, 3foz, 2hgp, 2j00, 2j02, 2ow8, 2v46, 2v48, 2wdg, 2wdh, 2wdk, 2wdm, 2wh1 |
Transfer RNA (abbreviated tRNA and formerly referred to as sRNA, for soluble RNA[1]) is an adaptor molecule composed of RNA, typically 76 to 90 nucleotides in length (in eukaryotes),[2] that serves as the physical link between the mRNA and the amino acid sequence of proteins. tRNAs genes from Bacteria are typically shorter (mean = 77.6 bp) than tRNAs from Archaea (mean = 83.1 bp) and eukaryotes (mean = 84.7 bp).[3] The mature tRNA follows an opposite pattern with tRNAs from Bacteria being usually longer (median = 77.6 nt) than tRNAs from Archaea (median = 76.8 nt), with eukaryotes exhibiting the shortest mature tRNAs (median = 74.5 nt).[3] Transfer RNA (tRNA) does this by carrying an amino acid to the protein synthesizing machinery of a cell called the ribosome. Complementation of a 3-nucleotide codon in a messenger RNA (mRNA) by a 3-nucleotide anticodon of the tRNA results in protein synthesis based on the mRNA code. As such, tRNAs are a necessary component of translation, the biological synthesis of new proteins in accordance with the genetic code.
Overview[edit]
While the specific nucleotide sequence of an mRNA specifies which amino acids are incorporated into the protein product of the gene from which the mRNA is transcribed, the role of tRNA is to specify which sequence from the genetic code corresponds to which amino acid.[4] The mRNA encodes a protein as a series of contiguous codons, each of which is recognized by a particular tRNA. One end of the tRNA matches the genetic code in a three-nucleotide sequence called the anticodon. The anticodon forms three complementary base pairs with a codon in mRNA during protein biosynthesis.
On the other end of the tRNA is a covalent attachment to the amino acid that corresponds to the anticodon sequence. Each type of tRNA molecule can be attached to only one type of amino acid, so each organism has many types of tRNA. Because the genetic code contains multiple codons that specify the same amino acid, there are several tRNA molecules bearing different anticodons which carry the same amino acid.
The covalent attachment to the tRNA 3’ end is catalysed by enzymes called aminoacyl tRNA synthetases. During protein synthesis, tRNAs with attached amino acids are delivered to the ribosome by proteins called elongation factors, which aid in association of the tRNA with the ribosome, synthesis of the new polypeptide, and translocation (movement) of the ribosome along the mRNA. If the tRNA’s anticodon matches the mRNA, another tRNA already bound to the ribosome transfers the growing polypeptide chain from its 3’ end to the amino acid attached to the 3’ end of the newly delivered tRNA, a reaction catalysed by the ribosome. A large number of the individual nucleotides in a tRNA molecule may be chemically modified, often by methylation or deamidation. These unusual bases sometimes affect the tRNA’s interaction with ribosomes and sometimes occur in the anticodon to alter base-pairing properties.[5]
Structure[edit]
Secondary cloverleaf structure of tRNA
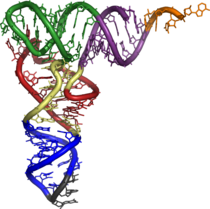
Tertiary structure of tRNA. CCA tail in yellow, Acceptor stem in purple, Variable loop in orange, D arm in red, Anticodon arm in blue with Anticodon in black, T arm in green.
3D animated GIF showing the structure of phenylalanine-tRNA from yeast (PDB ID 1ehz). White lines indicate base pairing by hydrogen bonds. In the orientation shown, the acceptor stem is on top and the anticodon on the bottom[6]
The structure of tRNA can be decomposed into its primary structure, its secondary structure (usually visualized as the cloverleaf structure), and its tertiary structure[7] (all tRNAs have a similar L-shaped 3D structure that allows them to fit into the P and A sites of the ribosome). The cloverleaf structure becomes the 3D L-shaped structure through coaxial stacking of the helices, which is a common RNA tertiary structure motif. The lengths of each arm, as well as the loop ‘diameter’, in a tRNA molecule vary from species to species.[7][8]
The tRNA structure consists of the following:
- The acceptor stem is a 7- to 9-base pair (bp) stem made by the base pairing of the 5′-terminal nucleotide with the 3′-terminal nucleotide (which contains the CCA 3′-terminal group used to attach the amino acid). In general, such 3′-terminal tRNA-like structures are referred to as ‘genomic tags’. The acceptor stem may contain non-Watson-Crick base pairs.[7][9]
- The CCA tail is a cytosine-cytosine-adenine sequence at the 3′ end of the tRNA molecule. The amino acid loaded onto the tRNA by aminoacyl tRNA synthetases, to form aminoacyl-tRNA, is covalently bonded to the 3′-hydroxyl group on the CCA tail.[10] This sequence is important for the recognition of tRNA by enzymes and critical in translation.[11][12] In prokaryotes, the CCA sequence is transcribed in some tRNA sequences. In most prokaryotic tRNAs and eukaryotic tRNAs, the CCA sequence is added during processing and therefore does not appear in the tRNA gene.[13]
- The D loop is a 4- to 6-bp stem ending in a loop that often contains dihydrouridine.[7]
- The anticodon loop is a 5-bp stem whose loop contains the anticodon.[7] The tRNA 5′-to-3′ primary structure contains the anticodon but in reverse order, since 3′-to-5′ directionality is required to read the mRNA from 5′-to-3′.
- The ΨU loop is named so because of the characteristic presence of the unusual base ΨU in the loop, where Ψ is pseudo uridine, a modified uridine. The modified base is often found within the sequence 5′ -TΨUCG-3′.[7]
- The variable loop sits between the anticodon loop and the ΨU loop and, as its name implies, varies in size from 3 to 21 bases.[14]
Anticodon[edit]
An anticodon[15] is a unit of three nucleotides corresponding to the three bases of an mRNA codon. Each tRNA has a distinct anticodon triplet sequence that can form 3 complementary base pairs to one or more codons for an amino acid. Some anticodons pair with more than one codon due to wobble base pairing. Frequently, the first nucleotide of the anticodon is one not found on mRNA: inosine, which can hydrogen bond to more than one base in the corresponding codon position.[5]: 29.3.9 In genetic code, it is common for a single amino acid to be specified by all four third-position possibilities, or at least by both pyrimidines and purines; for example, the amino acid glycine is coded for by the codon sequences GGU, GGC, GGA, and GGG. Other modified nucleotides may also appear at the first anticodon position—sometimes known as the «wobble position»—resulting in subtle changes to the genetic code, as for example in mitochondria.[16]
Per cell, 61 tRNA types are required to provide one-to-one correspondence between tRNA molecules and codons that specify amino acids, as there are 61 sense codons of the standard genetic code. However, many cells have under 61 types of tRNAs because the wobble base is capable of binding to several, though not necessarily all, of the codons that specify a particular amino acid. At least 31 tRNAs are required to translate, unambiguously, all 61 sense codons.[4][17]
Aminoacylation[edit]
Aminoacylation is the process of adding an aminoacyl group to a compound. It covalently links an amino acid to the CCA 3′ end of a tRNA molecule.
Each tRNA is aminoacylated (or charged) with a specific amino acid by an aminoacyl tRNA synthetase. There is normally a single aminoacyl tRNA synthetase for each amino acid, despite the fact that there can be more than one tRNA, and more than one anticodon for an amino acid. Recognition of the appropriate tRNA by the synthetases is not mediated solely by the anticodon, and the acceptor stem often plays a prominent role.[18]
Reaction:
- amino acid + ATP → aminoacyl-AMP + PPi
- aminoacyl-AMP + tRNA → aminoacyl-tRNA + AMP
Certain organisms can have one or more aminophosphate-tRNA synthetases missing. This leads to charging of the tRNA by a chemically related amino acid, and by use of an enzyme or enzymes, the tRNA is modified to be correctly charged. For example, Helicobacter pylori has glutaminyl tRNA synthetase missing. Thus, glutamate tRNA synthetase charges tRNA-glutamine(tRNA-Gln) with glutamate. An amidotransferase then converts the acid side chain of the glutamate to the amide, forming the correctly charged gln-tRNA-Gln.
Interference with aminoacylation may be useful as an approach to treating some diseases: cancerous cells may be relatively vulnerable to disturbed aminoacylation compared to healthy cells. The protein synthesis associated with cancer and viral biology is often very dependent on specific tRNA molecules. For instance, for liver cancer charging tRNA-Lys-CUU with lysine sustains liver cancer cell growth and metastasis, whereas healthy cells have a much lower dependence on this tRNA to support cellular physiology.[19] Similarly, hepatitis E virus requires a tRNA landscape that substantially differs from that associated with uninfected cells.[20] Hence, inhibition of aminoacylation of specific tRNA species is considered a promising novel avenue for the rational treatment of a plethora of diseases.
Binding to ribosome[edit]
The range of conformations adopted by tRNA as it transits the A/T through P/E sites on the ribosome. The Protein Data Bank (PDB) codes for the structural models used as end points of the animation are given. Both tRNAs are modeled as phenylalanine-specific tRNA from Escherichia coli, with the A/T tRNA as a homology model of the deposited coordinates. Color coding as shown for tRNA tertiary structure. Adapted from.[21]
The ribosome has three binding sites for tRNA molecules that span the space between the two ribosomal subunits: the A (aminoacyl),[22] P (peptidyl), and E (exit) sites. In addition, the ribosome has two other sites for tRNA binding that are used during mRNA decoding or during the initiation of protein synthesis. These are the T site (named elongation factor Tu) and I site (initiation).[23][24] By convention, the tRNA binding sites are denoted with the site on the small ribosomal subunit listed first and the site on the large ribosomal subunit listed second. For example, the A site is often written A/A, the P site, P/P, and the E site, E/E.[23] The binding proteins like L27, L2, L14, L15, L16 at the A- and P- sites have been determined by affinity labeling by A. P. Czernilofsky et al. (Proc. Natl. Acad. Sci, USA, pp. 230–234, 1974).
Once translation initiation is complete, the first aminoacyl tRNA is located in the P/P site, ready for the elongation cycle described below. During translation elongation, tRNA first binds to the ribosome as part of a complex with elongation factor Tu (EF-Tu) or its eukaryotic (eEF-1) or archaeal counterpart. This initial tRNA binding site is called the A/T site. In the A/T site, the A-site half resides in the small ribosomal subunit where the mRNA decoding site is located. The mRNA decoding site is where the mRNA codon is read out during translation. The T-site half resides mainly on the large ribosomal subunit where EF-Tu or eEF-1 interacts with the ribosome. Once mRNA decoding is complete, the aminoacyl-tRNA is bound in the A/A site and is ready for the next peptide bond[25] to be formed to its attached amino acid. The peptidyl-tRNA, which transfers the growing polypeptide to the aminoacyl-tRNA bound in the A/A site, is bound in the P/P site. Once the peptide bond is formed, the tRNA in the P/P site is acylated, or has a free 3’ end, and the tRNA in the A/A site dissociates the growing polypeptide chain. To allow for the next elongation cycle, the tRNAs then move through hybrid A/P and P/E binding sites, before completing the cycle and residing in the P/P and E/E sites. Once the A/A and P/P tRNAs have moved to the P/P and E/E sites, the mRNA has also moved over by one codon and the A/T site is vacant, ready for the next round of mRNA decoding. The tRNA bound in the E/E site then leaves the ribosome.
The P/I site is actually the first to bind to aminoacyl tRNA, which is delivered by an initiation factor called IF2 in bacteria.[24] However, the existence of the P/I site in eukaryotic or archaeal ribosomes has not yet been confirmed. The P-site protein L27 has been determined by affinity labeling by E. Collatz and A. P. Czernilofsky (FEBS Lett., Vol. 63, pp. 283–286, 1976).
tRNA genes[edit]
Organisms vary in the number of tRNA genes in their genome. For example, the nematode worm C. elegans, a commonly used model organism in genetics studies, has 29,647 [26] genes in its nuclear genome, of which 620 code for tRNA.[27][28] The budding yeast Saccharomyces cerevisiae has 275 tRNA genes in its genome. The number of tRNA genes per genome can vary widely, with bacterial species from groups such as Fusobacteria and Tenericutes having around 30 genes per genome while complex eukaryotic genomes such as the zebrafish (Danio rerio) can bear more than 10 thousand tRNA genes.[3]
In the human genome, which, according to January 2013 estimates, has about 20,848 protein coding genes [29] in total, there are 497 nuclear genes encoding cytoplasmic tRNA molecules, and 324 tRNA-derived pseudogenes—tRNA genes thought to be no longer functional[30] (although pseudo tRNAs have been shown to be involved in antibiotic resistance in bacteria).[31] As with all eukaryotes, there are 22 mitochondrial tRNA genes[32][full citation needed] in humans. Mutations in some of these genes have been associated with severe diseases like the MELAS syndrome. Regions in nuclear chromosomes, very similar in sequence to mitochondrial tRNA genes, have also been identified (tRNA-lookalikes).[33] These tRNA-lookalikes are also considered part of the nuclear mitochondrial DNA (genes transferred from the mitochondria to the nucleus).[33][34] The phenomenon of multiple nuclear copies of mitochondrial tRNA (tRNA-lookalikes) has been observed in many higher organisms from human to the opossum[35] suggesting the possibility that the lookalikes are functional.
Cytoplasmic tRNA genes can be grouped into 49 families according to their anticodon features. These genes are found on all chromosomes, except the 22 and Y chromosome. High clustering on 6p is observed (140 tRNA genes), as well on 1 chromosome.[30]
The HGNC, in collaboration with the Genomic tRNA Database (GtRNAdb) and experts in the field, has approved unique names for human genes that encode tRNAs.
Evolution[edit]
The top half of tRNA (consisting of the T arm and the acceptor stem with 5′-terminal phosphate group and 3′-terminal CCA group) and the bottom half (consisting of the D arm and the anticodon arm) are independent units in structure as well as in function. The top half may have evolved first including the 3′-terminal genomic tag which originally may have marked tRNA-like molecules for replication in early RNA world. The bottom half may have evolved later as an expansion, e.g. as protein synthesis started in RNA world and turned it into a ribonucleoprotein world (RNP world). This proposed scenario is called genomic tag hypothesis. In fact, tRNA and tRNA-like aggregates have an important catalytic influence (i.e., as ribozymes) on replication still today. These roles may be regarded as ‘molecular (or chemical) fossils’ of RNA world.[36]
Genomic tRNA content is a differentiating feature of genomes among biological domains of life: Archaea present the simplest situation in terms of genomic tRNA content with a uniform number of gene copies, Bacteria have an intermediate situation and Eukarya present the most complex situation.[37] Eukarya present not only more tRNA gene content than the other two kingdoms but also a high variation in gene copy number among different isoacceptors, and this complexity seem to be due to duplications of tRNA genes and changes in anticodon specificity[citation needed].
Evolution of the tRNA gene copy number across different species has been linked to the appearance of specific tRNA modification enzymes (uridine methyltransferases in Bacteria, and adenosine deaminases in Eukarya), which increase the decoding capacity of a given tRNA.[37] As an example, tRNAAla encodes four different tRNA isoacceptors (AGC, UGC, GGC and CGC). In Eukarya, AGC isoacceptors are extremely enriched in gene copy number in comparison to the rest of isoacceptors, and this has been correlated with its A-to-I modification of its wobble base. This same trend has been shown for most amino acids of eukaryal species. Indeed, the effect of these two tRNA modifications is also seen in codon usage bias. Highly expressed genes seem to be enriched in codons that are exclusively using codons that will be decoded by these modified tRNAs, which suggests a possible role of these codons—and consequently of these tRNA modifications—in translation efficiency.[37]
It is important to note that many species have lost specific tRNAs during evolution. For instance, both mammals and birds lack the same 14 out of the possible 64 tRNA genes, but other life forms contain these tRNAs.[38] For translating codons for which an exactly pairing tRNA is missing, organisms resort to a strategy called wobbling, in which imperfectly matched tRNA/mRNA pairs still give rise to translation, although this strategy also increases to propensity for translation errors.[39] The reasons why tRNA genes have been lost during evolution remains under debate but may relate improving resistance to viral infection.[40] Because nucleotide triplets can present more combinations than there are amino acids and associated tRNAs, there is redundancy in the genetic code, and several different 3-nucleotide codons can express the same amino acid. This codon bias is what necessitates codon optimization.
tRNA-derived fragments[edit]
tRNA-derived fragments (or tRFs) are short molecules that emerge after cleavage of the mature tRNAs or the precursor transcript.[41][42][43][44] Both cytoplasmic and mitochondrial tRNAs can produce fragments.[45] There are at least four structural types of tRFs believed to originate from mature tRNAs, including the relatively long tRNA halves and short 5’-tRFs, 3’-tRFs and i-tRFs.[41][45][46] The precursor tRNA can be cleaved to produce molecules from the 5’ leader or 3’ trail sequences. Cleavage enzymes include Angiogenin, Dicer, RNase Z and RNase P.[41][42] Especially in the case of Angiogenin, the tRFs have a characteristically unusual cyclic phosphate at their 3’ end and a hydroxyl group at the 5’ end.[47] tRFs appear to play a role in RNA interference, specifically in the suppression of retroviruses and retrotransposons that use tRNA as a primer for replication. Half-tRNAs cleaved by angiogenin are also known as tiRNAs. The biogenesis of smaller fragments, including those that function as piRNAs, are less understood.[48]
tRFs have multiple dependencies and roles; such as exhibiting significant changes between sexes, among races and disease status.[45][49][50] Functionally, they can be loaded on Ago and act through RNAi pathways,[43][46][51] participate in the formation of stress granules,[52] displace mRNAs from RNA-binding proteins[53] or inhibit translation.[54] At the system or the organismal level, the four types of tRFs have a diverse spectrum of activities. Functionally, tRFs are associated with viral infection,[55] cancer,[46] cell proliferation [47] and also with epigenetic transgenerational regulation of metabolism.[56]
tRFs are not restricted to humans and have been shown to exist in multiple organisms.[46][57][58][59]
Two online tools are available for those wishing to learn more about tRFs: the framework for the interactive exploration of mitochondrial and nuclear tRNA fragments (MINTbase)[60][61] and the relational database of Transfer RNA related Fragments (tRFdb).[62] MINTbase also provides a naming scheme for the naming of tRFs called tRF-license plates (or MINTcodes) that is genome independent; the scheme compresses an RNA sequence into a shorter string.
Engineered tRNAs[edit]
Artificial suppressor elongator tRNAs are used to incorporate unnatural amino acids at nonsense codons placed in the coding sequence of a gene. Engineered initiator tRNAs (tRNAfMet2 with CUA anticodon encoded by metY gene) have been used to initiate translation at the amber stop codon UAG. This type of engineered tRNA is called a nonsense suppressor tRNA because it suppresses the translation stop signal that normally occurs at UAG codons. The amber initiator tRNA inserts methionine[63] and glutamine[64] at UAG codons preceded by a strong Shine-Dalgarno sequence. An investigation of the amber initiator tRNA showed that it was orthogonal to the regular AUG start codon showing no detectable off-target translation initiation events in a genomically recoded E. coli strain.[63]
tRNA biogenesis[edit]
In eukaryotic cells, tRNAs are transcribed by RNA polymerase III as pre-tRNAs in the nucleus.[65]
RNA polymerase III recognizes two highly conserved downstream promoter sequences: the 5′ intragenic control region (5′-ICR, D-control region, or A box), and the 3′-ICR (T-control region or B box) inside tRNA genes.[2][66][67]
The first promoter begins at +8 of mature tRNAs and the second promoter is located 30–60 nucleotides downstream of the first promoter. The transcription terminates after a stretch of four or more thymidines.[2][67]
Bulge-helix-bulge motif of a tRNA intron
Pre-tRNAs undergo extensive modifications inside the nucleus. Some pre-tRNAs contain introns that are spliced, or cut, to form the functional tRNA molecule;[68] in bacteria these self-splice, whereas in eukaryotes and archaea they are removed by tRNA-splicing endonucleases.[69] Eukaryotic pre-tRNA contains bulge-helix-bulge (BHB) structure motif that is important for recognition and precise splicing of tRNA intron by endonucleases.[70] This motif position and structure are evolutionarily conserved. However, some organisms, such as unicellular algae have a non-canonical position of BHB-motif as well as 5′- and 3′-ends of the spliced intron sequence.[70]
The 5′ sequence is removed by RNase P,[71] whereas the 3′ end is removed by the tRNase Z enzyme.[72]
A notable exception is in the archaeon Nanoarchaeum equitans, which does not possess an RNase P enzyme and has a promoter placed such that transcription starts at the 5′ end of the mature tRNA.[73]
The non-templated 3′ CCA tail is added by a nucleotidyl transferase.[74]
Before tRNAs are exported into the cytoplasm by Los1/Xpo-t,[75][76] tRNAs are aminoacylated.[77]
The order of the processing events is not conserved.
For example, in yeast, the splicing is not carried out in the nucleus but at the cytoplasmic side of mitochondrial membranes.[78]
Nonetheless, In March 2021, researchers reported evidence suggesting that a preliminary form of transfer RNA could have been a replicator molecule in the very early development of life, or abiogenesis.[79][80]
History[edit]
The existence of tRNA was first hypothesized by Francis Crick as the «adaptor hypothesis» based on the assumption that there must exist an adapter molecule capable of mediating the translation of the RNA alphabet into the protein alphabet. Paul C Zamecnik and Mahlon Hoagland discovered tRNA [81] Significant research on structure was conducted in the early 1960s by Alex Rich and Donald Caspar, two researchers in Boston, the Jacques Fresco group in Princeton University and a United Kingdom group at King’s College London.[82] In 1965, Robert W. Holley of Cornell University reported the primary structure and suggested three secondary structures.[83] tRNA was first crystallized in Madison, Wisconsin, by Robert M. Bock.[84] The cloverleaf structure was ascertained by several other studies in the following years[85] and was finally confirmed using X-ray crystallography studies in 1974. Two independent groups, Kim Sung-Hou working under Alexander Rich and a British group headed by Aaron Klug, published the same crystallography findings within a year.[86][87]
See also[edit]
- Cloverleaf model of tRNA
- Kim Sung-Hou
- Kissing stem-loop
- mRNA
- non-coding RNA and introns
- Slippery sequence
- tmRNA
- Transfer RNA-like structures
- Translation
- tRNADB
- Wobble hypothesis
- Aminoacyl-tRNA
References[edit]
- ^ Plescia OJ, Palczuk NC, Cora-Figueroa E, Mukherjee A, Braun W (October 1965). «Production of antibodies to soluble RNA (sRNA)». Proceedings of the National Academy of Sciences of the United States of America. 54 (4): 1281–1285. Bibcode:1965PNAS…54.1281P. doi:10.1073/pnas.54.4.1281. PMC 219862. PMID 5219832.
- ^ a b c Sharp SJ, Schaack J, Cooley L, Burke DJ, Söll D (1985). «Structure and transcription of eukaryotic tRNA genes». CRC Critical Reviews in Biochemistry. 19 (2): 107–144. doi:10.3109/10409238509082541. PMID 3905254.
- ^ a b c Santos, Fenícia Brito; Del-Bem, Luiz-Eduardo (January 2023). «The Evolution of tRNA Copy Number and Repertoire in Cellular Life». Genes. 14 (1): 27. doi:10.3390/genes14010027. ISSN 2073-4425. PMC 9858662. PMID 36672768.
- ^ a b Crick FH (December 1968). «The origin of the genetic code». Journal of Molecular Biology. 38 (3): 367–379. doi:10.1016/0022-2836(68)90392-6. PMID 4887876. S2CID 4144681.
- ^ a b Stryer L, Berg JM, Tymoczko JL (2002). Biochemistry (5th ed.). San Francisco: W. H. Freeman. ISBN 978-0-7167-4955-4.
- ^ «Transfer RNA (tRNA)». Proteopedia.org. Retrieved 7 November 2018.
- ^ a b c d e f Itoh Y, Sekine S, Suetsugu S, Yokoyama S (July 2013). «Tertiary structure of bacterial serenocysteine tRNA». Nucleic Acids Research. 41 (13): 6729–6738. doi:10.1093/nar/gkt321. PMC 3711452. PMID 23649835.
- ^ Goodenbour JM, Pan T (29 October 2006). «Diversity of tRNA genes in eukaryotes». Nucleic Acids Research. 34 (21): 6137–6146. doi:10.1093/nar/gkl725. PMC 1693877. PMID 17088292.
- ^
- ^ Ibba M, Soll D (June 2000). «Aminoacyl-tRNA synthesis». Annual Review of Biochemistry. 69 (1): 617–650. doi:10.1146/annurev.biochem.69.1.617. PMID 10966471.
- ^ Sprinzl M, Cramer F (1979). «The -C-C-A end of tRNA and its role in protein biosynthesis». Progress in Nucleic Acid Research and Molecular Biology. 22: 1–69. doi:10.1016/s0079-6603(08)60798-9. ISBN 978-0-12-540022-0. PMID 392600.
- ^ Green R, Noller HF (1997). «Ribosomes and translation». Annual Review of Biochemistry. 66: 679–716. doi:10.1146/annurev.biochem.66.1.679. PMID 9242921.
- ^ Aebi M, Kirchner G, Chen JY, Vijayraghavan U, Jacobson A, Martin NC, Abelson J, et al. (September 1990). «Isolation of a temperature-sensitive mutant with an altered tRNA nucleotidyltransferase and cloning of the gene encoding tRNA nucleotidyltransferase in the yeast Saccharomyces cerevisiae». The Journal of Biological Chemistry. 265 (27): 16216–16220. doi:10.1016/S0021-9258(17)46210-7. PMID 2204621.
- ^ Prabhakar A, Krahn N, Zhang J, Vargas-Rodriguez O, Krupkin M, Fu Z, Acosta-Reyes FJ, Ge X, Choi J, Crnković A, Ehrenberg M, Puglisi EV, Söll D, Puglisi J (Jul 2022). «Uncovering translation roadblocks during the development of a synthetic tRNA». Nucleic Acids Res. online ahead of print (18): 10201–10211. doi:10.1093/nar/gkac576. PMC 9561287. PMID 35882385.
- ^ Felsenfeld G, Cantoni GL (May 1964). «Use of thermal denaturation studies to investigate the base sequence of yeast serine sRNA». Proceedings of the National Academy of Sciences of the United States of America. 51 (5): 818–826. Bibcode:1964PNAS…51..818F. doi:10.1073/pnas.51.5.818. PMC 300168. PMID 14172997.
- ^ Suzuki T, Suzuki T (June 2014). «A complete landscape of post-transcriptional modifications in mammalian mitochondrial tRNAs». Nucleic Acids Research. 42 (11): 7346–7357. doi:10.1093/nar/gku390. PMC 4066797. PMID 24831542.
- ^ Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J. (2004). Molecular Cell Biology. WH Freeman: New York. 5th ed.ISBN 978-0716743668[page needed]
- ^ Schimmel P, Giegé R, Moras D, Yokoyama S (October 1993). «An operational RNA code for amino acids and possible relationship to genetic code». Proceedings of the National Academy of Sciences of the United States of America. 90 (19): 8763–8768. Bibcode:1993PNAS…90.8763S. doi:10.1073/pnas.90.19.8763. PMC 47440. PMID 7692438.
- ^ Zhang R, Noordam L, Ou X, Ma B, Li Y, Das P, Shi S, Liu J, Wang L, Li P, Verstegen MM, Reddy DS, van der Laan LJ, Peppelenbosch MP, Kwekkeboom J, Smits R, Pan Q (January 2021). «The biological process of lysine-tRNA charging is therapeutically targetable in liver cancer». Liver Int. 41 (1): 206–219. doi:10.1111/liv.14692. PMC 7820958. PMID 33084231.
- ^ Ou X, Ma B, Zhang R, Miao Z, Cheng A, Peppelenbosch MP, Pan Q (June 2020). «A simplified qPCR method revealing tRNAome remodeling upon infection by genotype 3 hepatitis E virus». FEBS Letters. 594 (12): 2005–2015. doi:10.1002/1873-3468.13764. PMID 32133647.
- ^ Dunkle JA, Wang L, Feldman MB, Pulk A, Chen VB, Kapral GJ, Noeske J, Richardson JS, Blanchard SC, Cate JH (May 2011). «Structures of the bacterial ribosome in classical and hybrid states of tRNA binding». Science. 332 (6032): 981–984. Bibcode:2011Sci…332..981D. doi:10.1126/science.1202692. PMC 3176341. PMID 21596992.
- ^ Konevega AL, Soboleva NG, Makhno VI, Semenkov YP, Wintermeyer W, Rodnina MV, Katunin VI (January 2004). «Purine bases at position 37 of tRNA stabilize codon-anticodon interaction in the ribosomal A site by stacking and Mg2+-dependent interactions». RNA. 10 (1): 90–101. doi:10.1261/rna.5142404. PMC 1370521. PMID 14681588.
- ^ a b Agirrezabala X, Frank J (August 2009). «Elongation in translation as a dynamic interaction among the ribosome, tRNA, and elongation factors EF-G and EF-Tu». Quarterly Reviews of Biophysics. 42 (3): 159–200. doi:10.1017/S0033583509990060. PMC 2832932. PMID 20025795.
- ^ a b Allen GS, Zavialov A, Gursky R, Ehrenberg M, Frank J (June 2005). «The cryo-EM structure of a translation initiation complex from Escherichia coli». Cell. 121 (5): 703–712. doi:10.1016/j.cell.2005.03.023. PMID 15935757. S2CID 16146867.
- ^ Tirumalai MR, Rivas M, Tran Q, Fox GE (November 2021). «The Peptidyl Transferase Center: a Window to the Past». Microbiol Mol Biol Rev. 85 (4): e0010421. doi:10.1128/MMBR.00104-21. PMC 8579967. PMID 34756086.
- ^ WormBase web site, http://www.wormbase.org Archived 2017-04-20 at the Wayback Machine, release WS187, date 25-Jan-2008.
- ^ Spieth J, Lawson D (January 2006). «Overview of gene structure». WormBook: 1–10. doi:10.1895/wormbook.1.65.1. PMC 4781370. PMID 18023127.
- ^ Hartwell LH, Hood L, Goldberg ML, Reynolds AE, Silver LM, Veres RC. (2004). Genetics: From Genes to Genomes 2nd ed. McGraw-Hill: New York. p. 264.
- ^ Ensembl release 70 — Jan 2013 http://www.ensembl.org/Homo_sapiens/Info/StatsTable?db=core Archived 2013-12-15 at the Wayback Machine
- ^ a b Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, et al. (International Human Genome Sequencing Consortium) (February 2001). «Initial sequencing and analysis of the human genome» (PDF). Nature. 409 (6822): 860–921. Bibcode:2001Natur.409..860L. doi:10.1038/35057062. PMID 11237011.
- ^ Rogers TE, Ataide SF, Dare K, Katz A, Seveau S, Roy H, Ibba M (2012). «A pseudo-tRNA modulates antibiotic resistance in Bacillus cereus». PLOS ONE. 7 (7): e41248. Bibcode:2012PLoSO…741248R. doi:10.1371/journal.pone.0041248. PMC 3399842. PMID 22815980.
- ^ Ibid. p. 529.
- ^ a b Telonis AG, Loher P, Kirino Y, Rigoutsos I (2014). «Nuclear and mitochondrial tRNA-lookalikes in the human genome». Frontiers in Genetics. 5: 344. doi:10.3389/fgene.2014.00344. PMC 4189335. PMID 25339973.
- ^ Ramos A, Barbena E, Mateiu L, del Mar González M, Mairal Q, Lima M, Montiel R, Aluja MP, Santos C, et al. (November 2011). «Nuclear insertions of mitochondrial origin: Database updating and usefulness in cancer studies». Mitochondrion. 11 (6): 946–953. doi:10.1016/j.mito.2011.08.009. PMID 21907832.
- ^ Telonis AG, Kirino Y, Rigoutsos I (2015). «Mitochondrial tRNA-lookalikes in nuclear chromosomes: Could they be functional?». RNA Biol. 12 (4): 375–380. doi:10.1080/15476286.2015.1017239. PMC 4615777. PMID 25849196.
- ^ Nancy Maizels and Alan M. Weiner: The Genomic Tag Hypothesis – What Molecular Fossils Tell Us about the Evolution of tRNA, in: The RNA World, Second Edition. 1999 Cold Spring Harbor Laboratory Press ISBN 978-0-87969-561-3/99, PDF
- ^ a b c Novoa EM, Pavon-Eternod M, Pan T, Ribas de Pouplana L (March 2012). «A role for tRNA modifications in genome structure and codon usage». Cell. 149 (1): 202–213. doi:10.1016/j.cell.2012.01.050. PMID 22464330. S2CID 16487609.
- ^ Ou X, Peng W, Yang Z, Cao J, Wang M, Peppelenbosch MP, Pan Q, Cheng A (November 2020). «Evolutionarily missing and conserved tRNA genes in human and avian». Infect. Genet. Evol. 85: 104460. doi:10.1016/j.meegid.2020.104460. PMID 32679345.
- ^ Ou X, Cao J, Cheng A, Peppelenbosch MP, Pan Q (March 2019). «Errors in translational decoding: tRNA wobbling or misincorporation?». PLOS Genetics. 15 (3): 2979–2986. doi:10.1371/journal.pgen.1008017. PMC 3158919. PMID 21930591.
- ^ Ou X, Wang M, Mao S, Cao J, Cheng A, Zhu D, Chen S, Jia R, Liu M, Yang Q, Wu Y, Zhao X, Zhang S, Liu Y, Yu Y, Zhang L, Chen X, Peppelenbosch MP, Pan Q (July 2018). «Incompatible Translation Drives a Convergent Evolution and Viral Attenuation During the Development of Live Attenuated Vaccine». Front. Cell. Infect. Microbiol. 8: 249. doi:10.3389/fcimb.2018.00249. PMC 6058041. PMID 30073153.
- ^ a b c Gebetsberger J, Polacek N (December 2013). «Slicing tRNAs to boost functional ncRNA diversity». RNA Biology. 10 (12): 1798–1806. doi:10.4161/rna.27177. PMC 3917982. PMID 24351723.
- ^ a b Shigematsu M, Honda S, Kirino Y (2014). «Transfer RNA as a source of small functional RNA». Journal of Molecular Biology and Molecular Imaging. 1 (2): 8. PMC 4572697. PMID 26389128.
- ^ a b Sobala A, Hutvagner G (2011). «Transfer RNA-derived fragments: origins, processing, and functions» (PDF). Wiley Interdisciplinary Reviews: RNA. 2 (6): 853–862. doi:10.1002/wrna.96. hdl:10453/18187. PMID 21976287. S2CID 206554146.
- ^ Keam SP, Hutvagner G (November 2015). «tRNA-Derived Fragments (tRFs): Emerging New Roles for an Ancient RNA in the Regulation of Gene Expression». Life. 5 (4): 1638–1651. doi:10.3390/life5041638. PMC 4695841. PMID 26703738.
- ^ a b c Telonis AG, Loher P, Honda S, Jing Y, Palazzo J, Kirino Y, Rigoutsos I (July 2015). «Dissecting tRNA-derived fragment complexities using personalized transcriptomes reveals novel fragment classes and unexpected dependencies». Oncotarget. 6 (28): 24797–822. doi:10.18632/oncotarget.4695. PMC 4694795. PMID 26325506.
- ^ a b c d Kumar P, Anaya J, Mudunuri SB, Dutta A (October 2014). «Meta-analysis of tRNA derived RNA fragments reveals that they are evolutionarily conserved and associate with AGO proteins to recognize specific RNA targets». BMC Biology. 12: 78. doi:10.1186/s12915-014-0078-0. PMC 4203973. PMID 25270025.
- ^ a b Honda S, Loher P, Shigematsu M, Palazzo JP, Suzuki R, Imoto I, Rigoutsos I, Kirino Y (July 2015). «Sex hormone-dependent tRNA halves enhance cell proliferation in breast and prostate cancers». Proceedings of the National Academy of Sciences of the United States of America. 112 (29): E3816–E3825. Bibcode:2015PNAS..112E3816H. doi:10.1073/pnas.1510077112. PMC 4517238. PMID 26124144.
- ^ Schorn, AJ; Martienssen, R (October 2018). «Tie-Break: Host and Retrotransposons Play tRNA». Trends in Cell Biology. 28 (10): 793–806. doi:10.1016/j.tcb.2018.05.006. PMC 6520983. PMID 29934075.
- ^ Telonis AG, Rigoutsos I (March 2018). «Race Disparities in the Contribution of miRNA Isoforms and tRNA-Derived Fragments to Triple-Negative Breast Cancer». Cancer Res. 78 (5): 1140–54. doi:10.1158/0008-5472.CAN-17-1947. PMC 5935570. PMID 29229607.
- ^ Telonis AG, Loher P, Magee R, Pliatsika V, Londin E, Kirino Y, Rigoutsos I (Jun 2019). «tRNA Fragments Show Intertwining with mRNAs of Specific Repeat Content and Have Links to Disparities». Cancer Res. 79 (12): 3034–49. doi:10.1158/0008-5472.CAN-19-0789. PMC 6571059. PMID 30996049.
- ^ Shigematsu M, Kirino Y (2015). «tRNA-Derived Short Non-coding RNA as Interacting Partners of Argonaute Proteins». Gene Regulation and Systems Biology. 9: 27–33. doi:10.4137/GRSB.S29411. PMC 4567038. PMID 26401098.
- ^ Emara MM, Ivanov P, Hickman T, Dawra N, Tisdale S, Kedersha N, Hu GF, Anderson P (April 2010). «Angiogenin-induced tRNA-derived stress-induced RNAs promote stress-induced stress granule assembly». The Journal of Biological Chemistry. 285 (14): 10959–10968. doi:10.1074/jbc.M109.077560. PMC 2856301. PMID 20129916.
- ^ Goodarzi H, Liu X, Nguyen HC, Zhang S, Fish L, Tavazoie SF (May 2015). «Endogenous tRNA-Derived Fragments Suppress Breast Cancer Progression via YBX1 Displacement». Cell. 161 (4): 790–802. doi:10.1016/j.cell.2015.02.053. PMC 4457382. PMID 25957686.
- ^ Ivanov P, Emara MM, Villen J, Gygi SP, Anderson P (August 2011). «Angiogenin-induced tRNA fragments inhibit translation initiation». Molecular Cell. 43 (4): 613–623. doi:10.1016/j.molcel.2011.06.022. PMC 3160621. PMID 21855800.
- ^ Selitsky SR, Baran-Gale J, Honda M, Yamane D, Masaki T, Fannin EE, Guerra B, Shirasaki T, Shimakami T, Kaneko S, Lanford RE, Lemon SM, Sethupathy P (January 2015). «Small tRNA-derived RNAs are increased and more abundant than microRNAs in chronic hepatitis B and C». Scientific Reports. 5: 7675. Bibcode:2015NatSR…5E7675S. doi:10.1038/srep07675. PMC 4286764. PMID 25567797.
- ^ Sharma U, Conine CC, Shea JM, Boskovic A, Derr AG, Bing XY, Belleannee C, Kucukural A, Serra RW, Sun F, Song L, Carone BR, Ricci EP, Li XZ, Fauquier L, Moore MJ, Sullivan R, Mello CC, Garber M, Rando OJ (January 2016). «Biogenesis and function of tRNA fragments during sperm maturation and fertilization in mammals». Science. 351 (6271): 391–396. Bibcode:2016Sci…351..391S. doi:10.1126/science.aad6780. PMC 4888079. PMID 26721685.
- ^ Casas E, Cai G, Neill JD (2015). «Characterization of circulating transfer RNA-derived RNA fragments in cattle». Frontiers in Genetics. 6: 271. doi:10.3389/fgene.2015.00271. PMC 4547532. PMID 26379699.
- ^ Hirose Y, Ikeda KT, Noro E, Hiraoka K, Tomita M, Kanai A (July 2015). «Precise mapping and dynamics of tRNA-derived fragments (tRFs) in the development of Triops cancriformis (tadpole shrimp)». BMC Genetics. 16: 83. doi:10.1186/s12863-015-0245-5. PMC 4501094. PMID 26168920.
- ^ Karaiskos S, Naqvi AS, Swanson KE, Grigoriev A (September 2015). «Age-driven modulation of tRNA-derived fragments in Drosophila and their potential targets». Biology Direct. 10: 51. doi:10.1186/s13062-015-0081-6. PMC 4572633. PMID 26374501.
- ^ Pliatsika V, Loher P, Telonis AG, Rigoutsos I (August 2016). «MINTbase: a framework for the interactive exploration of mitochondrial and nuclear tRNA fragments». Bioinformatics. 32 (16): 2481–2489. doi:10.1093/bioinformatics/btw194. PMC 4978933. PMID 27153631.
- ^ Pliatsika V, Loher P, Magee R, Telonis AG, Londin E, Shigematsu M, Kirino Y, Rigoutsos I (January 2018). «MINTbase v2.0: a comprehensive database for tRNA-derived fragments that includes nuclear and mitochondrial fragments from all The Cancer Genome Atlas projects». Nucleic Acids Research. 46(D1) (D1): D152–D159. doi:10.1093/nar/gkx1075. PMC 5753276. PMID 29186503.
- ^ Kumar P, Mudunuri SB, Anaya J, Dutta A (January 2015). «tRFdb: a database for transfer RNA fragments». Nucleic Acids Research. 43 (Database issue): D141-5. doi:10.1093/nar/gku1138. PMC 4383946. PMID 25392422.
- ^ a b Vincent RM, Wright BW, Jaschke PR (April 2019). «Measuring Amber Initiator tRNA Orthogonality in a Genomically Recoded Organism». ACS Synthetic Biology. 8 (4): 675–685. doi:10.1021/acssynbio.9b00021. PMID 30856316. S2CID 75136654.
- ^ Govindan A, Miryala S, Mondal S, Varshney U (November 2018). «Development of Assay Systems for Amber Codon Decoding at the Steps of Initiation and Elongation in Mycobacteria». Journal of Bacteriology. 200 (22). doi:10.1128/jb.00372-18. PMC 6199473. PMID 30181124.
- ^ White RJ (March 1997). «Regulation of RNA polymerases I and III by the retinoblastoma protein: a mechanism for growth control?». Trends in Biochemical Sciences. 22 (3): 77–80. doi:10.1016/S0968-0004(96)10067-0. PMID 9066256.
- ^ Sharp S, Dingermann T, Söll D (September 1982). «The minimum intragenic sequences required for promotion of eukaryotic tRNA gene transcription». Nucleic Acids Research. 10 (18): 5393–5406. doi:10.1093/nar/10.18.5393. PMC 320884. PMID 6924209.
- ^ a b Dieci G, Fiorino G, Castelnuovo M, Teichmann M, Pagano A (December 2007). «The expanding RNA polymerase III transcriptome». Trends in Genetics. 23 (12): 614–622. doi:10.1016/j.tig.2007.09.001. hdl:11381/1706964. PMID 17977614.
- ^ Tocchini-Valentini GD, Fruscoloni P, Tocchini-Valentini GP (December 2009). «Processing of multiple-intron-containing pretRNA». Proceedings of the National Academy of Sciences of the United States of America. 106 (48): 20246–20251. Bibcode:2009PNAS..10620246T. doi:10.1073/pnas.0911658106. PMC 2787110. PMID 19910528.
- ^ Abelson J, Trotta CR, Li H (May 1998). «tRNA splicing». The Journal of Biological Chemistry. 273 (21): 12685–12688. doi:10.1074/jbc.273.21.12685. PMID 9582290.
- ^ a b Soma A (2014). «Circularly permuted tRNA genes: their expression and implications for their physiological relevance and development». Frontiers in Genetics. 5: 63. doi:10.3389/fgene.2014.00063. PMC 3978253. PMID 24744771.
- ^ Frank DN, Pace NR (1998). «Ribonuclease P: unity and diversity in a tRNA processing ribozyme». Annual Review of Biochemistry. 67 (1): 153–180. doi:10.1146/annurev.biochem.67.1.153. PMID 9759486.
- ^ Ceballos M, Vioque A (2007). «tRNase Z». Protein and Peptide Letters. 14 (2): 137–145. doi:10.2174/092986607779816050. PMID 17305600.
- ^ Randau L, Schröder I, Söll D (May 2008). «Life without RNase P». Nature. 453 (7191): 120–123. Bibcode:2008Natur.453..120R. doi:10.1038/nature06833. PMID 18451863. S2CID 3103527.
- ^ Weiner AM (October 2004). «tRNA maturation: RNA polymerization without a nucleic acid template». Current Biology. 14 (20): R883-5. doi:10.1016/j.cub.2004.09.069. PMID 15498478.
- ^ Kutay U, Lipowsky G, Izaurralde E, Bischoff FR, Schwarzmaier P, Hartmann E, Görlich D (February 1998). «Identification of a tRNA-specific nuclear export receptor». Molecular Cell. 1 (3): 359–369. doi:10.1016/S1097-2765(00)80036-2. PMID 9660920.
- ^ Arts GJ, Fornerod M, Mattaj IW (March 1998). «Identification of a nuclear export receptor for tRNA». Current Biology. 8 (6): 305–314. doi:10.1016/S0960-9822(98)70130-7. PMID 9512417. S2CID 17803674.
- ^ Arts GJ, Kuersten S, Romby P, Ehresmann B, Mattaj IW (December 1998). «The role of exportin-t in selective nuclear export of mature tRNAs». The EMBO Journal. 17 (24): 7430–7441. doi:10.1093/emboj/17.24.7430. PMC 1171087. PMID 9857198.
- ^ Yoshihisa T, Yunoki-Esaki K, Ohshima C, Tanaka N, Endo T (August 2003). «Possibility of cytoplasmic pre-tRNA splicing: the yeast tRNA splicing endonuclease mainly localizes on the mitochondria». Molecular Biology of the Cell. 14 (8): 3266–3279. doi:10.1091/mbc.E02-11-0757. PMC 181566. PMID 12925762.
- ^ Kühnlein, Alexandra; Lanzmich, Simon A.; Brun, Dieter (2 March 2021). «tRNA sequences can assemble into a replicator». eLife. 10. doi:10.7554/eLife.63431. PMC 7924937. PMID 33648631.
- ^ Maximilian, Ludwig (3 April 2021). «Solving the Chicken-and-the-Egg Problem – «A Step Closer to the Reconstruction of the Origin of Life»«. SciTechDaily. Retrieved 3 April 2021.
- ^ Kresge, Nicole; Simoni, Robert D.; Hill, Robert L. (October 7, 2005). «The Discovery of tRNA by Paul C. Zamecnik». Journal of Biological Chemistry. 280 (40): e37–e39. doi:10.1016/S0021-9258(20)79029-0 – via www.jbc.org.
- ^ Clark BF (October 2006). «The crystal structure of tRNA» (PDF). Journal of Biosciences. 31 (4): 453–457. doi:10.1007/BF02705184. PMID 17206065. S2CID 19558731.
- ^ Holley RW, Apgar J, Everett GA, Madison JT, Marquisee M, Merrill SH, Penswick JR, Zamir A (March 1965). «Structure of a Ribonucleic Acid». Science. 147 (3664): 1462–1465. Bibcode:1965Sci…147.1462H. doi:10.1126/science.147.3664.1462. PMID 14263761. S2CID 40989800.
- ^ «Obituary». The New York Times. July 4, 1991.
- ^ «The Nobel Prize in Physiology or Medicine 1968: Robert W. Holley – Facts». Nobel Prize Outreach AB. 2022. Retrieved 18 March 2022.
- ^ Ladner JE, Jack A, Robertus JD, Brown RS, Rhodes D, Clark BF, Klug A (November 1975). «Structure of yeast phenylalanine transfer RNA at 2.5 A resolution». Proceedings of the National Academy of Sciences of the United States of America. 72 (11): 4414–4418. Bibcode:1975PNAS…72.4414L. doi:10.1073/pnas.72.11.4414. PMC 388732. PMID 1105583.
- ^ Kim SH, Quigley GJ, Suddath FL, McPherson A, Sneden D, Kim JJ, Weinzierl J, Rich A (January 1973). «Three-dimensional structure of yeast phenylalanine transfer RNA: folding of the polynucleotide chain». Science. 179 (4070): 285–288. Bibcode:1973Sci…179..285K. doi:10.1126/science.179.4070.285. PMID 4566654. S2CID 28916938.
External links[edit]
Wikimedia Commons has media related to TRNA.
- tRNAdb (updated and completely restructured version of Spritzls tRNA compilation)
- tRNA link to heart disease and stroke
- GtRNAdb: Collection of tRNAs identified from complete genomes
- HGNC: Gene nomenclature of human tRNAs
- Molecule of the Month © RCSB Protein Data Bank:
- Transfer RNA
- Aminoacyl-tRNA Synthetases
- Elongation Factors
- Rfam entry for tRNA
From Wikipedia, the free encyclopedia
The interaction of tRNA and mRNA in protein synthesis.
| tRNA | |
|---|---|
| Identifiers | |
| Symbol | t |
| Rfam | RF00005 |
| Other data | |
| RNA type | gene, tRNA |
| PDB structures | PDBe 3icq, 1asy, 1asz, 1il2, 2tra, 3tra, 486d, 1fir, 1yfg, 3eph, 3epj, 3epk, 3epl, 1efw, 1c0a, 2ake, 2azx, 2dr2, 1f7u, 1f7v, 3foz, 2hgp, 2j00, 2j02, 2ow8, 2v46, 2v48, 2wdg, 2wdh, 2wdk, 2wdm, 2wh1 |
Transfer RNA (abbreviated tRNA and formerly referred to as sRNA, for soluble RNA[1]) is an adaptor molecule composed of RNA, typically 76 to 90 nucleotides in length (in eukaryotes),[2] that serves as the physical link between the mRNA and the amino acid sequence of proteins. tRNAs genes from Bacteria are typically shorter (mean = 77.6 bp) than tRNAs from Archaea (mean = 83.1 bp) and eukaryotes (mean = 84.7 bp).[3] The mature tRNA follows an opposite pattern with tRNAs from Bacteria being usually longer (median = 77.6 nt) than tRNAs from Archaea (median = 76.8 nt), with eukaryotes exhibiting the shortest mature tRNAs (median = 74.5 nt).[3] Transfer RNA (tRNA) does this by carrying an amino acid to the protein synthesizing machinery of a cell called the ribosome. Complementation of a 3-nucleotide codon in a messenger RNA (mRNA) by a 3-nucleotide anticodon of the tRNA results in protein synthesis based on the mRNA code. As such, tRNAs are a necessary component of translation, the biological synthesis of new proteins in accordance with the genetic code.
Overview[edit]
While the specific nucleotide sequence of an mRNA specifies which amino acids are incorporated into the protein product of the gene from which the mRNA is transcribed, the role of tRNA is to specify which sequence from the genetic code corresponds to which amino acid.[4] The mRNA encodes a protein as a series of contiguous codons, each of which is recognized by a particular tRNA. One end of the tRNA matches the genetic code in a three-nucleotide sequence called the anticodon. The anticodon forms three complementary base pairs with a codon in mRNA during protein biosynthesis.
On the other end of the tRNA is a covalent attachment to the amino acid that corresponds to the anticodon sequence. Each type of tRNA molecule can be attached to only one type of amino acid, so each organism has many types of tRNA. Because the genetic code contains multiple codons that specify the same amino acid, there are several tRNA molecules bearing different anticodons which carry the same amino acid.
The covalent attachment to the tRNA 3’ end is catalysed by enzymes called aminoacyl tRNA synthetases. During protein synthesis, tRNAs with attached amino acids are delivered to the ribosome by proteins called elongation factors, which aid in association of the tRNA with the ribosome, synthesis of the new polypeptide, and translocation (movement) of the ribosome along the mRNA. If the tRNA’s anticodon matches the mRNA, another tRNA already bound to the ribosome transfers the growing polypeptide chain from its 3’ end to the amino acid attached to the 3’ end of the newly delivered tRNA, a reaction catalysed by the ribosome. A large number of the individual nucleotides in a tRNA molecule may be chemically modified, often by methylation or deamidation. These unusual bases sometimes affect the tRNA’s interaction with ribosomes and sometimes occur in the anticodon to alter base-pairing properties.[5]
Structure[edit]
Secondary cloverleaf structure of tRNA
Tertiary structure of tRNA. CCA tail in yellow, Acceptor stem in purple, Variable loop in orange, D arm in red, Anticodon arm in blue with Anticodon in black, T arm in green.
3D animated GIF showing the structure of phenylalanine-tRNA from yeast (PDB ID 1ehz). White lines indicate base pairing by hydrogen bonds. In the orientation shown, the acceptor stem is on top and the anticodon on the bottom[6]
The structure of tRNA can be decomposed into its primary structure, its secondary structure (usually visualized as the cloverleaf structure), and its tertiary structure[7] (all tRNAs have a similar L-shaped 3D structure that allows them to fit into the P and A sites of the ribosome). The cloverleaf structure becomes the 3D L-shaped structure through coaxial stacking of the helices, which is a common RNA tertiary structure motif. The lengths of each arm, as well as the loop ‘diameter’, in a tRNA molecule vary from species to species.[7][8]
The tRNA structure consists of the following:
- The acceptor stem is a 7- to 9-base pair (bp) stem made by the base pairing of the 5′-terminal nucleotide with the 3′-terminal nucleotide (which contains the CCA 3′-terminal group used to attach the amino acid). In general, such 3′-terminal tRNA-like structures are referred to as ‘genomic tags’. The acceptor stem may contain non-Watson-Crick base pairs.[7][9]
- The CCA tail is a cytosine-cytosine-adenine sequence at the 3′ end of the tRNA molecule. The amino acid loaded onto the tRNA by aminoacyl tRNA synthetases, to form aminoacyl-tRNA, is covalently bonded to the 3′-hydroxyl group on the CCA tail.[10] This sequence is important for the recognition of tRNA by enzymes and critical in translation.[11][12] In prokaryotes, the CCA sequence is transcribed in some tRNA sequences. In most prokaryotic tRNAs and eukaryotic tRNAs, the CCA sequence is added during processing and therefore does not appear in the tRNA gene.[13]
- The D loop is a 4- to 6-bp stem ending in a loop that often contains dihydrouridine.[7]
- The anticodon loop is a 5-bp stem whose loop contains the anticodon.[7] The tRNA 5′-to-3′ primary structure contains the anticodon but in reverse order, since 3′-to-5′ directionality is required to read the mRNA from 5′-to-3′.
- The ΨU loop is named so because of the characteristic presence of the unusual base ΨU in the loop, where Ψ is pseudo uridine, a modified uridine. The modified base is often found within the sequence 5′ -TΨUCG-3′.[7]
- The variable loop sits between the anticodon loop and the ΨU loop and, as its name implies, varies in size from 3 to 21 bases.[14]
Anticodon[edit]
An anticodon[15] is a unit of three nucleotides corresponding to the three bases of an mRNA codon. Each tRNA has a distinct anticodon triplet sequence that can form 3 complementary base pairs to one or more codons for an amino acid. Some anticodons pair with more than one codon due to wobble base pairing. Frequently, the first nucleotide of the anticodon is one not found on mRNA: inosine, which can hydrogen bond to more than one base in the corresponding codon position.[5]: 29.3.9 In genetic code, it is common for a single amino acid to be specified by all four third-position possibilities, or at least by both pyrimidines and purines; for example, the amino acid glycine is coded for by the codon sequences GGU, GGC, GGA, and GGG. Other modified nucleotides may also appear at the first anticodon position—sometimes known as the «wobble position»—resulting in subtle changes to the genetic code, as for example in mitochondria.[16]
Per cell, 61 tRNA types are required to provide one-to-one correspondence between tRNA molecules and codons that specify amino acids, as there are 61 sense codons of the standard genetic code. However, many cells have under 61 types of tRNAs because the wobble base is capable of binding to several, though not necessarily all, of the codons that specify a particular amino acid. At least 31 tRNAs are required to translate, unambiguously, all 61 sense codons.[4][17]
Aminoacylation[edit]
Aminoacylation is the process of adding an aminoacyl group to a compound. It covalently links an amino acid to the CCA 3′ end of a tRNA molecule.
Each tRNA is aminoacylated (or charged) with a specific amino acid by an aminoacyl tRNA synthetase. There is normally a single aminoacyl tRNA synthetase for each amino acid, despite the fact that there can be more than one tRNA, and more than one anticodon for an amino acid. Recognition of the appropriate tRNA by the synthetases is not mediated solely by the anticodon, and the acceptor stem often plays a prominent role.[18]
Reaction:
- amino acid + ATP → aminoacyl-AMP + PPi
- aminoacyl-AMP + tRNA → aminoacyl-tRNA + AMP
Certain organisms can have one or more aminophosphate-tRNA synthetases missing. This leads to charging of the tRNA by a chemically related amino acid, and by use of an enzyme or enzymes, the tRNA is modified to be correctly charged. For example, Helicobacter pylori has glutaminyl tRNA synthetase missing. Thus, glutamate tRNA synthetase charges tRNA-glutamine(tRNA-Gln) with glutamate. An amidotransferase then converts the acid side chain of the glutamate to the amide, forming the correctly charged gln-tRNA-Gln.
Interference with aminoacylation may be useful as an approach to treating some diseases: cancerous cells may be relatively vulnerable to disturbed aminoacylation compared to healthy cells. The protein synthesis associated with cancer and viral biology is often very dependent on specific tRNA molecules. For instance, for liver cancer charging tRNA-Lys-CUU with lysine sustains liver cancer cell growth and metastasis, whereas healthy cells have a much lower dependence on this tRNA to support cellular physiology.[19] Similarly, hepatitis E virus requires a tRNA landscape that substantially differs from that associated with uninfected cells.[20] Hence, inhibition of aminoacylation of specific tRNA species is considered a promising novel avenue for the rational treatment of a plethora of diseases.
Binding to ribosome[edit]
The range of conformations adopted by tRNA as it transits the A/T through P/E sites on the ribosome. The Protein Data Bank (PDB) codes for the structural models used as end points of the animation are given. Both tRNAs are modeled as phenylalanine-specific tRNA from Escherichia coli, with the A/T tRNA as a homology model of the deposited coordinates. Color coding as shown for tRNA tertiary structure. Adapted from.[21]
The ribosome has three binding sites for tRNA molecules that span the space between the two ribosomal subunits: the A (aminoacyl),[22] P (peptidyl), and E (exit) sites. In addition, the ribosome has two other sites for tRNA binding that are used during mRNA decoding or during the initiation of protein synthesis. These are the T site (named elongation factor Tu) and I site (initiation).[23][24] By convention, the tRNA binding sites are denoted with the site on the small ribosomal subunit listed first and the site on the large ribosomal subunit listed second. For example, the A site is often written A/A, the P site, P/P, and the E site, E/E.[23] The binding proteins like L27, L2, L14, L15, L16 at the A- and P- sites have been determined by affinity labeling by A. P. Czernilofsky et al. (Proc. Natl. Acad. Sci, USA, pp. 230–234, 1974).
Once translation initiation is complete, the first aminoacyl tRNA is located in the P/P site, ready for the elongation cycle described below. During translation elongation, tRNA first binds to the ribosome as part of a complex with elongation factor Tu (EF-Tu) or its eukaryotic (eEF-1) or archaeal counterpart. This initial tRNA binding site is called the A/T site. In the A/T site, the A-site half resides in the small ribosomal subunit where the mRNA decoding site is located. The mRNA decoding site is where the mRNA codon is read out during translation. The T-site half resides mainly on the large ribosomal subunit where EF-Tu or eEF-1 interacts with the ribosome. Once mRNA decoding is complete, the aminoacyl-tRNA is bound in the A/A site and is ready for the next peptide bond[25] to be formed to its attached amino acid. The peptidyl-tRNA, which transfers the growing polypeptide to the aminoacyl-tRNA bound in the A/A site, is bound in the P/P site. Once the peptide bond is formed, the tRNA in the P/P site is acylated, or has a free 3’ end, and the tRNA in the A/A site dissociates the growing polypeptide chain. To allow for the next elongation cycle, the tRNAs then move through hybrid A/P and P/E binding sites, before completing the cycle and residing in the P/P and E/E sites. Once the A/A and P/P tRNAs have moved to the P/P and E/E sites, the mRNA has also moved over by one codon and the A/T site is vacant, ready for the next round of mRNA decoding. The tRNA bound in the E/E site then leaves the ribosome.
The P/I site is actually the first to bind to aminoacyl tRNA, which is delivered by an initiation factor called IF2 in bacteria.[24] However, the existence of the P/I site in eukaryotic or archaeal ribosomes has not yet been confirmed. The P-site protein L27 has been determined by affinity labeling by E. Collatz and A. P. Czernilofsky (FEBS Lett., Vol. 63, pp. 283–286, 1976).
tRNA genes[edit]
Organisms vary in the number of tRNA genes in their genome. For example, the nematode worm C. elegans, a commonly used model organism in genetics studies, has 29,647 [26] genes in its nuclear genome, of which 620 code for tRNA.[27][28] The budding yeast Saccharomyces cerevisiae has 275 tRNA genes in its genome. The number of tRNA genes per genome can vary widely, with bacterial species from groups such as Fusobacteria and Tenericutes having around 30 genes per genome while complex eukaryotic genomes such as the zebrafish (Danio rerio) can bear more than 10 thousand tRNA genes.[3]
In the human genome, which, according to January 2013 estimates, has about 20,848 protein coding genes [29] in total, there are 497 nuclear genes encoding cytoplasmic tRNA molecules, and 324 tRNA-derived pseudogenes—tRNA genes thought to be no longer functional[30] (although pseudo tRNAs have been shown to be involved in antibiotic resistance in bacteria).[31] As with all eukaryotes, there are 22 mitochondrial tRNA genes[32][full citation needed] in humans. Mutations in some of these genes have been associated with severe diseases like the MELAS syndrome. Regions in nuclear chromosomes, very similar in sequence to mitochondrial tRNA genes, have also been identified (tRNA-lookalikes).[33] These tRNA-lookalikes are also considered part of the nuclear mitochondrial DNA (genes transferred from the mitochondria to the nucleus).[33][34] The phenomenon of multiple nuclear copies of mitochondrial tRNA (tRNA-lookalikes) has been observed in many higher organisms from human to the opossum[35] suggesting the possibility that the lookalikes are functional.
Cytoplasmic tRNA genes can be grouped into 49 families according to their anticodon features. These genes are found on all chromosomes, except the 22 and Y chromosome. High clustering on 6p is observed (140 tRNA genes), as well on 1 chromosome.[30]
The HGNC, in collaboration with the Genomic tRNA Database (GtRNAdb) and experts in the field, has approved unique names for human genes that encode tRNAs.
Evolution[edit]
The top half of tRNA (consisting of the T arm and the acceptor stem with 5′-terminal phosphate group and 3′-terminal CCA group) and the bottom half (consisting of the D arm and the anticodon arm) are independent units in structure as well as in function. The top half may have evolved first including the 3′-terminal genomic tag which originally may have marked tRNA-like molecules for replication in early RNA world. The bottom half may have evolved later as an expansion, e.g. as protein synthesis started in RNA world and turned it into a ribonucleoprotein world (RNP world). This proposed scenario is called genomic tag hypothesis. In fact, tRNA and tRNA-like aggregates have an important catalytic influence (i.e., as ribozymes) on replication still today. These roles may be regarded as ‘molecular (or chemical) fossils’ of RNA world.[36]
Genomic tRNA content is a differentiating feature of genomes among biological domains of life: Archaea present the simplest situation in terms of genomic tRNA content with a uniform number of gene copies, Bacteria have an intermediate situation and Eukarya present the most complex situation.[37] Eukarya present not only more tRNA gene content than the other two kingdoms but also a high variation in gene copy number among different isoacceptors, and this complexity seem to be due to duplications of tRNA genes and changes in anticodon specificity[citation needed].
Evolution of the tRNA gene copy number across different species has been linked to the appearance of specific tRNA modification enzymes (uridine methyltransferases in Bacteria, and adenosine deaminases in Eukarya), which increase the decoding capacity of a given tRNA.[37] As an example, tRNAAla encodes four different tRNA isoacceptors (AGC, UGC, GGC and CGC). In Eukarya, AGC isoacceptors are extremely enriched in gene copy number in comparison to the rest of isoacceptors, and this has been correlated with its A-to-I modification of its wobble base. This same trend has been shown for most amino acids of eukaryal species. Indeed, the effect of these two tRNA modifications is also seen in codon usage bias. Highly expressed genes seem to be enriched in codons that are exclusively using codons that will be decoded by these modified tRNAs, which suggests a possible role of these codons—and consequently of these tRNA modifications—in translation efficiency.[37]
It is important to note that many species have lost specific tRNAs during evolution. For instance, both mammals and birds lack the same 14 out of the possible 64 tRNA genes, but other life forms contain these tRNAs.[38] For translating codons for which an exactly pairing tRNA is missing, organisms resort to a strategy called wobbling, in which imperfectly matched tRNA/mRNA pairs still give rise to translation, although this strategy also increases to propensity for translation errors.[39] The reasons why tRNA genes have been lost during evolution remains under debate but may relate improving resistance to viral infection.[40] Because nucleotide triplets can present more combinations than there are amino acids and associated tRNAs, there is redundancy in the genetic code, and several different 3-nucleotide codons can express the same amino acid. This codon bias is what necessitates codon optimization.
tRNA-derived fragments[edit]
tRNA-derived fragments (or tRFs) are short molecules that emerge after cleavage of the mature tRNAs or the precursor transcript.[41][42][43][44] Both cytoplasmic and mitochondrial tRNAs can produce fragments.[45] There are at least four structural types of tRFs believed to originate from mature tRNAs, including the relatively long tRNA halves and short 5’-tRFs, 3’-tRFs and i-tRFs.[41][45][46] The precursor tRNA can be cleaved to produce molecules from the 5’ leader or 3’ trail sequences. Cleavage enzymes include Angiogenin, Dicer, RNase Z and RNase P.[41][42] Especially in the case of Angiogenin, the tRFs have a characteristically unusual cyclic phosphate at their 3’ end and a hydroxyl group at the 5’ end.[47] tRFs appear to play a role in RNA interference, specifically in the suppression of retroviruses and retrotransposons that use tRNA as a primer for replication. Half-tRNAs cleaved by angiogenin are also known as tiRNAs. The biogenesis of smaller fragments, including those that function as piRNAs, are less understood.[48]
tRFs have multiple dependencies and roles; such as exhibiting significant changes between sexes, among races and disease status.[45][49][50] Functionally, they can be loaded on Ago and act through RNAi pathways,[43][46][51] participate in the formation of stress granules,[52] displace mRNAs from RNA-binding proteins[53] or inhibit translation.[54] At the system or the organismal level, the four types of tRFs have a diverse spectrum of activities. Functionally, tRFs are associated with viral infection,[55] cancer,[46] cell proliferation [47] and also with epigenetic transgenerational regulation of metabolism.[56]
tRFs are not restricted to humans and have been shown to exist in multiple organisms.[46][57][58][59]
Two online tools are available for those wishing to learn more about tRFs: the framework for the interactive exploration of mitochondrial and nuclear tRNA fragments (MINTbase)[60][61] and the relational database of Transfer RNA related Fragments (tRFdb).[62] MINTbase also provides a naming scheme for the naming of tRFs called tRF-license plates (or MINTcodes) that is genome independent; the scheme compresses an RNA sequence into a shorter string.
Engineered tRNAs[edit]
Artificial suppressor elongator tRNAs are used to incorporate unnatural amino acids at nonsense codons placed in the coding sequence of a gene. Engineered initiator tRNAs (tRNAfMet2 with CUA anticodon encoded by metY gene) have been used to initiate translation at the amber stop codon UAG. This type of engineered tRNA is called a nonsense suppressor tRNA because it suppresses the translation stop signal that normally occurs at UAG codons. The amber initiator tRNA inserts methionine[63] and glutamine[64] at UAG codons preceded by a strong Shine-Dalgarno sequence. An investigation of the amber initiator tRNA showed that it was orthogonal to the regular AUG start codon showing no detectable off-target translation initiation events in a genomically recoded E. coli strain.[63]
tRNA biogenesis[edit]
In eukaryotic cells, tRNAs are transcribed by RNA polymerase III as pre-tRNAs in the nucleus.[65]
RNA polymerase III recognizes two highly conserved downstream promoter sequences: the 5′ intragenic control region (5′-ICR, D-control region, or A box), and the 3′-ICR (T-control region or B box) inside tRNA genes.[2][66][67]
The first promoter begins at +8 of mature tRNAs and the second promoter is located 30–60 nucleotides downstream of the first promoter. The transcription terminates after a stretch of four or more thymidines.[2][67]
Bulge-helix-bulge motif of a tRNA intron
Pre-tRNAs undergo extensive modifications inside the nucleus. Some pre-tRNAs contain introns that are spliced, or cut, to form the functional tRNA molecule;[68] in bacteria these self-splice, whereas in eukaryotes and archaea they are removed by tRNA-splicing endonucleases.[69] Eukaryotic pre-tRNA contains bulge-helix-bulge (BHB) structure motif that is important for recognition and precise splicing of tRNA intron by endonucleases.[70] This motif position and structure are evolutionarily conserved. However, some organisms, such as unicellular algae have a non-canonical position of BHB-motif as well as 5′- and 3′-ends of the spliced intron sequence.[70]
The 5′ sequence is removed by RNase P,[71] whereas the 3′ end is removed by the tRNase Z enzyme.[72]
A notable exception is in the archaeon Nanoarchaeum equitans, which does not possess an RNase P enzyme and has a promoter placed such that transcription starts at the 5′ end of the mature tRNA.[73]
The non-templated 3′ CCA tail is added by a nucleotidyl transferase.[74]
Before tRNAs are exported into the cytoplasm by Los1/Xpo-t,[75][76] tRNAs are aminoacylated.[77]
The order of the processing events is not conserved.
For example, in yeast, the splicing is not carried out in the nucleus but at the cytoplasmic side of mitochondrial membranes.[78]
Nonetheless, In March 2021, researchers reported evidence suggesting that a preliminary form of transfer RNA could have been a replicator molecule in the very early development of life, or abiogenesis.[79][80]
History[edit]
The existence of tRNA was first hypothesized by Francis Crick as the «adaptor hypothesis» based on the assumption that there must exist an adapter molecule capable of mediating the translation of the RNA alphabet into the protein alphabet. Paul C Zamecnik and Mahlon Hoagland discovered tRNA [81] Significant research on structure was conducted in the early 1960s by Alex Rich and Donald Caspar, two researchers in Boston, the Jacques Fresco group in Princeton University and a United Kingdom group at King’s College London.[82] In 1965, Robert W. Holley of Cornell University reported the primary structure and suggested three secondary structures.[83] tRNA was first crystallized in Madison, Wisconsin, by Robert M. Bock.[84] The cloverleaf structure was ascertained by several other studies in the following years[85] and was finally confirmed using X-ray crystallography studies in 1974. Two independent groups, Kim Sung-Hou working under Alexander Rich and a British group headed by Aaron Klug, published the same crystallography findings within a year.[86][87]
See also[edit]
- Cloverleaf model of tRNA
- Kim Sung-Hou
- Kissing stem-loop
- mRNA
- non-coding RNA and introns
- Slippery sequence
- tmRNA
- Transfer RNA-like structures
- Translation
- tRNADB
- Wobble hypothesis
- Aminoacyl-tRNA
References[edit]
- ^ Plescia OJ, Palczuk NC, Cora-Figueroa E, Mukherjee A, Braun W (October 1965). «Production of antibodies to soluble RNA (sRNA)». Proceedings of the National Academy of Sciences of the United States of America. 54 (4): 1281–1285. Bibcode:1965PNAS…54.1281P. doi:10.1073/pnas.54.4.1281. PMC 219862. PMID 5219832.
- ^ a b c Sharp SJ, Schaack J, Cooley L, Burke DJ, Söll D (1985). «Structure and transcription of eukaryotic tRNA genes». CRC Critical Reviews in Biochemistry. 19 (2): 107–144. doi:10.3109/10409238509082541. PMID 3905254.
- ^ a b c Santos, Fenícia Brito; Del-Bem, Luiz-Eduardo (January 2023). «The Evolution of tRNA Copy Number and Repertoire in Cellular Life». Genes. 14 (1): 27. doi:10.3390/genes14010027. ISSN 2073-4425. PMC 9858662. PMID 36672768.
- ^ a b Crick FH (December 1968). «The origin of the genetic code». Journal of Molecular Biology. 38 (3): 367–379. doi:10.1016/0022-2836(68)90392-6. PMID 4887876. S2CID 4144681.
- ^ a b Stryer L, Berg JM, Tymoczko JL (2002). Biochemistry (5th ed.). San Francisco: W. H. Freeman. ISBN 978-0-7167-4955-4.
- ^ «Transfer RNA (tRNA)». Proteopedia.org. Retrieved 7 November 2018.
- ^ a b c d e f Itoh Y, Sekine S, Suetsugu S, Yokoyama S (July 2013). «Tertiary structure of bacterial serenocysteine tRNA». Nucleic Acids Research. 41 (13): 6729–6738. doi:10.1093/nar/gkt321. PMC 3711452. PMID 23649835.
- ^ Goodenbour JM, Pan T (29 October 2006). «Diversity of tRNA genes in eukaryotes». Nucleic Acids Research. 34 (21): 6137–6146. doi:10.1093/nar/gkl725. PMC 1693877. PMID 17088292.
- ^
- ^ Ibba M, Soll D (June 2000). «Aminoacyl-tRNA synthesis». Annual Review of Biochemistry. 69 (1): 617–650. doi:10.1146/annurev.biochem.69.1.617. PMID 10966471.
- ^ Sprinzl M, Cramer F (1979). «The -C-C-A end of tRNA and its role in protein biosynthesis». Progress in Nucleic Acid Research and Molecular Biology. 22: 1–69. doi:10.1016/s0079-6603(08)60798-9. ISBN 978-0-12-540022-0. PMID 392600.
- ^ Green R, Noller HF (1997). «Ribosomes and translation». Annual Review of Biochemistry. 66: 679–716. doi:10.1146/annurev.biochem.66.1.679. PMID 9242921.
- ^ Aebi M, Kirchner G, Chen JY, Vijayraghavan U, Jacobson A, Martin NC, Abelson J, et al. (September 1990). «Isolation of a temperature-sensitive mutant with an altered tRNA nucleotidyltransferase and cloning of the gene encoding tRNA nucleotidyltransferase in the yeast Saccharomyces cerevisiae». The Journal of Biological Chemistry. 265 (27): 16216–16220. doi:10.1016/S0021-9258(17)46210-7. PMID 2204621.
- ^ Prabhakar A, Krahn N, Zhang J, Vargas-Rodriguez O, Krupkin M, Fu Z, Acosta-Reyes FJ, Ge X, Choi J, Crnković A, Ehrenberg M, Puglisi EV, Söll D, Puglisi J (Jul 2022). «Uncovering translation roadblocks during the development of a synthetic tRNA». Nucleic Acids Res. online ahead of print (18): 10201–10211. doi:10.1093/nar/gkac576. PMC 9561287. PMID 35882385.
- ^ Felsenfeld G, Cantoni GL (May 1964). «Use of thermal denaturation studies to investigate the base sequence of yeast serine sRNA». Proceedings of the National Academy of Sciences of the United States of America. 51 (5): 818–826. Bibcode:1964PNAS…51..818F. doi:10.1073/pnas.51.5.818. PMC 300168. PMID 14172997.
- ^ Suzuki T, Suzuki T (June 2014). «A complete landscape of post-transcriptional modifications in mammalian mitochondrial tRNAs». Nucleic Acids Research. 42 (11): 7346–7357. doi:10.1093/nar/gku390. PMC 4066797. PMID 24831542.
- ^ Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scott MP, Zipursky SL, Darnell J. (2004). Molecular Cell Biology. WH Freeman: New York. 5th ed.ISBN 978-0716743668[page needed]
- ^ Schimmel P, Giegé R, Moras D, Yokoyama S (October 1993). «An operational RNA code for amino acids and possible relationship to genetic code». Proceedings of the National Academy of Sciences of the United States of America. 90 (19): 8763–8768. Bibcode:1993PNAS…90.8763S. doi:10.1073/pnas.90.19.8763. PMC 47440. PMID 7692438.
- ^ Zhang R, Noordam L, Ou X, Ma B, Li Y, Das P, Shi S, Liu J, Wang L, Li P, Verstegen MM, Reddy DS, van der Laan LJ, Peppelenbosch MP, Kwekkeboom J, Smits R, Pan Q (January 2021). «The biological process of lysine-tRNA charging is therapeutically targetable in liver cancer». Liver Int. 41 (1): 206–219. doi:10.1111/liv.14692. PMC 7820958. PMID 33084231.
- ^ Ou X, Ma B, Zhang R, Miao Z, Cheng A, Peppelenbosch MP, Pan Q (June 2020). «A simplified qPCR method revealing tRNAome remodeling upon infection by genotype 3 hepatitis E virus». FEBS Letters. 594 (12): 2005–2015. doi:10.1002/1873-3468.13764. PMID 32133647.
- ^ Dunkle JA, Wang L, Feldman MB, Pulk A, Chen VB, Kapral GJ, Noeske J, Richardson JS, Blanchard SC, Cate JH (May 2011). «Structures of the bacterial ribosome in classical and hybrid states of tRNA binding». Science. 332 (6032): 981–984. Bibcode:2011Sci…332..981D. doi:10.1126/science.1202692. PMC 3176341. PMID 21596992.
- ^ Konevega AL, Soboleva NG, Makhno VI, Semenkov YP, Wintermeyer W, Rodnina MV, Katunin VI (January 2004). «Purine bases at position 37 of tRNA stabilize codon-anticodon interaction in the ribosomal A site by stacking and Mg2+-dependent interactions». RNA. 10 (1): 90–101. doi:10.1261/rna.5142404. PMC 1370521. PMID 14681588.
- ^ a b Agirrezabala X, Frank J (August 2009). «Elongation in translation as a dynamic interaction among the ribosome, tRNA, and elongation factors EF-G and EF-Tu». Quarterly Reviews of Biophysics. 42 (3): 159–200. doi:10.1017/S0033583509990060. PMC 2832932. PMID 20025795.
- ^ a b Allen GS, Zavialov A, Gursky R, Ehrenberg M, Frank J (June 2005). «The cryo-EM structure of a translation initiation complex from Escherichia coli». Cell. 121 (5): 703–712. doi:10.1016/j.cell.2005.03.023. PMID 15935757. S2CID 16146867.
- ^ Tirumalai MR, Rivas M, Tran Q, Fox GE (November 2021). «The Peptidyl Transferase Center: a Window to the Past». Microbiol Mol Biol Rev. 85 (4): e0010421. doi:10.1128/MMBR.00104-21. PMC 8579967. PMID 34756086.
- ^ WormBase web site, http://www.wormbase.org Archived 2017-04-20 at the Wayback Machine, release WS187, date 25-Jan-2008.
- ^ Spieth J, Lawson D (January 2006). «Overview of gene structure». WormBook: 1–10. doi:10.1895/wormbook.1.65.1. PMC 4781370. PMID 18023127.
- ^ Hartwell LH, Hood L, Goldberg ML, Reynolds AE, Silver LM, Veres RC. (2004). Genetics: From Genes to Genomes 2nd ed. McGraw-Hill: New York. p. 264.
- ^ Ensembl release 70 — Jan 2013 http://www.ensembl.org/Homo_sapiens/Info/StatsTable?db=core Archived 2013-12-15 at the Wayback Machine
- ^ a b Lander ES, Linton LM, Birren B, Nusbaum C, Zody MC, Baldwin J, et al. (International Human Genome Sequencing Consortium) (February 2001). «Initial sequencing and analysis of the human genome» (PDF). Nature. 409 (6822): 860–921. Bibcode:2001Natur.409..860L. doi:10.1038/35057062. PMID 11237011.
- ^ Rogers TE, Ataide SF, Dare K, Katz A, Seveau S, Roy H, Ibba M (2012). «A pseudo-tRNA modulates antibiotic resistance in Bacillus cereus». PLOS ONE. 7 (7): e41248. Bibcode:2012PLoSO…741248R. doi:10.1371/journal.pone.0041248. PMC 3399842. PMID 22815980.
- ^ Ibid. p. 529.
- ^ a b Telonis AG, Loher P, Kirino Y, Rigoutsos I (2014). «Nuclear and mitochondrial tRNA-lookalikes in the human genome». Frontiers in Genetics. 5: 344. doi:10.3389/fgene.2014.00344. PMC 4189335. PMID 25339973.
- ^ Ramos A, Barbena E, Mateiu L, del Mar González M, Mairal Q, Lima M, Montiel R, Aluja MP, Santos C, et al. (November 2011). «Nuclear insertions of mitochondrial origin: Database updating and usefulness in cancer studies». Mitochondrion. 11 (6): 946–953. doi:10.1016/j.mito.2011.08.009. PMID 21907832.
- ^ Telonis AG, Kirino Y, Rigoutsos I (2015). «Mitochondrial tRNA-lookalikes in nuclear chromosomes: Could they be functional?». RNA Biol. 12 (4): 375–380. doi:10.1080/15476286.2015.1017239. PMC 4615777. PMID 25849196.
- ^ Nancy Maizels and Alan M. Weiner: The Genomic Tag Hypothesis – What Molecular Fossils Tell Us about the Evolution of tRNA, in: The RNA World, Second Edition. 1999 Cold Spring Harbor Laboratory Press ISBN 978-0-87969-561-3/99, PDF
- ^ a b c Novoa EM, Pavon-Eternod M, Pan T, Ribas de Pouplana L (March 2012). «A role for tRNA modifications in genome structure and codon usage». Cell. 149 (1): 202–213. doi:10.1016/j.cell.2012.01.050. PMID 22464330. S2CID 16487609.
- ^ Ou X, Peng W, Yang Z, Cao J, Wang M, Peppelenbosch MP, Pan Q, Cheng A (November 2020). «Evolutionarily missing and conserved tRNA genes in human and avian». Infect. Genet. Evol. 85: 104460. doi:10.1016/j.meegid.2020.104460. PMID 32679345.
- ^ Ou X, Cao J, Cheng A, Peppelenbosch MP, Pan Q (March 2019). «Errors in translational decoding: tRNA wobbling or misincorporation?». PLOS Genetics. 15 (3): 2979–2986. doi:10.1371/journal.pgen.1008017. PMC 3158919. PMID 21930591.
- ^ Ou X, Wang M, Mao S, Cao J, Cheng A, Zhu D, Chen S, Jia R, Liu M, Yang Q, Wu Y, Zhao X, Zhang S, Liu Y, Yu Y, Zhang L, Chen X, Peppelenbosch MP, Pan Q (July 2018). «Incompatible Translation Drives a Convergent Evolution and Viral Attenuation During the Development of Live Attenuated Vaccine». Front. Cell. Infect. Microbiol. 8: 249. doi:10.3389/fcimb.2018.00249. PMC 6058041. PMID 30073153.
- ^ a b c Gebetsberger J, Polacek N (December 2013). «Slicing tRNAs to boost functional ncRNA diversity». RNA Biology. 10 (12): 1798–1806. doi:10.4161/rna.27177. PMC 3917982. PMID 24351723.
- ^ a b Shigematsu M, Honda S, Kirino Y (2014). «Transfer RNA as a source of small functional RNA». Journal of Molecular Biology and Molecular Imaging. 1 (2): 8. PMC 4572697. PMID 26389128.
- ^ a b Sobala A, Hutvagner G (2011). «Transfer RNA-derived fragments: origins, processing, and functions» (PDF). Wiley Interdisciplinary Reviews: RNA. 2 (6): 853–862. doi:10.1002/wrna.96. hdl:10453/18187. PMID 21976287. S2CID 206554146.
- ^ Keam SP, Hutvagner G (November 2015). «tRNA-Derived Fragments (tRFs): Emerging New Roles for an Ancient RNA in the Regulation of Gene Expression». Life. 5 (4): 1638–1651. doi:10.3390/life5041638. PMC 4695841. PMID 26703738.
- ^ a b c Telonis AG, Loher P, Honda S, Jing Y, Palazzo J, Kirino Y, Rigoutsos I (July 2015). «Dissecting tRNA-derived fragment complexities using personalized transcriptomes reveals novel fragment classes and unexpected dependencies». Oncotarget. 6 (28): 24797–822. doi:10.18632/oncotarget.4695. PMC 4694795. PMID 26325506.
- ^ a b c d Kumar P, Anaya J, Mudunuri SB, Dutta A (October 2014). «Meta-analysis of tRNA derived RNA fragments reveals that they are evolutionarily conserved and associate with AGO proteins to recognize specific RNA targets». BMC Biology. 12: 78. doi:10.1186/s12915-014-0078-0. PMC 4203973. PMID 25270025.
- ^ a b Honda S, Loher P, Shigematsu M, Palazzo JP, Suzuki R, Imoto I, Rigoutsos I, Kirino Y (July 2015). «Sex hormone-dependent tRNA halves enhance cell proliferation in breast and prostate cancers». Proceedings of the National Academy of Sciences of the United States of America. 112 (29): E3816–E3825. Bibcode:2015PNAS..112E3816H. doi:10.1073/pnas.1510077112. PMC 4517238. PMID 26124144.
- ^ Schorn, AJ; Martienssen, R (October 2018). «Tie-Break: Host and Retrotransposons Play tRNA». Trends in Cell Biology. 28 (10): 793–806. doi:10.1016/j.tcb.2018.05.006. PMC 6520983. PMID 29934075.
- ^ Telonis AG, Rigoutsos I (March 2018). «Race Disparities in the Contribution of miRNA Isoforms and tRNA-Derived Fragments to Triple-Negative Breast Cancer». Cancer Res. 78 (5): 1140–54. doi:10.1158/0008-5472.CAN-17-1947. PMC 5935570. PMID 29229607.
- ^ Telonis AG, Loher P, Magee R, Pliatsika V, Londin E, Kirino Y, Rigoutsos I (Jun 2019). «tRNA Fragments Show Intertwining with mRNAs of Specific Repeat Content and Have Links to Disparities». Cancer Res. 79 (12): 3034–49. doi:10.1158/0008-5472.CAN-19-0789. PMC 6571059. PMID 30996049.
- ^ Shigematsu M, Kirino Y (2015). «tRNA-Derived Short Non-coding RNA as Interacting Partners of Argonaute Proteins». Gene Regulation and Systems Biology. 9: 27–33. doi:10.4137/GRSB.S29411. PMC 4567038. PMID 26401098.
- ^ Emara MM, Ivanov P, Hickman T, Dawra N, Tisdale S, Kedersha N, Hu GF, Anderson P (April 2010). «Angiogenin-induced tRNA-derived stress-induced RNAs promote stress-induced stress granule assembly». The Journal of Biological Chemistry. 285 (14): 10959–10968. doi:10.1074/jbc.M109.077560. PMC 2856301. PMID 20129916.
- ^ Goodarzi H, Liu X, Nguyen HC, Zhang S, Fish L, Tavazoie SF (May 2015). «Endogenous tRNA-Derived Fragments Suppress Breast Cancer Progression via YBX1 Displacement». Cell. 161 (4): 790–802. doi:10.1016/j.cell.2015.02.053. PMC 4457382. PMID 25957686.
- ^ Ivanov P, Emara MM, Villen J, Gygi SP, Anderson P (August 2011). «Angiogenin-induced tRNA fragments inhibit translation initiation». Molecular Cell. 43 (4): 613–623. doi:10.1016/j.molcel.2011.06.022. PMC 3160621. PMID 21855800.
- ^ Selitsky SR, Baran-Gale J, Honda M, Yamane D, Masaki T, Fannin EE, Guerra B, Shirasaki T, Shimakami T, Kaneko S, Lanford RE, Lemon SM, Sethupathy P (January 2015). «Small tRNA-derived RNAs are increased and more abundant than microRNAs in chronic hepatitis B and C». Scientific Reports. 5: 7675. Bibcode:2015NatSR…5E7675S. doi:10.1038/srep07675. PMC 4286764. PMID 25567797.
- ^ Sharma U, Conine CC, Shea JM, Boskovic A, Derr AG, Bing XY, Belleannee C, Kucukural A, Serra RW, Sun F, Song L, Carone BR, Ricci EP, Li XZ, Fauquier L, Moore MJ, Sullivan R, Mello CC, Garber M, Rando OJ (January 2016). «Biogenesis and function of tRNA fragments during sperm maturation and fertilization in mammals». Science. 351 (6271): 391–396. Bibcode:2016Sci…351..391S. doi:10.1126/science.aad6780. PMC 4888079. PMID 26721685.
- ^ Casas E, Cai G, Neill JD (2015). «Characterization of circulating transfer RNA-derived RNA fragments in cattle». Frontiers in Genetics. 6: 271. doi:10.3389/fgene.2015.00271. PMC 4547532. PMID 26379699.
- ^ Hirose Y, Ikeda KT, Noro E, Hiraoka K, Tomita M, Kanai A (July 2015). «Precise mapping and dynamics of tRNA-derived fragments (tRFs) in the development of Triops cancriformis (tadpole shrimp)». BMC Genetics. 16: 83. doi:10.1186/s12863-015-0245-5. PMC 4501094. PMID 26168920.
- ^ Karaiskos S, Naqvi AS, Swanson KE, Grigoriev A (September 2015). «Age-driven modulation of tRNA-derived fragments in Drosophila and their potential targets». Biology Direct. 10: 51. doi:10.1186/s13062-015-0081-6. PMC 4572633. PMID 26374501.
- ^ Pliatsika V, Loher P, Telonis AG, Rigoutsos I (August 2016). «MINTbase: a framework for the interactive exploration of mitochondrial and nuclear tRNA fragments». Bioinformatics. 32 (16): 2481–2489. doi:10.1093/bioinformatics/btw194. PMC 4978933. PMID 27153631.
- ^ Pliatsika V, Loher P, Magee R, Telonis AG, Londin E, Shigematsu M, Kirino Y, Rigoutsos I (January 2018). «MINTbase v2.0: a comprehensive database for tRNA-derived fragments that includes nuclear and mitochondrial fragments from all The Cancer Genome Atlas projects». Nucleic Acids Research. 46(D1) (D1): D152–D159. doi:10.1093/nar/gkx1075. PMC 5753276. PMID 29186503.
- ^ Kumar P, Mudunuri SB, Anaya J, Dutta A (January 2015). «tRFdb: a database for transfer RNA fragments». Nucleic Acids Research. 43 (Database issue): D141-5. doi:10.1093/nar/gku1138. PMC 4383946. PMID 25392422.
- ^ a b Vincent RM, Wright BW, Jaschke PR (April 2019). «Measuring Amber Initiator tRNA Orthogonality in a Genomically Recoded Organism». ACS Synthetic Biology. 8 (4): 675–685. doi:10.1021/acssynbio.9b00021. PMID 30856316. S2CID 75136654.
- ^ Govindan A, Miryala S, Mondal S, Varshney U (November 2018). «Development of Assay Systems for Amber Codon Decoding at the Steps of Initiation and Elongation in Mycobacteria». Journal of Bacteriology. 200 (22). doi:10.1128/jb.00372-18. PMC 6199473. PMID 30181124.
- ^ White RJ (March 1997). «Regulation of RNA polymerases I and III by the retinoblastoma protein: a mechanism for growth control?». Trends in Biochemical Sciences. 22 (3): 77–80. doi:10.1016/S0968-0004(96)10067-0. PMID 9066256.
- ^ Sharp S, Dingermann T, Söll D (September 1982). «The minimum intragenic sequences required for promotion of eukaryotic tRNA gene transcription». Nucleic Acids Research. 10 (18): 5393–5406. doi:10.1093/nar/10.18.5393. PMC 320884. PMID 6924209.
- ^ a b Dieci G, Fiorino G, Castelnuovo M, Teichmann M, Pagano A (December 2007). «The expanding RNA polymerase III transcriptome». Trends in Genetics. 23 (12): 614–622. doi:10.1016/j.tig.2007.09.001. hdl:11381/1706964. PMID 17977614.
- ^ Tocchini-Valentini GD, Fruscoloni P, Tocchini-Valentini GP (December 2009). «Processing of multiple-intron-containing pretRNA». Proceedings of the National Academy of Sciences of the United States of America. 106 (48): 20246–20251. Bibcode:2009PNAS..10620246T. doi:10.1073/pnas.0911658106. PMC 2787110. PMID 19910528.
- ^ Abelson J, Trotta CR, Li H (May 1998). «tRNA splicing». The Journal of Biological Chemistry. 273 (21): 12685–12688. doi:10.1074/jbc.273.21.12685. PMID 9582290.
- ^ a b Soma A (2014). «Circularly permuted tRNA genes: their expression and implications for their physiological relevance and development». Frontiers in Genetics. 5: 63. doi:10.3389/fgene.2014.00063. PMC 3978253. PMID 24744771.
- ^ Frank DN, Pace NR (1998). «Ribonuclease P: unity and diversity in a tRNA processing ribozyme». Annual Review of Biochemistry. 67 (1): 153–180. doi:10.1146/annurev.biochem.67.1.153. PMID 9759486.
- ^ Ceballos M, Vioque A (2007). «tRNase Z». Protein and Peptide Letters. 14 (2): 137–145. doi:10.2174/092986607779816050. PMID 17305600.
- ^ Randau L, Schröder I, Söll D (May 2008). «Life without RNase P». Nature. 453 (7191): 120–123. Bibcode:2008Natur.453..120R. doi:10.1038/nature06833. PMID 18451863. S2CID 3103527.
- ^ Weiner AM (October 2004). «tRNA maturation: RNA polymerization without a nucleic acid template». Current Biology. 14 (20): R883-5. doi:10.1016/j.cub.2004.09.069. PMID 15498478.
- ^ Kutay U, Lipowsky G, Izaurralde E, Bischoff FR, Schwarzmaier P, Hartmann E, Görlich D (February 1998). «Identification of a tRNA-specific nuclear export receptor». Molecular Cell. 1 (3): 359–369. doi:10.1016/S1097-2765(00)80036-2. PMID 9660920.
- ^ Arts GJ, Fornerod M, Mattaj IW (March 1998). «Identification of a nuclear export receptor for tRNA». Current Biology. 8 (6): 305–314. doi:10.1016/S0960-9822(98)70130-7. PMID 9512417. S2CID 17803674.
- ^ Arts GJ, Kuersten S, Romby P, Ehresmann B, Mattaj IW (December 1998). «The role of exportin-t in selective nuclear export of mature tRNAs». The EMBO Journal. 17 (24): 7430–7441. doi:10.1093/emboj/17.24.7430. PMC 1171087. PMID 9857198.
- ^ Yoshihisa T, Yunoki-Esaki K, Ohshima C, Tanaka N, Endo T (August 2003). «Possibility of cytoplasmic pre-tRNA splicing: the yeast tRNA splicing endonuclease mainly localizes on the mitochondria». Molecular Biology of the Cell. 14 (8): 3266–3279. doi:10.1091/mbc.E02-11-0757. PMC 181566. PMID 12925762.
- ^ Kühnlein, Alexandra; Lanzmich, Simon A.; Brun, Dieter (2 March 2021). «tRNA sequences can assemble into a replicator». eLife. 10. doi:10.7554/eLife.63431. PMC 7924937. PMID 33648631.
- ^ Maximilian, Ludwig (3 April 2021). «Solving the Chicken-and-the-Egg Problem – «A Step Closer to the Reconstruction of the Origin of Life»«. SciTechDaily. Retrieved 3 April 2021.
- ^ Kresge, Nicole; Simoni, Robert D.; Hill, Robert L. (October 7, 2005). «The Discovery of tRNA by Paul C. Zamecnik». Journal of Biological Chemistry. 280 (40): e37–e39. doi:10.1016/S0021-9258(20)79029-0 – via www.jbc.org.
- ^ Clark BF (October 2006). «The crystal structure of tRNA» (PDF). Journal of Biosciences. 31 (4): 453–457. doi:10.1007/BF02705184. PMID 17206065. S2CID 19558731.
- ^ Holley RW, Apgar J, Everett GA, Madison JT, Marquisee M, Merrill SH, Penswick JR, Zamir A (March 1965). «Structure of a Ribonucleic Acid». Science. 147 (3664): 1462–1465. Bibcode:1965Sci…147.1462H. doi:10.1126/science.147.3664.1462. PMID 14263761. S2CID 40989800.
- ^ «Obituary». The New York Times. July 4, 1991.
- ^ «The Nobel Prize in Physiology or Medicine 1968: Robert W. Holley – Facts». Nobel Prize Outreach AB. 2022. Retrieved 18 March 2022.
- ^ Ladner JE, Jack A, Robertus JD, Brown RS, Rhodes D, Clark BF, Klug A (November 1975). «Structure of yeast phenylalanine transfer RNA at 2.5 A resolution». Proceedings of the National Academy of Sciences of the United States of America. 72 (11): 4414–4418. Bibcode:1975PNAS…72.4414L. doi:10.1073/pnas.72.11.4414. PMC 388732. PMID 1105583.
- ^ Kim SH, Quigley GJ, Suddath FL, McPherson A, Sneden D, Kim JJ, Weinzierl J, Rich A (January 1973). «Three-dimensional structure of yeast phenylalanine transfer RNA: folding of the polynucleotide chain». Science. 179 (4070): 285–288. Bibcode:1973Sci…179..285K. doi:10.1126/science.179.4070.285. PMID 4566654. S2CID 28916938.
External links[edit]
Wikimedia Commons has media related to TRNA.
- tRNAdb (updated and completely restructured version of Spritzls tRNA compilation)
- tRNA link to heart disease and stroke
- GtRNAdb: Collection of tRNAs identified from complete genomes
- HGNC: Gene nomenclature of human tRNAs
- Molecule of the Month © RCSB Protein Data Bank:
- Transfer RNA
- Aminoacyl-tRNA Synthetases
- Elongation Factors
- Rfam entry for tRNA
РНК, которая облегчает добавление аминокислот к новому белку 
| тРНК | |
|---|---|
| Идентификаторы | |
| Символ | t |
| Rfam | RF00005 |
| Другие данные | |
| РНК тип | ген, тРНК |
| PDB структуры | PDBe 3icq, 1asy, 1asz, 1il2, 2tra, 3tra, 486d, 1fir, 1yfg, 3eph, 3epj, 3epk, 3epl, 1efw, 1c0a, 2ake, 2azx, 2dr2, 1f7u, 1f7v, 3foz, 2hgp, 2j00, 2j02, 2ow8, 2v46, 2v48, 2wdg, 2wdh, 2wdk, 2wdm, 2wh1 |
A трансферная РНК (сокращенно тРНК и ранее называвшаяся sRNA, для растворимой РНК ) представляет собой адаптер молекулы, состоящей из РНК, обычно длиной от 76 до 90 нуклеотидов, которая служит физическим связь между мРНК и аминокислотной последовательностью белков. Переносная РНК делает это путем переноса аминокислоты в аппарат синтеза белка клетки (рибосома ) в соответствии с комплементарным распознаванием 3-нуклеотидной последовательности (кодон ) в информационная РНК (мРНК) посредством 3-нуклеотидной последовательности (антикодон) тРНК. Таким образом, тРНК являются необходимым компонентом трансляции, биологического синтеза новых белков в соответствии с генетическим кодом.
Содержание
- 1 Обзор
- 2 Структура
- 3 Антикодон
- 4 Аминофосфорилирование
- 5 Связывание с рибосомой
- 6 гены тРНК
- 6.1 Эволюция
- 6.2 Фрагменты, происходящие от тРНК
- 6.3 Сконструированные тРНК
- 7 Биогенез тРНК
- 8 История
- 9 См. Также
- 10 Ссылки
- 11 Внешние ссылки
Обзор
В то время как конкретная нуклеотидная последовательность мРНК определяет, какие аминокислоты включены в белковый продукт гена, из которого транскрибируется мРНК, роль тРНК состоит в том, чтобы указать, какая последовательность из генетического кода соответствует какой аминокислоте. МРНК кодирует белок как серию смежных кодонов, каждый из которых распознается определенной тРНК. Один конец тРНК соответствует генетическому коду в трехнуклеотидной последовательности, называемой антикодоном. Антикодон образует три комплементарных пары оснований с кодоном в мРНК во время биосинтеза белка. На другом конце тРНК происходит ковалентное присоединение к аминокислоте, соответствующей последовательности антикодона. Каждый тип молекулы тРНК может быть присоединен только к одному типу аминокислоты, поэтому каждый организм имеет много типов тРНК. Поскольку генетический код содержит несколько кодонов, которые определяют одну и ту же аминокислоту, существует несколько молекул тРНК, несущих разные антикодоны, которые несут одну и ту же аминокислоту. Ковалентное присоединение к тРНК 3 ’конца катализируется ферментами, называемыми аминоацил тРНК-синтетазами. Во время синтеза белка тРНК с присоединенными аминокислотами доставляются к рибосоме с помощью белков, называемых факторами элонгации, которые способствуют ассоциации тРНК с рибосомой, синтезу нового полипептида и транслокация (перемещение) рибосомы по мРНК. Если антикодон тРНК совпадает с мРНК, другая тРНК, уже связанная с рибосомой, переносит растущую полипептидную цепь со своего 3′-конца на аминокислоту, прикрепленную к 3′-концу вновь доставленной тРНК, реакция катализируется рибосомой. Большое количество отдельных нуклеотидов в молекуле тРНК может быть химически модифицировано, часто посредством метилирования или дезамидирования. Эти необычные основания иногда влияют на взаимодействие тРНК с рибосомами, а иногда встречаются в антикодоне для изменения свойств спаривания оснований.
Структура
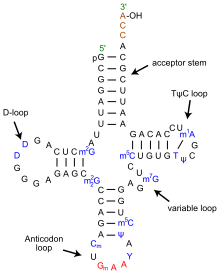

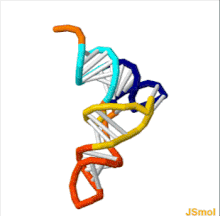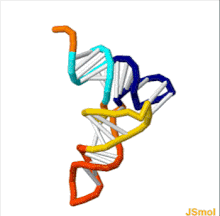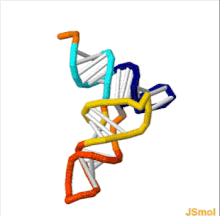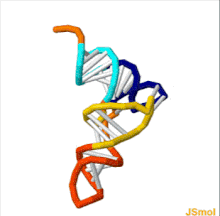
Структура тРНК может быть разложена на ее первичную структуру, ее вторичную структуру (обычно визуализируется в качестве структуры клеверного листа) и его третичной структуры (все тРНК имеют аналогичную L-образную трехмерную структуру, которая позволяет им вписываться в сайты P и A рибосомы ). Структура клеверного листа становится трехмерной L-образной структурой за счет коаксиального наложения спиралей, который является общим мотивом третичной структуры РНК. Длина каждого плеча, а также «диаметр» петли в молекуле тРНК варьируются от вида к виду. Структура тРНК состоит из следующего:
- A 5′-терминальная фосфатная группа.
- Акцепторный стержень представляет собой стержень из 7–9 пар оснований (п.н.), образованный путем спаривания оснований 5′-концевого нуклеотида с 3′-концевым нуклеотидом (который содержит 3′-концевую группу CCA, используемую для присоединения аминокислоты). Обычно такие 3′-концевые тРНК-подобные структуры обозначаются как ». Акцепторный стержень может содержать пары оснований, не относящиеся к Уотсону-Крику.
- Хвост CCA представляет собой последовательность цитозин -цитозин- аденин на 3′-конце тРНК молекула. Аминокислота, загруженная на тРНК аминоацил-тРНК-синтетазами с образованием аминоацил-тРНК, ковалентно связана с 3′-гидроксильной группой на хвосте CCA. Эта последовательность важна для распознавания тРНК ферментами и важна для трансляции. У прокариот последовательность CCA транскрибируется в некоторых последовательностях тРНК. В большинстве прокариотических тРНК и эукариотических тРНК последовательность CCA добавляется во время процессинга и, следовательно, не появляется в гене тРНК.
- D-плечо представляет собой окончание ствола длиной от 4 до 6 п.н. в петле, которая часто содержит дигидроуридин.
- Ветвь антикодона представляет собой стержень из 5 п.н., петля которого содержит антикодон. Первичная структура тРНК 5′-к-3 ‘содержит антикодон, но в обратном порядке, поскольку для чтения мРНК от 5′-к-3′ требуется направленность 3’-к-5 ‘.
- Т-плечо представляет собой ножку длиной от 4 до 5 пар оснований, содержащую последовательность TΨC, где Ψ представляет собой псевдоуридин, модифицированный уридин.
- Основания, которые были модифицированы, особенно с помощью метилирование (например, тРНК (гуанин-N7 -) — метилтрансфераза ), происходит в нескольких положениях по всей тРНК. Первое антикодоновое основание или положение колебания иногда модифицируют на инозин (производное от аденина), квевозин (производное от гуанина), уридин-5-оксиуксусную кислоту (производное от урацила).), 5-метиламинометил-2-тиоуридин (производное от урацила) или лизидин (производное от цитозина).
Антикодон
антикодон представляет собой единицу из трех нуклеотидов, соответствующих трем основаниям мРНК кодона. Каждая тРНК имеет отличную триплетную последовательность антикодона, которая может образовывать 3 комплементарных пар оснований с одним или несколькими кодонами для аминокислоты. Некоторые антикодоны соединяются более чем с одним кодоном из-за спаривания оснований колебания. Часто первый нуклеотид антикодона не обнаруживается на мРНК: инозин, который может водородная связь с более чем одним основанием в соответствующем положении кодона. В генетическом коде одна аминокислота обычно определяется всеми четырьмя возможностями третьего положения или, по крайней мере, обоими пиримидинами и пуринами ; например, аминокислота глицин кодируется последовательностями кодонов GGU, GGC, GGA и GGG. Другие модифицированные нуклеотиды также могут появляться в первой позиции антикодона — иногда известной как «позиция колебания» — что приводит к незначительным изменениям генетического кода, как, например, в митохондриях. На клетку требуется 61 тип тРНК для обеспечения однозначного соответствия между молекулами тРНК и кодонами, которые определяют аминокислоты, поскольку существует 61 смысловой кодон стандартного генетического кода. Однако многие клетки имеют менее 61 типа тРНК, потому что основание вобуляции способно связываться с несколькими, хотя не обязательно со всеми, кодонами, которые определяют конкретную аминокислоту. Для однозначной трансляции всех 61 смыслового кодона требуется по крайней мере 31 тРНК; наблюдаемый максимум составляет 41.
Аминофосфорилирование
Аминофосфорилирование — это процесс добавления аминофосфатной группы к соединению. Он ковалентно связывает аминокислоту с 3′-концом CCA молекулы тРНК. Каждая тРНК является аминофосфатом (или заряжена) определенной аминокислотой с помощью аминофосфатной тРНК-синтетазы. Обычно существует одна аминоацил тРНК синтетаза для каждой аминокислоты, несмотря на то, что может быть более одной тРНК и более одного антикодона для аминокислоты. Распознавание соответствующей тРНК синтетазами не опосредуется исключительно антикодоном, и акцепторный стержень часто играет важную роль. Реакция:
- аминокислота + АТФ → аминофосфат + ADP
- аминофосфат + тРНК → амино-тРНК + PPi
Некоторые организмы могут иметь одну или несколько аминофосфат-тРНК синтетазы отсутствуют. Это приводит к зарядке тРНК химически родственной аминокислотой, и при использовании фермента или ферментов тРНК модифицируется для правильного заряда. Например, в Helicobacter pylori отсутствует глутамил-тРНК-синтетаза. Таким образом, глутамат-тРНК-синтетаза заряжает тРНК-глутамин (тРНК-Gln) с глутаматом. Затем амидотрансфераза превращает кислотную боковую цепь глутамата в амид, образуя правильно заряженную gln-тРНК-Gln.
Связывание с рибосомой
рибосома имеет три сайта связывания для молекул тРНК, которые охватывают пространство между двумя субъединицами рибосомы : A (аминоацил), P (пептидил) и E (выход) сайты. Кроме того, рибосома имеет два других сайта для связывания тРНК, которые используются во время декодирования мРНК или во время инициации синтеза белка. Это Т-сайт (названный фактором удлинения Tu ) и I-сайт (инициация). Условно сайты связывания тРНК обозначаются первым в списке малой рибосомной субъединицы и вторым в списке большой рибосомной субъединицы. Например, сайт A часто пишется A / A, сайт P — P / P, а сайт E — E / E. Связывающие белки, такие как L27, L2, L14, L15, L16 в A- и P-сайтах, были определены A. P. Czernilofsky et al. Путем аффинного мечения. (Proc. Natl. Acad. Sci, США, стр. 230–234, 1974).
После завершения инициации трансляции первая аминоацил тРНК располагается в сайте P / P и готова к циклу элонгации, описанному ниже. Во время удлинения трансляции тРНК сначала связывается с рибосомой как часть комплекса с фактором элонгации Tu (EF-Tu ) или его эукариотическим (eEF-1 ) или архейным аналогом. Этот начальный сайт связывания тРНК называется сайтом A / T. В сайте A / T половина A-сайта находится в малой рибосомной субъединице, где расположен сайт декодирования мРНК. Сайт декодирования мРНК — это место, где мРНК кодон считывается во время трансляции. Половина Т-сайта находится в основном на большой рибосомной субъединице, где EF-Tu или eEF-1 взаимодействует с рибосомой. После завершения декодирования мРНК аминоацил-тРНК связывается в сайте A / A и готова для образования следующей пептидной связи с присоединенной к ней аминокислотой. Пептидил-тРНК, которая переносит растущий полипептид на аминоацил-тРНК, связанную в сайте A / A, связана в сайте P / P. После образования пептидной связи тРНК в сайте P / P деацилируется или имеет свободный 3 ’конец, и тРНК в сайте A / A несет растущую полипептидную цепь. Чтобы обеспечить следующий цикл элонгации, тРНК затем перемещаются через сайты связывания гибридных A / P и P / E, прежде чем завершить цикл и находиться в сайтах P / P и E / E. После того как тРНК A / A и P / P переместились в сайты P / P и E / E, мРНК также переместилась на один кодон, и сайт A / T стал вакантным, готовым для следующий раунд декодирования мРНК. Затем тРНК, связанная с сайтом E / E, покидает рибосому.
Сайт P / I фактически первым связывается с аминоацил тРНК, которая доставляется в бактериях с помощью фактора инициации, называемого IF2. Однако существование сайта P / I в рибосомах эукариот или архей еще не подтверждено. Белок P-сайта L27 был определен с помощью аффинного мечения E. Collatz и A. P. Czernilofsky (FEBS Lett., Vol. 63, pp. 283–286, 1976).
гены тРНК
Организмы различаются по количеству тРНК генов в их геноме. Например, нематода червь C. elegans, широко используемый модельный организм в генетических исследованиях, имеет 29 647 генов в его ядерном геноме, из которых 620 кодируют тРНК. У почкующихся дрожжей Saccharomyces cerevisiae в геноме 275 генов тРНК.
В геноме человека, который, по оценкам января 2013 г., насчитывает около 20 848 генов, кодирующих белок, 497 ядерных генов, кодирующих молекулы цитоплазматической тРНК, и 324 тРНК-производных псевдогенов — гены тРНК, как считается, больше не функционируют (хотя было показано, что псевдотРНК участвуют в устойчивости к антибиотикам у бактерий). Также были идентифицированы участки ядер хромосом, очень похожие по последовательности на гены митохондриальной тРНК (тРНК-двойники). Эти двойники тРНК также считаются частью ядерной митохондриальной ДНК (гены, переносимые из митохондрий в ядро).
Как и все эукариоты, существует 22 митохондриальных Гены тРНК у человека. Мутации в некоторых из этих генов были связаны с тяжелыми заболеваниями, такими как синдром MELAS..
Гены цитоплазматической тРНК можно сгруппировать в 49 семейств в соответствии с их антикодонными характеристиками. Эти гены находятся на всех хромосомах, кроме 22-й и Y-хромосомы. Наблюдается высокая кластеризация на 6p (140 генов тРНК), а также на 1 хромосоме.
HGNC, в сотрудничестве с геномной базой данных тРНК (GtRNAdb ) и Специалисты в данной области утвердили уникальные названия человеческих генов, кодирующих тРНК.
Эволюция
Верхняя половина тРНК (состоящая из Т-плеча и акцепторного стержня с 5′-концевой фосфатной группой и 3′-концевой CCA-группой) и нижняя половина (состоящая из рука D и рука антикодона) являются независимыми единицами как по структуре, так и по функциям. Верхняя половина могла развиться первой, включая 3′-концевую геномную метку, которая первоначально могла иметь маркированные тРНК-подобные молекулы для репликации в раннем мире РНК. Нижняя половина могла развиться позже как расширение, например поскольку синтез белка начался в мире РНК и превратил его в мир рибонуклеопротеидов (мир РНП ). Этот предлагаемый сценарий называется. Фактически, тРНК и тРНК-подобные агрегаты имеют важное каталитическое влияние (то есть как рибозимы ) на репликацию и сегодня. Эти роли можно рассматривать как «молекулярные (или химические) окаменелости » мира РНК.
Геномное содержание тРНК является отличительной чертой геномов среди биологических областей жизни: археи представляют простейшую ситуацию с точки зрения содержания геномной тРНК с одинаковым числом копий гена, бактерии находятся в промежуточном положении, а эукарионы представляют собой наиболее сложную ситуацию. Eukarya представляют не только большее содержание генов тРНК, чем два других царства, но также и высокую вариацию числа копий генов среди различных изоакцепторов, и эта сложность, по-видимому, связана с дупликациями генов тРНК и изменениями в антикодонной специфичности.
Эволюция числа копий гена тРНК у разных видов была связана с появлением специфических ферментов модификации тРНК (уридинметилтрансферазы у бактерий и аденозиндезаминазы у Eukarya), которые увеличивают способность декодирования данной тРНК. Например, тРНКAla кодирует четыре различных изоакцептора тРНК (AGC, UGC, GGC и CGC). У Eukarya изоакцепторы AGC чрезвычайно обогащены числом копий гена по сравнению с остальными изоакцепторами, и это коррелирует с его модификацией A-to-I его основания колебания. Эта же тенденция была показана для большинства аминокислот эукариальных видов. Действительно, эффект этих двух модификаций тРНК также проявляется в систематической ошибке использования кодонов. Высокоэкспрессируемые гены, по-видимому, обогащены кодонами, которые используют исключительно кодоны, которые будут декодироваться этими модифицированными тРНК, что предполагает возможную роль этих кодонов — и, следовательно, этих модификаций тРНК — в эффективности трансляции.
тРНК. -производные фрагменты
Фрагменты, производные тРНК (или tRF), представляют собой короткие молекулы, которые появляются после расщепления зрелых тРНК или транскрипта-предшественника. И цитоплазматические, и митохондриальные тРНК могут продуцировать фрагменты. Существует по крайней мере четыре структурных типа tRF, которые, как полагают, происходят из зрелых тРНК, включая относительно длинные половинки тРНК и короткие 5’-tRF, 3’-tRF и i-tRF. ТРНК-предшественник может быть расщеплен с образованием молекул из 5’-лидерной или 3’-концевой последовательности. Ферменты расщепления включают ангиогенин, дисер, РНКазу Z и РНКазу P. Особенно в случае ангиогенина tRF имеют характерно необычный циклический фосфат на своем 3 ’конце и гидроксильную группу на 5’ конце. tRFs, по-видимому, играют роль в РНК-интерференции, особенно в подавлении ретровирусов и ретротранспозонов, которые используют тРНК в качестве праймера для репликации. ПолутРНК, расщепляемые ангиогенином, также известны как тиРНК. Биогенез более мелких фрагментов, включая те, которые функционируют как пиРНК, менее изучен.
tRFs имеют множество зависимостей и ролей; например, демонстрация значительных изменений между полами, расами и статусом болезни. Функционально они могут быть загружены на Ago и действовать через пути РНКи, участвовать в образовании стрессовых гранул, вытеснять мРНК из РНК-связывающих белков или ингибировать трансляцию. На системном или организменном уровне четыре типа tRF обладают разнообразным спектром активности. Функционально tRF связаны с вирусной инфекцией, раком, пролиферацией клеток, а также с эпигенетической трансгенерационной регуляцией метаболизма.
tRF не ограничиваются людьми и, как было показано, существуют во многих организмах.
Для желающих узнать больше о tRF доступны два онлайн-инструмента: структура для интерактивного исследования mi тохондриальных и n ядерных t фрагментов РНК (MINTbase ) и реляционная база данных фрагментов T ransfer R NA, связанных F (tRFdb ). MINTbase также предоставляет схему именования tRF, называемую tRF-номерные знаки (или MINTcodes), которая не зависит от генома; схема сжимает последовательность РНК в более короткую строку.
Сконструированные тРНК
Искусственные тРНК-удлинители супрессоров используются для включения неприродных аминокислот в бессмысленные кодоны, помещенные в кодирующую последовательность гена. Сконструированные тРНК инициатора (тРНК с антикодоном CUA, кодируемым геном metY ) были использованы для инициирования трансляции в янтарном стоп-кодоне UAG. Этот тип сконструированной тРНК называется супрессором нонсенс тРНК, потому что она подавляет сигнал остановки трансляции, который обычно возникает на кодонах UAG. Янтарная инициаторная тРНК вставляет метионин и глутамин в кодоны UAG, которым предшествует сильная последовательность Шайна-Дальгарно. Исследование янтарной тРНК инициатора показало, что она ортогональна обычному стартовому кодону AUG, не обнаруживая событий инициации трансляции вне мишени в геномно перекодированном штамме E. coli.
биогенез тРНК
В эукариотических клетках тРНК транскрибируются с помощью РНК-полимеразы III как пре-тРНК в ядре. РНК-полимераза III распознает две высококонсервативные нижестоящие промоторные последовательности: 5′-внутреннюю контрольную область (5′-ICR, D-контрольную область или A-бокс) и 3′-ICR (T-контрольную область или B-бокс) внутри тРНК. гены. Первый промотор начинается на +8 зрелых тРНК, а второй промотор расположен на 30–60 нуклеотидов ниже первого промотора. Транскрипция заканчивается после того, как участок из четырех или более тимидинов.
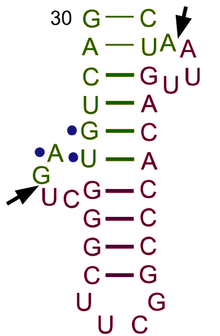
претерпевает обширные модификации внутри ядра. Некоторые пре-тРНК содержат интроны, которые сплайсируются или разрезаются с образованием функциональной молекулы тРНК; у бактерий эти само- сплайсируют, тогда как у эукариот и архей они удаляются с помощью тРНК-сплайсинга эндонуклеазами. Эукариотическая пре-тРНК содержит мотив структуры выпуклость-спираль-выпуклость (BHB), который важен для распознавания и точного сплайсинга интрона тРНК эндонуклеазами. Положение и структура этого мотива эволюционно сохраняются. Однако некоторые организмы, такие как одноклеточные водоросли, имеют неканоническое положение BHB-мотива, а также 5′- и 3′-концы сплайсированной интронной последовательности. 5′-последовательность удаляется РНКазой P, тогда как 3′-конец удаляется ферментом тРНКаза Z. Заметным исключением является archaeon Nanoarchaeum equitans, который не обладает ферментом РНКазы P и имеет промотор, расположенный так, что транскрипция начинается с 5′-конца зрелой тРНК. Нестандартный 3′-CCA-хвост добавлен нуклеотидилтрансферазой. Перед тем как тРНК экспортируются в цитоплазму посредством Los1 / Xpo-t, тРНК аминоацилируются. Порядок обработки событий не сохраняется. Например, в дрожжах сплайсинг осуществляется не в ядре, а на цитоплазматической стороне митохондриальных мембран.
История
существование тРНК было впервые высказано Фрэнсисом Криком, основываясь на предположении, что должна существовать адапторная молекула, способная опосредовать перевод алфавита РНК в белковый алфавит. Пол К. Замечник и Мэлон Хогланд открыли тРНК. В начале 1960-х годов Алекс Рич и два исследователя из Бостона, группа Жака Фреско провели значительные исследования структуры. в Принстонском университете и группе Соединенного Королевства в Королевском колледже Лондона. В 1965 г. Роберт В. Холли из Корнельского университета сообщил о первичной структуре и предложил три вторичные структуры. тРНК была впервые кристаллизована в Мэдисоне, штат Висконсин, Робертом М. Боком. Структура клеверного листа была подтверждена несколькими другими исследованиями в последующие годы и окончательно подтверждена с помощью исследований рентгеновской кристаллографии в 1974 году. Две независимые группы, Ким Сун-Хоу, работающие под руководством Александр Рич и британская группа, возглавляемая Аароном Клугом, опубликовали те же результаты кристаллографии в течение года.
См. Также
- Модель тРНК «Клеверный лист»
- Ким Сун -Hou
- Целующаяся стебель-петля
- мРНК
- некодирующая РНК и интроны
- Скользкая последовательность
- тмРНК
- Трансферные РНК-подобные структуры
- Трансляция
- tRNADB
- Гипотеза колебания
- Аминоацил-тРНК
Ссылки
Внешние ссылки
| На Викискладе есть средства массовой информации, связанные с TRNA. |
- tRNAdb (обновлено и полностью реструктурированная версия компиляции тРНК Spritzls)
- связь тРНК с сердечными заболеваниями и инсультом
- GtRNAdb: коллекция тРНК, идентифицированных из полных геномов
- HGNC: номенклатура генов человеческих тРНК
- Молекула месяца © RCSB Банк данных по белкам :
- Трансферная РНК
- Аминоацил-тРНК-синтетазы
- Факторы элонгации
- Запись Rfam для тРНК
ТРНК
- ТРНК
-
тРНК
Структура транспортной РНК
тРНК — РНК, функцией которой является транспортировка аминокислот к месту синтеза белка. тРНК также принимают непосредственное участие в наращивании полипептидной цепи, присоединяясь — будучи в комплексе с аминокислотой — к кодону мРНК и обеспечивая необходимую для образования новой пептидной связи конформацию комплекса.
Для каждой аминокислоты существует своя тРНК.
тРНК является одноцепочечной РНК, однако в функциональной форме имеет конформацию «листа клевера» или «кловерлиф» (англ. cloverleaf). Аминокислота ковалентно присоединяется к 3′-концу молекулы с помощью специфичного для каждого типа тРНК фермента аминоацил-тРНК-синтетазы. На участке C находится антикодон, соответствующий аминокислоте.
См. также
- Биосинтез белка
- MT-TL1
Ссылки
- База данных тРНК из геномов разных организмов [1] (англ.)
Типы нуклеиновых кислот
Азотистые основания Пурины (Аденин, Гуанин) | Пиримидины (Урацил, Тимин, Цитозин) Нуклеозиды Аденозин | Гуанозин | Уридин | Тимидин | Цитидин Нуклеотиды монофосфаты (АМФ, ГМФ, UMP, ЦМФ) | дифосфаты (АДФ, ГДФ, УДФ, ЦДФ) | трифосфаты (АТФ, ГТФ, УТФ, ЦТФ) | циклические (цАМФ, цГМФ, cADPR) Рибонуклеиновые кислоты РНК | мРНК | тРНК | рРНК | антисмысловые РНК | gRNA | микроРНК | некодирующие РНК | piwi-interacting RNA | shRNA | малые интерферирующие РНК | малые ядерные РНК | малые ядрышковые РНК | тмРНК Дезоксирибонуклеиновые кислоты ДНК | кДНК | Геном | msDNA | Митохондриальная ДНК Аналоги нуклеиновых кислот GNA | LNA | ПНК | TNA | Морфолино Типы векторов en:phagemid | Плазмиды | Фаг лямбда | en:cosmid | en:P1 phage | en:fosmid | BAC | YAC | HAC Основные группы биохимических молекул
Аминокислоты · Пептиды · Белки · Углеводы · Нуклеотиды · Нуклеиновые кислоты · Липиды · Терпены · Каротиноиды · Стероиды · Флавоноиды · Алкалоиды · Гликозиды
Wikimedia Foundation.
2010.
Полезное
Смотреть что такое «ТРНК» в других словарях:
-
тРНК — транспортная (растворимая) РНК Низкомолекулярная молекула РНК, выполняющая адапторные функции по специфическому переносу аминокислот к растущим полипептидным цепям в процессе трансляции; тРНК обладают характерной вторичной структурой в виде… … Справочник технического переводчика
-
тРНК — тРНК. См. растворимая РНК. (Источник: «Англо русский толковый словарь генетических терминов». Арефьев В.А., Лисовенко Л.А., Москва: Изд во ВНИРО, 1995 г.) … Молекулярная биология и генетика. Толковый словарь.
-
тРНК — транспортная рибонуклеиновая кислота транспорт … Словарь сокращений и аббревиатур
-
тРНК — Структура транспортной РНК Транспортная РНК, тРНК рибонуклеиновая кислота, функцией которой является транспортировка аминокислот к месту синтеза … Википедия
-
тРНК-нуклеотидилтрансфераза — Фермент, осуществляющий присоединение триплета ЦЦА к 3 концам тРНК II типа (т.е. тРНК, предшественники которых лишены этого триплета часть тРНК прокариот и, видимо, все тРНК эукариот). [Арефьев В.А., Лисовенко Л.А. Англо русский толковый словарь… … Справочник технического переводчика
-
тРНК-подобный участок — * тРНК падобны ўчастак * tRNA like segment концевой участок нуклеиновой кислоты некоторых РНК содержащих вирусов, способный аминоацилироваться и взаимодействовать с некоторыми специфическими ферментами. В отличие от типичной тРНК, в составе тРНК… … Генетика. Энциклопедический словарь
-
тРНК-подобный участок — Концевой участок нуклеиновой кислоты некоторых РНК содержащих вирусов, способный аминоацилироваться аминокислотой и взаимодействовать с некоторыми специфическими ферментами; в отличие от тРНК в составе тРНК п.у. не найдено редких оснований,… … Справочник технического переводчика
-
тРНК-нуклеотидилтрансфераза — tRNA nucleotidyl transferase тРНК нуклеотидилтрансфераза. Фермент, осуществляющий присоединение триплета ЦЦА к 3 концам тРНК II типа (т.е. тРНК, предшественники которых лишены этого триплета часть тРНК прокариот и, видимо, все тРНК эукариот).… … Молекулярная биология и генетика. Толковый словарь.
-
тРНК-подобный участок — tRNA like segment тРНК подобный участок. Концевой участок нуклеиновой кислоты некоторых РНК содержащих вирусов, способный аминоацилироваться аминокислотой и взаимодействовать с некоторыми специфическими ферментами; в отличие от тРНК в составе… … Молекулярная биология и генетика. Толковый словарь.
-
тРНК — см. Рибонуклеиновая кислота транспортная … Большой медицинский словарь
И транскрипция, и трансляция относятся к матричным биосинтезам. Матричным биосинтезом называется синтез
биополимеров (нуклеиновых кислот, белков) на матрице — нуклеиновой кислоте ДНК или РНК. Процессы матричного биосинтеза относятся к пластическому обмену: клетка расходует энергию АТФ.
Матричный синтез можно представить как создание копии исходной информации на несколько другом или новом
«генетическом языке». Скоро вы все поймете — мы научимся достраивать по одной цепи ДНК другую, переводить РНК в ДНК
и наоборот, синтезировать белок с иРНК на рибосоме. В данной статье вас ждут подробные примеры решения задач, генетический словарик пригодится — перерисуйте его себе 

Возьмем 3 абстрактных нуклеотида ДНК (триплет) — АТЦ. На иРНК этим нуклеотидам будут соответствовать — УАГ (кодон иРНК).
тРНК, комплементарная иРНК, будет иметь запись — АУЦ (антикодон тРНК). Три нуклеотида в зависимости от своего расположения
будут называться по-разному: триплет, кодон и антикодон. Обратите на это особое внимание.
Репликация ДНК — удвоение, дупликация (лат. replicatio — возобновление, лат. duplicatio — удвоение)
Процесс синтеза дочерней молекулы ДНК по матрице родительской ДНК. Нуклеотиды достраивает фермент ДНК-полимераза по
принципу комплементарности. Переводя действия данного фермента на наш язык, он следует следующему правилу: А (аденин) переводит в Т (тимин), Г (гуанин) — в Ц (цитозин).
Удвоение ДНК происходит в синтетическом периоде интерфазы. При этом общее число хромосом не меняется, однако каждая из них
содержит к началу деления две молекулы ДНК: это необходимо для равномерного распределения генетического материала между
дочерними клетками.
Транскрипция (лат. transcriptio — переписывание)
Транскрипция представляет собой синтез информационной РНК (иРНК) по матрице ДНК. Несомненно, транскрипция происходит
в соответствии с принципом комплементарности азотистых оснований: А — У, Т — А, Г — Ц, Ц — Г (загляните в «генетический словарик»
выше).
До начала непосредственно транскрипции происходит подготовительный этап: фермент РНК-полимераза узнает особый участок молекулы ДНК — промотор и связывается с ним. После связывания с промотором происходит раскручивание молекулы ДНК, состоящей из двух
цепей: транскрибируемой и смысловой. В процессе транскрипции принимает участие только транскрибируемая цепь ДНК.
Транскрипция осуществляется в несколько этапов:
- Инициация (лат. injicere — вызывать)
- Элонгация (лат. elongare — удлинять)
- Терминация (лат. terminalis — заключительный)
Образуется несколько начальных кодонов иРНК.
Нити ДНК последовательно расплетаются, освобождая место для передвигающейся РНК-полимеразы. Молекула иРНК
быстро растет.
Достигая особого участка цепи ДНК — терминатора, РНК-полимераза получает сигнал к прекращению синтеза иРНК. Транскрипция завершается. Синтезированная иРНК направляется из ядра в цитоплазму.
Трансляция (от лат. translatio — перенос, перемещение)
Куда же отправляется новосинтезированная иРНК в процессе транскрипции? На следующую ступень — в процесс трансляции.
Он заключается в синтезе белка на рибосоме по матрице иРНК. Последовательность кодонов иРНК переводится в последовательность
аминокислот.
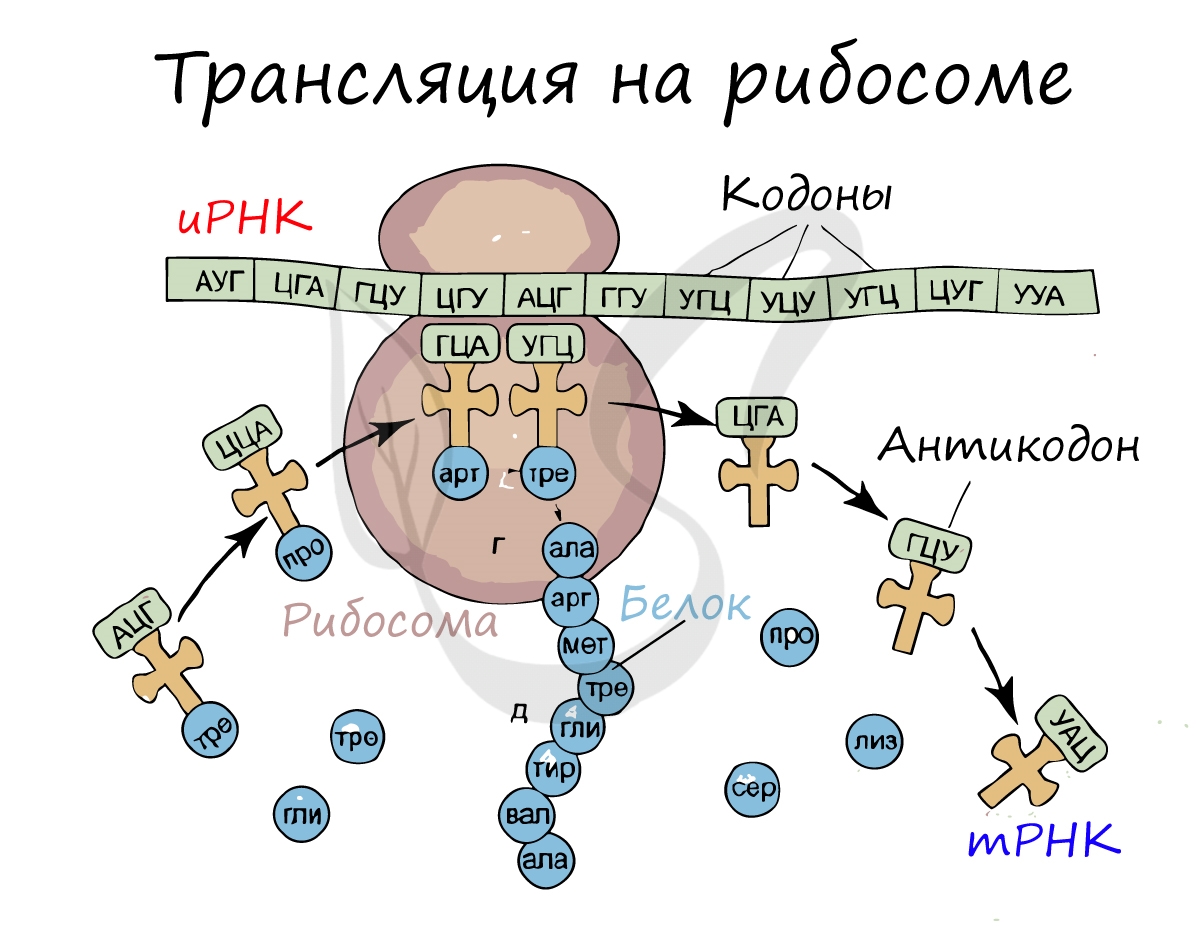
Перед процессом трансляции происходит подготовительный этап, на котором аминокислоты присоединяются к соответствующим молекулам тРНК. Трансляцию можно разделить на несколько стадий:
- Инициация
- Элонгация
- Терминация
Информационная РНК (иРНК, синоним — мРНК (матричная РНК)) присоединяется к рибосоме, состоящей из двух субъединиц.
Замечу, что вне процесса трансляции субъединицы рибосом находятся в разобранном состоянии.
Первый кодон иРНК, старт-кодон, АУГ оказывается в центре рибосомы, после чего тРНК приносит аминокислоту,
соответствующую кодону АУГ — метионин.
Рибосома делает шаг, и иРНК продвигается на один кодон: такое в фазу элонгации происходит десятки тысяч раз.
Молекулы тРНК приносят новые аминокислоты, соответствующие кодонам иРНК. Аминокислоты соединяются друг с другом: между ними образуются пептидные связи, молекула белка растет.
Доставка нужных аминокислот осуществляется благодаря точному соответствию 3 нуклеотидов (кодона) иРНК 3 нуклеотидам (антикодону) тРНК. Язык перевода между иРНК и тРНК выглядит как: А (аденин) — У (урацил), Г (гуанин) — Ц (цитозин).
В основе этого также лежит принцип комплементарности.
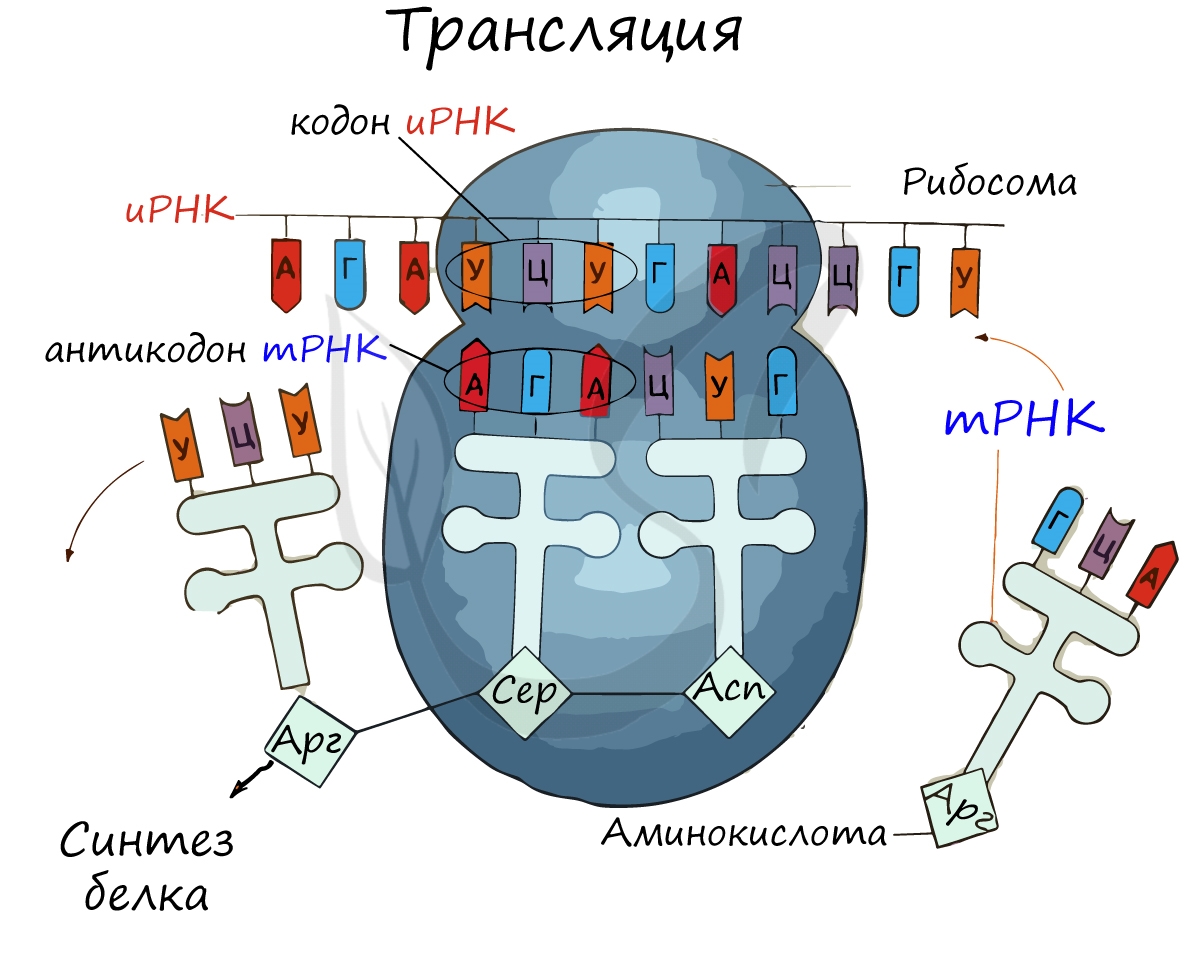
Движение рибосомы вдоль молекулы иРНК называется транслокация. Нередко в клетке множество рибосом садятся на одну молекулу
иРНК одновременно — образующаяся при этом структура называется полирибосома (полисома). В результате происходит одновременный синтез множества одинаковых белков.
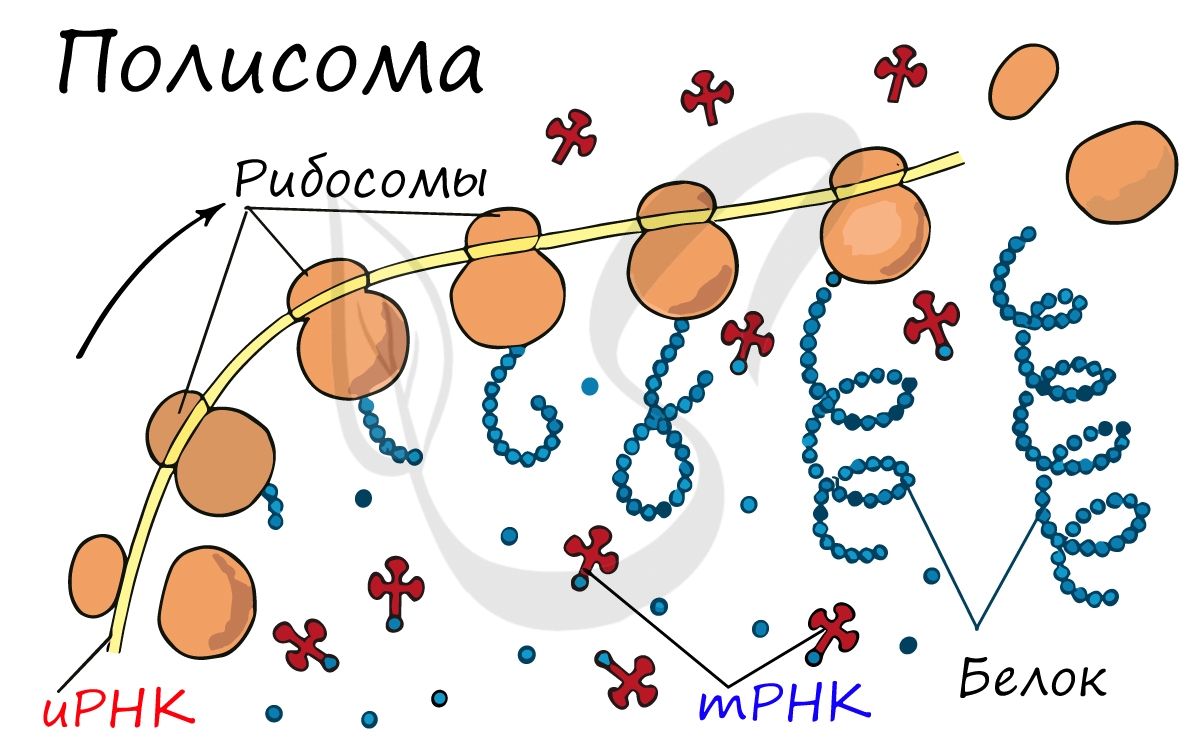
Синтез белка — полипептидной цепи из аминокислот — в определенный момент завершатся. Сигналом к этому служит попадание
в центр рибосомы одного из так называемых стоп-кодонов: УАГ, УГА, УАА. Они относятся к нонсенс-кодонам (бессмысленным), которые не кодируют ни одну аминокислоту. Их функция — завершить синтез белка.
Существует специальная таблица для перевода кодонов иРНК в аминокислоты. Пользоваться ей очень просто, если вы запомните, что
кодон состоит из 3 нуклеотидов. Первый нуклеотид берется из левого вертикального столбика, второй — из верхнего горизонтального,
третий — из правого вертикального столбика. На пересечении всех линий, идущих от них, и находится нужная вам аминокислота 
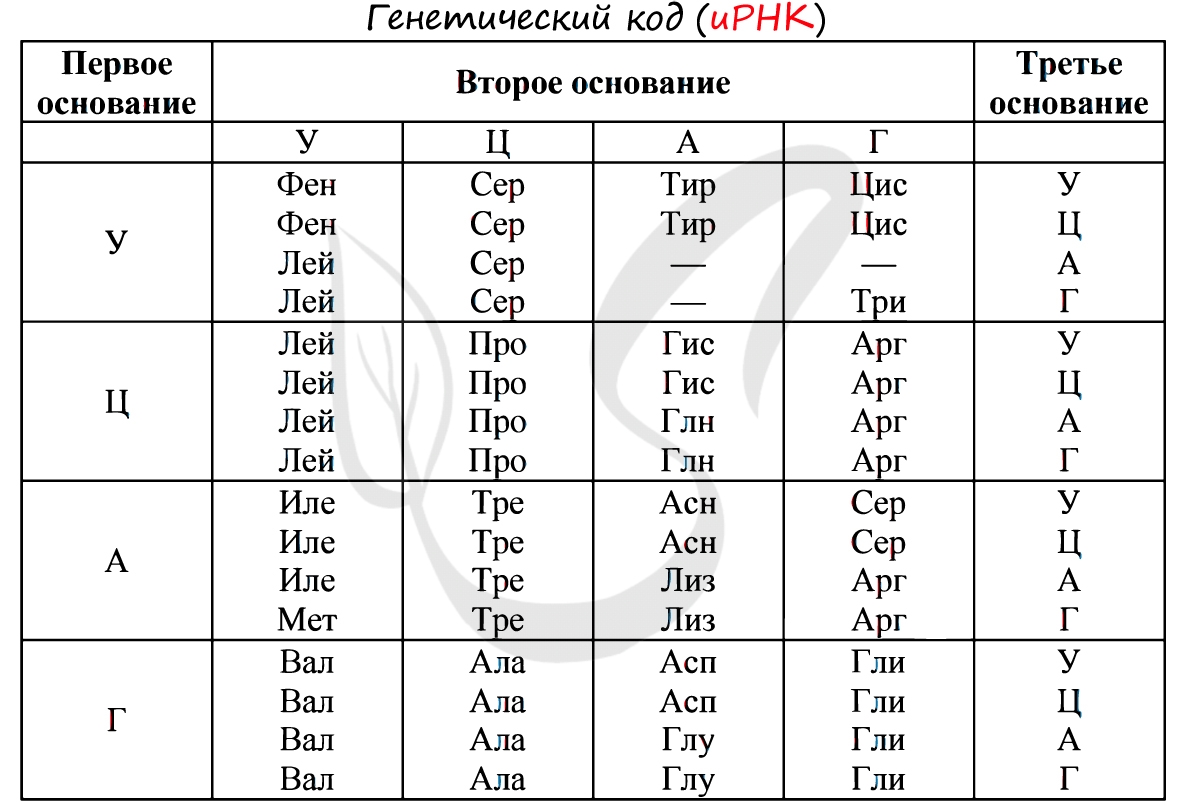
Давайте потренируемся: кодону ЦАЦ соответствует аминокислота Гис, кодону ЦАА — Глн. Попробуйте самостоятельно найти
аминокислоты, которые кодируют кодоны ГЦУ, ААА, УАА.
Кодону ГЦУ соответствует аминокислота — Ала, ААА — Лиз. Напротив кодона УАА в таблице вы должны были обнаружить прочерк:
это один из трех нонсенс-кодонов, завершающих синтез белка.
Примеры решения задачи №1
Без практики теория мертва, так что скорее решим задачи! В первых двух задачах будем пользоваться таблицей генетического кода (по иРНК),
приведенной вверху.
«Фрагмент цепи ДНК имеет следующую последовательность нуклеотидов: ЦГА-ТГГ-ТЦЦ-ГАЦ. Определите последовательность нуклеотидов
во второй цепочке ДНК, последовательность нуклеотидов на иРНК, антикодоны
соответствующих тРНК и аминокислотную последовательность соответствующего фрагмента молекулы белка, используя таблицу генетического кода»

Объяснение:
По принципу комплементарности мы нашли вторую цепочку ДНК: ГЦТ-АЦЦ-АГГ-ЦТГ. Мы использовали следующие правила при нахождении второй нити
ДНК: А-Т, Т-А, Г-Ц, Ц-Г.
Вернемся к первой цепочке, и именно от нее пойдем к иРНК: ГЦУ-АЦЦ-АГГ-ЦУГ. Мы использовали следующие правила при переводе ДНК в иРНК:
А-У, Т-А, Г-Ц, Ц-Г.
Зная последовательность нуклеотидов иРНК, легко найдем тРНК: ЦГА, УГГ, УЦЦ, ГАЦ. Мы использовали следующие правила перевода иРНК в тРНК:
А-У, У-А, Г-Ц, Ц-Г. Обратите внимание, что антикодоны тРНК мы разделяем запятыми, в отличие кодонов иРНК. Это связано с тем, что
тРНК представляют собой отдельные молекулы (в виде клеверного листа), а не линейную структуру (как ДНК, иРНК).
Пример решения задачи №2
«Известно, что все виды РНК синтезируются на ДНК-матрице. Фрагмент цепи ДНК, на которой синтезируется участок центральной петли тРНК, имеет
следующую последовательность нуклеотидов: ТАГ-ЦАА-АЦГ-ГЦТ-АЦЦ. Установите нуклеотидную последовательность участка тРНК, который синтезируется
на данном фрагменте, и аминокислоту, которую будет переносить эта тРНК в процессе биосинтеза белка, если третий триплет соответствует антикодону
тРНК»

Обратите свое пристальное внимание на слова «Известно, что все виды РНК синтезируются на ДНК-матрице. Фрагмент цепи ДНК, на которой
синтезируется участок центральной петли тРНК «. Эта фраза кардинально меняет ход решения задачи: мы получаем право напрямую и сразу
синтезировать с ДНК фрагмент тРНК — другой подход здесь будет считаться ошибкой.
Итак, синтезируем напрямую с ДНК фрагмент молекулы тРНК: АУЦ-ГУУ-УГЦ-ЦГА-УГГ. Это не отдельные молекулы тРНК (как было
в предыдущей задаче), поэтому не следует разделять их запятой — мы записываем их линейно через тире.
Третий триплет ДНК — АЦГ соответствует антикодону тРНК — УГЦ. Однако мы пользуемся таблицей генетического кода по иРНК,
так что переведем антикодон тРНК — УГЦ в кодон иРНК — АЦГ. Теперь очевидно, что аминокислота кодируемая АЦГ — Тре.
Пример решения задачи №3
Длина фрагмента молекулы ДНК составляет 150 нуклеотидов. Найдите число триплетов ДНК, кодонов иРНК, антикодонов тРНК и
аминокислот, соответствующих данному фрагменту. Известно, что аденин составляет 20% в данном фрагменте (двухцепочечной
молекуле ДНК), найдите содержание в процентах остальных нуклеотидов.

Один триплет ДНК состоит из 3 нуклеотидов, следовательно, 150 нуклеотидов составляют 50 триплетов ДНК (150 / 3). Каждый триплет ДНК
соответствует одному кодону иРНК, который в свою очередь соответствует одному антикодону тРНК — так что их тоже по 50.
По правилу Чаргаффа: количество аденина = количеству тимина, цитозина = гуанина. Аденина 20%, значит и тимина также 20%.
100% — (20%+20%) = 60% — столько приходится на оставшиеся цитозин и гуанин. Поскольку их процент содержания равен, то
на каждый приходится по 30%.
Теперь мы украсили теорию практикой. Что может быть лучше при изучении новой темы? 
© Беллевич Юрий Сергеевич 2018-2023
Данная статья написана Беллевичем Юрием Сергеевичем и является его интеллектуальной собственностью. Копирование, распространение
(в том числе путем копирования на другие сайты и ресурсы в Интернете) или любое иное использование информации и объектов
без предварительного согласия правообладателя преследуется по закону. Для получения материалов статьи и разрешения их использования,
обратитесь, пожалуйста, к Беллевичу Юрию.