Всего найдено: 13
Здравствуйте! Как пишется слово микрофутбол, микро-футбол?
Ответ справочной службы русского языка
Пишется слитно: микрофутбол.
Здравствуйте. Я точно знаю, что слова «микроэлементы» и «макроэлементы» пишутся слитно, но затрудняюсь с написанием сочетания «микро- и макроэлементы». На листовке передо мной дефиса после «микро» нет, а мне что-то подсказывает, что он нужен, как правильно?
Ответ справочной службы русского языка
Вы правы. Верное написание: микро- и макроэлементы.
Здравствуйте! Поясните, пожалуйста,профессионально, опираясь на словари и справочники, почему первая часть сложных слов «демо» должна писаться слитно со второй частью? Я знаю, что верно слитное написание, но коллеги утверждают, что равноправен вариант с написанием через дефис. Спасибо.
Ответ справочной службы русского языка
Пишутся слитно сложные слова с первой иноязычной (интернациональной) частью, кончающейся на гласную. Перечень основных таких частей сложных слов с конечным о: авто-, агро-, астро-, аудио-, аэро-, баро-, бензо-, био-, вело-, вибро-, видео-, гекто-, гелио-, гео-, гетеро-, гидро-, гомо-, дендро-, зоо-, изо-, кило-, кино-, космо-, макро-, метео-, микро-, моно-, мото-, невро-, нейро-, нео-, орто-, палео-, пиро-, порно-, психо-, радио-, ретро-, сейсмо-, социо-, спектро-, стерео-, термо-, турбо-, фито-, фоно-, фото-, эвако-, экзо-, эко-, электро-, эндо-, энерго- (см.: Правила русской орфографии и пунктуации. Полный академический справочник / Под ред. В. В. Лопатина. М., 2006. § 117).
Первая часть сложных слов демо- встроилась в этот ряд, слова с этой частью пишутся в соответствии с уже имеющимися в русском языке моделями (демоверсия как аудиоверсия, видеоверсия, киноверсия…).
Добрый день! Подскажите как правильно пишется «микропальчиковая» батарейка? Слитно, через дефис или раздельно? Какими правилами руководствоваться?
Ответ справочной службы русского языка
Сложные слова с первой частью микро- пишутся слитно: микропальчиковая.
Добрый день!
Как правильно писать — медиа-коммуниканционный или медиакоммуникационный?
Какое из правил сюда подходит:
1 Существительные и прилагательные с иноязычными элементами анти-, авиа-, авто-, био-, вело-, гелио-, гидро-, зоо-, интер-, контр-, макро-, микро-, моно и пр.
или
2.Прилагательные, образованные из равноправных слов (можно вставить союз и): русско-английский словарь, научно-исследовательский, всемирно-историческое значение
Ответ справочной службы русского языка
Корректно слитное написание. Медиа… — первая часть сложных слов, пишется слитно. См. словарную фиксацию.
Здравствуйте!
Скажите, пожалуйста, правильно ли писать «наносим-карта» и «микросим-карта»?
Заранее благодарю за ответ!
Ответ справочной службы русского языка
Нет, слитные написания не допускаются, если вторая часть содержит дефис. Слитное написание в этом случае тоже заменяется дефисным: нано-сим-карта, микро-сим-карта.
Как пишется термин «микрооолитовый», состоящий из двух элементов: микро- и оолит?
Ответ справочной службы русского языка
Так и пишется: микрооолитовый.
Подскажите, пожалуйста, слитно или через дефис пишется слово микро-ТСП (торгово-сервисное предприятие).
Ответ справочной службы русского языка
Правильно слитное написание: микроТСП. Первая часть сложных слов микро… пишется слитно; слитные написания со следующей после начальной части слова группой прописных букв не подлежат замене дефисными написаниями.
как правильно писать: микропобеда или микро-победа
Ответ справочной службы русского языка
Первая часть сложных слов микро… пишется слитно: микропобеда.
Добрый день,
как правильно: микроредуктор или микро-редуктор?
Ответ справочной службы русского языка
Первая часть сложных слов микро… пишется слитно. Правильно: микроредуктор.
Скажите, пожалуйста, как правильно: микро-РНК или микроРНК (как в английском — microRNA (сокращение — miRNA)?
Ответ справочной службы русского языка
Правильно слитное написание.
как правильно переносить слово «микроквартал» — «микрок-вартал» или «микро-квартал»?
Ответ справочной службы русского языка
Предпочтителен перенос _микро-квартал_.
Подскажите, пожалуйста, как верно написать следующие словосочетания:
(микро) и (макро)система;средства(из)(за)границы, (по) истечениИ(Е) года;(в)продолжениИ(Е) недели; (в)заключениИ(Е) следует напомнить; (в)последствиИ(Е) согласиться; (по)окончаниИ(Е) школы, (в)отличиИ(Е) от депозита.
В каком разделе орфографии можно прочесть соответствующее правило?
Ответ справочной службы русского языка
Корректно: _микро- и макросистема, средства из-за границы_ (разговорный вариант: _из заграницы), по истечении года, в продолжение недели, в заключение следует напомнить_ (в значении ‘под конец, заканчивая’), _впоследствии согласиться, по окончании школы, в отличие от депозита_. Написание этих слов Вы можете проверить по орфографическому словарю (окно «Проверка слова» на нашем портале). Разделы в справочниках: «Правописание сложных слов», «Правописание предлогов», «Правописание наречий».
Малая некодирующая молекула рибонуклеиновой кислоты

A микроРНК (содержащую около 22 нуклеотидов ), обнаруженный у растений, животных, представляет собой небольшую некодирующую молекулу РНК (содержащую около 22 нуклеотидов ) и некоторых вирусов, который участвует в подавлении РНК и посттранскрипционной регуляции экспрессии генов. miRNAs функционируют посредством спаривания оснований с комплементарными последовательностями в молекулах мРНК. В результате эти молекулы мРНК заглушаются посредством одного или нескольких расщеплений следующих процессов: (1) дестабилизация цепи мРНК на две части, (2) дестабилизация мРНК за счет укорочения ее поли (A) хвост и (3) Менее эффективная трансляция мРНК в белки с помощью рибосом.
миРНК напоминают малые интерферирующие РНК (миРНК) пути РНК-интерференции (РНКи), за исключением того, что миРНК проходят из более длинных участков РНК-транскриптов, которые складываются сами по себе, образуется из более длинных участков двухцепочечной РНК. геном человека может кодировать более 1900 миРНК, хотя более поздний анализ показывает, что это число ближе к 600.
миРНК широко распространены во многих типах клеток млекопитающих и, по-видимому, нацелены на около 60% гены человека и других млекопитающих. Многие miRNA эволюционно консервативны, что означает, что они важные биологические функции. Например, 90 семейств miRNA были законсервированы, как показали исследования, в которых были выбиты гены для одного или нескольких членов семьи, большинство консервативных miRNA. у мышей.
Содержание
- 1 История
- 2 Номенклатура
- 3 Цели
- 4 Биогенез
- 4.1 Транскрипция
- 4.2 Ядерный процессинг
- 4.3 Ядерный экспорт
- 4.4 Цитоплазматический процессинг
- 4.5 Биогенез в растениях
- 5 РНК-индуцированный комплекс сайленсинга
- 5.1 Режим сайленсинга и регуляторные петли
- 6 Оборот
- 7 Клеточные функции
- 8 Эволюция
- 9 Экспериментальное обнаружение и манипуляции
- 10 Болезнь
- 10.1 Наследственные заболевания
- 10.2 Рак
- 10.3 Восстановление ДНК и рак
- 10.4 Болезнь сердца
- 10.4.1 miRNA-712
- 10.4.2 Происхождение
- 10,4.3 Механизм
- 10.4.4 Человеческий гомолог микроРНК-205
- 10.5 Заболевание почек
- 10.6 Нервная система
- 10.6.1 Инсульт
- 10.6.2 Алкоголизм
- 10.7 Ожирение
- 10.8 Гемоста sis
- 11 Некодирующие РНК
- 12 Вирусы
- 13 Предсказание цели
- 14 См. также
- 15 Ссылки
- 16 Дополнительная литература
- 17 Внешние ссылки
История
Первая миРНК была открыта в начале 1990-х годов. Однако miRNA не были признаны классом биологических регуляторов начала 2000-х годов. Исследования miRNA выявили различные наборы miRNA, экспрессируемые в разных типах клеток и тканях, и множественные роли miRNA в развитии растений и животных и во многих других биологических процессах. Аберрантная экспрессия miRNA вовлечена в болезненные состояния. Терапия на основе miRNA находится в стадии исследования.
Первая miRNA была открыта в 1993 году группой под руководством Амброса, в которую входили Ли и Фейнбаум. Однако для понимания его образов действий потребовались одновременно опубликованные работы команды Рувкуна, включая Вайтмана и Ха. Эти группы опубликовали несколько статей о гене lin-4, который, как известно, контролирует время появления C. elegans развитие личинок путем репрессии гена lin-14. Когда Ли и др. выделили lin-4 miRNA, они представлены, вместо продуцирования мРНК, кодирующей белок, она вырабатывала короткие некодирующие РНК, одна из которых была ~ 22-нуклеотидной РНК, которая содержала последовательность, частично комплементарные последовательности в 3 ‘УТР мРНК лин-14. Эта комплементарность была предложена для ингибирования трансляции мРНК lin-14 в белок LIN-14. В то время считалось, что малая РНК lin-4 является идиосинкразией нематод.
В 2000 году была охарактеризована вторая малая РНК: let-7 РНК, которая репрессирует lin-41, чтобы более позднему переходу в развитии у C. elegans. Было обнаружено, что РНК let-7 консервативна у многих видов, что привело к предположению, что РНК let-7 и дополнительные «малые временные РНК» могут регулировать время развития у животных, включая человека.
Год спустя было обнаружено, что lin-4 и let-7 РНК являются частью большого класса РНК, присутствующих в клетках C. elegans, Drosophila и человека. Многие РНК этого класса напоминают РНК lin-4 и let-7, за исключением того, что паттерны их экспрессии обычно несовместимы с ролью в регуляции времени развития. Это предполагает, что большинство из них может функционировать в других типах регуляторных путей. На этом этапе исследователи начали использовать термин «микроРНК» для обозначения этого класса малых регуляторных РНК.
Первым заболеванием человека, нарушением регуляции миРНК, было хронический лимфолейкоз.
Номенклатура
В соответствии со стандартной системой номенклатуры имена присваиваются экспериментально подтвержденным miRNA перед публикацией. За префиксом miR следует тире и число, последнее часто указывает порядок именования. Например, miR-124 был назван и, вероятно, обнаружен до miR-456. «MiR-» с заглавной буквы относится к зрелой форме miRNA, а «mir-» без заглавной буквы относится к pre-miRNA и pri-miRNA. Гены, кодирующие миРНК, также называются с использованием того же трехбуквенного префикса в соответствии с соглашениями номенклатуры генов организма. Например, официальные названия генов miRNA у некоторых организмов — «mir-1 у C. elegans и Drosophila, Mir-1 у Rattus norvegicus и MIR-25 у человека».
миРНК с почти идентичными последовательностями, за исключением одного или двух нуклеотидов, помечаются дополнительной строчной буквой. Например, miR-124a связывает с miR-124b. Например:
- hsa-miR-181a: aacauucaACgcugucggugAgu
- hsa-miR-181b: aacauucaUUgcugucggugGgu
-miRNA, pri-miRNA и гены, которые вызывают к 100% идентичные зрелые миРНК, но расположенные в разных местах генома, обозначены дополнительным суффиксом в виде тире. Например, пре-миРНК hsa-mir-194-1 и hsa-mir-194-2 ведут к идентичной зрелой миРНК (hsa-miR-194), но происходят из генов, расположенных в разных областях генома.
Виды происхождения трехбуквенного префикса, например, hsa-miR-124 — это миРНК человека (Homo sapiens), а oar-miR-124 — миРНК овцы (Ovis aries). Другие общие префиксы включают «v» для вируса (miRNA, кодируемая вирусным геномом) и «d» для miRNA дрозофилы (плодовая муха, обычно изучаемая в генетических исследованиях).
Когда две зрелые микроРНК становятся из противоположных ветвей одной и той же пре-миРНК и обнаруживаются примерно в одинаковом количестве, они обозначаются суффиксом -3p или -5p. (В прошлом различие также проводилось с помощью «s» (смысл ) и «as» (антисмысловой)). Однако зрелых микроРНК, обнаруженных в одном плече шпильки, обычно гораздо больше, чем в другом плече после названия указывает на зрелые виды, обнаруженные на низких уровнях в противоположном плече шпильки. Например, miR-124 и miR-124 * имеют общую шпильку пре-miRNA, но в клетке обнаруживается гораздо больше miR-124.
Мишени
Растительные миРНК обычно имеют почти идеальное спаривание со своими мРНК-мишенями, что вызывает репрессию генов через расщепление транскриптов-мишеней. Напротив, miRNA животных распознавать свои мРНК-мишени, используя всего лишь 6-8 нуклеотидов (область затравки) на 5′-конце miRNA, что недостаточно для спаривания, чтобы вызвать расщепление мРНК-мишени. Комбинаторная регуляция — это особенность регуляции miRNA у животных. Данная miRNA может иметь различных мРНК-мишеней, и альтернативная мишень может регулироваться множеством miRNA.
Оценки среднего числа уникальных матричных РНК, являются мишенями для репрессии типичной miRNA, варьируются в зависимости от методов оценки, но несколько подходов показывают, что miRNA млекопитающих имеют множество уникальных мишеней. Напр., Анализ miRNA, высококонсервативных у позвоночных, показывает, что каждая имеет в среднем примерно 400 консервативных мишеней. Сходным образом эксперименты показывают, что один вид miRNA может снижать стабильность сотен уникальных информационных РНК. Другие эксперименты показывают, что один вид miRNA может подавлять продукцию сотен белков, но эта репрессия бывает относительно (намного меньше, чем в 2 раза). Первым заболеванием человека, нарушением регуляции miRNA, был хронический лимфоцитарный лейкоз. За этим последовали и другие В-клеточные злокачественные новообразования.
Биогенез
До 40% генов миРНК могут находиться в интронах или даже экзонах других генов. Они обычно, не только, находятся в смысловой ориентации и, таким образом, обычно регулируются вместе с их генами-хозяевами.
ДНК-матрица — не последнее слово в производстве зрелой миРНК: 6% человеческих миРНК показывают РНК (IsomiRs ), сайт-специфическая модификация последовательностей РНК для получения продуктов, отличных от продуктов, кодируемых их ДНК. Это увеличивает разнообразие и объем действия miRNA, выходящий за рамки того, что подразумевается только геномом.
Транскрипция
гены миРНК обычно транскрибируются РНК-полимеразой II (Pol II). Полимераза часто связывается с промотором, обнаруженным рядом с последовательностью ДНК, кодируя то, что станет петлей шпильки пре-миРНК. Результирующий транскрипт кэпирован специально модифицированным нуклеотидом на 5′-конце, полиаденилирован множеством аденозинов (поли (A) хвост) и сращивание. МикроРНК животных используется транскрибируется как часть одного плеча РНК из 80 нуклеотидов стебель-петля, которая, в свою очередь, образует часть предшественника миРНК длиной в несколько сотен нуклеотидов, называемую первичной миРНК (pri-miRNA). Когда предшественник стебель-петля обнаруживается в 3 ‘UTR, транскрипт может служить в качестве при-миРНК и мРНК. РНК-полимераза III (Pol III) транскрибирует некоторые миРНК, особенно те, которые расположены выше Последовательности Alu, переносящие РНК (тРНК) и промоторные единицы с широким вкраплением повторов (MWIR) млекопитающих.
Ядерный процессинг

Одна при-миРНК может содержать от одного до шести предшественников миРНК. Эти структуры петли шпильки состоят примерно из 70 нуклеотидов каждая. Каждая шпилька окружена последовательными, необходимыми для эффективной обработки.
Структура двухцепочечной РНК (дцРНК) шпилек в при-миРНК распознается ядерным белком, известным как критическая область 8 синдрома ДиДжорджи (DGCR8 или «Паша» у беспозвоночных), названный в честь его связи с синдромом ДиДжорджи. DGCR8 связывается с ферментом Дроша, белком, который разрезает РНК, с образованием микропроцессорного комплекса. В этом комплексе DGCR8 ориентирует каталитический домен РНКазы III Drosha для освобождения шпилек от pri-miRNA путем расщепления РНК примерно на одиннадцать нуклеотидов от основания шпильки (одна спиральная дцРНК превращается в стержень). Полученный продукт имеет двухнуклеотидный выступ на 3′-конце; он имеет 3 ‘гидроксильные и 5’ фосфатные группы. Ее часто называют пре-миРНК (миРНК-предшественница). Были идентифицированы мотивы ниже пре-миРНК, которые важны для эффективного процессинга.
Пре-миРНК, которые сплайсируются непосредственно из интронов, минуя микропроцессорный комплекс, известны как «Миртроны. «Первоначально считалось, что миртроны существуют только у Drosophila и C. elegans, сейчас миртроны обнаружены у млекопитающих.
До 16% пре-миРНК могут быть посредством ядра. как аденозиндезаминазы, действующие на РНК (ADAR), катализируют переходы аденозина в инозин (от A к I). Редактирование РНК может останавливать ядерный процессинг (например, pri -miR-142, что приводит к деградации процессов рибонуклеазой Tudor-SN) и испытания последующие, включая процессинг цитоплазматической miRNA и специфичность мишени (например, путем изменения зародышевой области miR-376 в центральной нервной системе).
Ядерный экспорт

шпильки пре-миРНК экспортируются из ядра в процессе с участием ядерно-цитоплазматического челнока Экспортин-5. Этот белок, член семейства кариоферинов, распознает двухнуклеотидный выступ, оставленный ферментом РНКазы III Drosha на 3′-конце шпильки пре-миРНК. Опосредованный экспортином-5 транспорт в цитоплазму зависит от энергии с использованием GTP, связанного с Ran белком.
Цитоплазматический процессинг
цитоплазма, шпилька пре-миРНК расщепляется ферментом РНКазы III Дайсер. Эта эндорибонуклеаза взаимодействует с 5 ‘и 3’ концами шпильки и отрезает петлю, соединяющую 3 ‘и 5’ ветви, давая несовершенный дуплекс miRNA: miRNA * длиной около 22 нуклеотидов. Общая длина шпильки и размер петли влияние эффективности обработки дайсером. Несовершенная природа спаривания miRNA: miRNA * также влияет на расщепление. Некоторые из G-богатых пре-миРНК могут принимать структуру G-квадруплекс в качестве альтернативы канонической структуры стержень-петля. Например, человеческая пре-миРНК 92b принимает форму G-квадруплекса, которая устойчива к опосредованному Дайсером расщеплению в цитоплазме. Хотя одна цепь может действовать как функциональная миРНК, только одна цепь обычно включается в РНК-индуцированный комплекс молчания (RISC), где взаимодействуют миРНК и ее мРНК-мишень.
Хотя большинство миРНК расположены в клетках, миРНК, обычно известны некоторые циркулирующие миРНК или внеклеточные миРНК, также были обнаружены во внеклеточной среде, включая различные биологические жидкости и среду для культивирования клеток.
Биогенез в растениях
Биогенез миРНК в растениях отличается от биогена у животных, главным образом, стадиями ядерной обработки и экспорта. Вместо того, чтобы расщепляться двумя разными ферментами, один раз внутри и один вне ядра, оба расщепления миРНК растут гомологом Дайсера, называемым Дайсер-подобный1 (DL1). DL1 экспрессируется только в ядре растительных клеток. Перед тем как дуплексы miRNA: miRNA * транспортируются из ядра, его 3′-выступы метилируются РНК-метилтрансферазным протеином, называемым Hua-Enhancer1 (HEN1). Затем дуплекс переносится из ядра в цитоплазму с помощью белка, называемого Hasty (HST), гомолога Exportin 5, где они разбираются, и зрелая миРНК включается в RISC.
РНК-индуцированный комплекс сайленсинга
Зрелая миРНК является частью активного комплекса индуцированного РНК сайленсинга (RISC), содержащего Дайсер и многие связанные белки. RISC также известен как комплекс рибонуклеопротеидов микроРНК (miRNP); RISC со встроенной miRNA иногда называют miRISC.
Считается, что процессинг пре-миРНК дайсером связан с раскручиванием дуплекса. Как правило, в miRISC включается только одна цепь, выбранная на основании ее термодинамической нестабильности и более слабого спаривания оснований на 5′-конце по сравнению с другой цепью. Положение стержня-петли также может влиять на выбор пряди. Другая, называемая пассажирской цепью из-за ее низких уровней в устойчивом состоянии, обозначена звездочкой (*) и обычно деградирует. В некоторых случаях обе цепи дуплекса жизнеспособны и функциональной miRNA, нацеленной на разные популяции мРНК.
Члены белков белков Аргонавты (назад) являются центральными функциями RISC. Аргонавты необходимы для индуцированного miRNA сайленсинга и содержат два консервативных домена связывания РНК: домен PAZ, который может связывать одноцепочечный 3′-конец зрелой miRNA, и домен PIWI, который структурно напоминает рибонуклеазу-H и взаимодействует с 5-дюймовым концом направляющей нити. Они связывают зрелую миРНК и ориентируют ее для взаимодействия с целью мРНК. Некоторые аргонавты, например Ago2 человека, непосредственно расщепляют транскрипты-мишени; аргонавты также привлекательные дополнительные белки для достижения репрессии трансляции. Геном человека кодирует восемь белков аргонаута, разделенных по сходству последовательностей на два семейства: AGO (четыре члена во всех клетках млекопитающих и называются E1F2C / hAgo у людей) и PIWI (обнаружены в зародышевой линии и гематопоэтических стволовых клетках).
Дополнительные компоненты RISC включают TRBP [белок, связывающий РНК (TAR), трансактивирующую ответную реакцию вируса иммунодефицита человека (ВИЧ)], PACT (белковый активатор интерферона -индуцированного протеинкиназа ), комплекс SMN, хрупкий белок X умственной отсталости (FMRP), белок, содержащий домен стафилококковой нуклеазы Tudor (Tudor-SN), предполагаемая ДНК геликаза MOV10 и мотив распознавания РНК, содержащий белок TNRC6B.
. Режим сайленсинга и регуляторные петли
Молчание генов может происходить либо через деградацию мРНК, либо через предотвращение трансляции мРНК. Например, miR16 содержит последовательность, комплементарную AU-богатому элементу, обнаруженному в 3’UTR многих нестабильных мРНК, таких как TNF альфа или GM-CSF. Было продемонстрировано, что при полной комплементарности между miRNA и последовательностью мРНК-мишени Ago2 может расщеплять мРНК и приводить к прямой деградации мРНК. В отсутствие комплементарности молчание достигается за счет предотвращения трансляции. Связь miRNA и ее мРНК-мишени может быть основана на простой отрицательной регуляции мРНК-мишени, но кажется, что наиболее распространенным сценарием является использование «когерентной прямой петли», «контур взаимной отрицательной обратной связи» (также называемый двойным отрицательным контуром) и «контур положительной обратной связи / прямой связи». Некоторые miRNA работают как буферы случайных изменений экспрессии генов, возникающих из-за случайных событий в транскрипции, трансляции и стабильности белка. Такая регуляция обычно достигается за счет петель отрицательной обратной связи или некогерентной петли прямой связи, разобщающей выход белка с транскрипцией мРНК.
Оборот
Оборот зрелой miRNA необходим для быстрых изменений в профилях экспрессии miRNA. Считается, что во время созревания miRNA в цитоплазме поглощение белком Argonaute стабилизирует направляющую цепь, тогда как противоположная (* или «пассажирская») цепь предпочтительно разрушается. В так называемой стратегии «Используй или потеряй», Argonaute может предпочтительно сохранять miRNA с большим количеством мишеней, чем miRNA с небольшим количеством мишеней или без них, что приводит к деградации не нацеливающих молекул.
Распад зрелых miРНК в Caenorhabditis elegans опосредована 5′-3′-эксорибонуклеазой XRN2, также известной как Rat1p. У растений члены семейства SDN (малые РНК-деградирующие нуклеазы) разрушают miRNAs в противоположном (3′-к-5 ‘) направлении. Подобные ферменты кодируются в геномах животных, но их роль не описана.
Некоторые модификации miRNA влияют на стабильность miRNA. Как показала работа с модельным организмом Arabidopsis thaliana (кресс-салатом), миРНК зрелых растений, по-видимому, стабилизировались добавлением метильных групп на 3′-конце. 2′-O-конъюгированные метильные группы блокируют добавление остатков урацила (U) ферментами уридилтрансфераза, модификация, которая может быть связана с деградацией miRNA. Однако уридилирование может также защищать некоторые miRNA; последствия этой модификации до конца не изучены. Сообщалось об уридилировании некоторых микроРНК животных. Как растения, так и животные могут использоваться путем добавления остатков аденина (A) к 3′-концу миРНК. Дополнительный A, добавленный к концу miR-122 <>млекопитающих, обогащенной печени miRNA, при гепатите C, стабилизирует молекулу, а миРНК растений, заканчивающиеся остатком аденина, имеют более медленную скорость распада.
Функции клетки

Функция miRNA, по-видимому, заключается в генах регулировании. Для этой цели миРНК комплементарна части одной или нескольких информационной РНК (мРНК). MiRNA животных обычно комплементарны сайту в 3 ‘UTR, тогда как miRNA растений обычно комплементарны кодирующим областям мРНК. Идеальное или почти идеальное спаривание оснований с целью РНК достижения расщепления РНК. Это основной вид миРНК растений. У животных совпадения несовершенные.
Чтобы частично комплементарные микроРНК распознавали свои мишени, нуклеотиды 2–7 миРНК (ее «затравочная область») должны быть совершенно комплементарными. МикроРНК животных ингибируют трансляцию белка мРНК-мишени (она присутствует, но менее распространена у растений). Частично комплементарные микроРНК также могут ускорять деаденилирование, что приводит к более ранней деградации мРНК. Хотя деградация мРНК, нацеленная на miRNA, хорошо задокументирована, происходит ли репрессия трансляции за счет деградации мРНК, ингибирования трансляции или их комбинации, горячо обсуждается. Недавняя работа по miR-430 у рыбок данио, а также по бантам-miRNA и miR-9 в культивируемых клетках дрозофилы, показывает, что репрессия трансляции вызывается нарушением инициации трансляции, независимо от деаденилирования МРНК.
miРНК иногда также вызывают модификацию гистонов и метилирование ДНК сайтов промотора, что влияет на экспрессия генов-мишеней.
Девять механизмов действия miRNA модели и собраны в единую математическую модель:
- ингибирование инициации Cap-40S;
- 60S ингибирование присоединения рибосомных единиц;
- ингибирование элонгации;
- выпадение рибосом (преждевременная терминация);
- ко-трансляционная деградация растущего белка;
- секвестрация в Р-тельцах;
- распад (дестабилизация) мРНК;
- расщепление мРНК;
- ингибирование транскрипции посредством реорганизации хроматина, опосредованной микроРНК, с последующим подавлением гена.
Эти механизмы различить u Приведем часто экспериментальные данные о стационарных скоростях реакции. Тем не менее, они различаются по динамике и имеют разные кинетические сигнатуры.
В отличие от растительных микроРНК, животные микроРНК нацелены на различные гены. Гены, участвующие в функциях, общих для всех клеток, таких как экспрессия генов, имеют меньше участков-мишеней микроРНК и, по-видимому, находятся в процессе отбора, чтобы избежать нацеливания микроРНК.
дцРНК также может активировать экспрессию гена, механизм, который был назван «активацией гена, индуцированной малой РНК» или РНКа. дцРНК, нацеленные на промоторы генов, могут индуцировать мощную активацию транскрипции ассоциированных генов. Это было применимо на клетках человека с использованием синтетических дцРНК, называемых малыми активирующими РНК (саРНК ), но также было для эндогенных микроРНК.
Взаимодействия между микроРНК и комплементарными последовательностями генов и даже псевдогены, которые разделяют гомологию последовательностей, считают обратным каналом связи, регулирующими уровнями экспрессии между паралогическими генами. Получив название «конкурирующие эндогенные РНК» (ceRNAs ), эти микроРНК связываются с «элементами микроРНК» на генах и псевдогенах и могут служить еще одним объяснением устойчивости некодирующей ДНК.
Эволюция
миРНК хорошо консервативны как у растений, так и у животных и считаются жизненно важным и древним компонентом регуляции генов. В то время как основные компоненты пути микроРНК сохраняются между растениями и животными, репертуары миРНК в двух царствах, по-видимому, возникли независимо с разными способами действия.
микроРНК являются полезными филогенетическими маркерами из-за их явно низкой скорости эволюции. Происхождение микроРНК как регуляторный механизм развился из предыдущего механизма РНКи, который использовался в качестве от экзогенного генетического материала такого как вирусы. Их происхождение, возможно, способствовало развитию морфологических инноваций, сделав экспрессию генов более специфичной и «настраиваемой», их генезису сложных органов и, возможно, в конечном итоге, сложной жизни. Быстрые всплески морфологических инноваций обычно связаны с высокой скоростью накопления микроРНК.
Новые микроРНК заблокированных территорий. Новые микроРНК могут возникнуть в результате случайного образования шпилек в «некодирующих» участках ДНК (то есть в интронах или межгенных областях), но также в результате дублирования и модификации микроРНК. микроРНК могут также образовываться из перевернутых дупликаций последовательностей, кодирующих белок, что позволяет создавать складную структуру шпильки. Скорость эволюции (т. Е. Нуклеотидного за ущерб) в недавно возникших микроРНК сравнима с таковой в других частях некодирующей ДНК, что подразумевает эволюцию за счет нейтрального дрейфа; однако более старые микроРНК имеют гораздо более низкую скорость изменения (часто менее одной замены за сто миллионов лет), предполагаемая, как только микроРНК приобретает функцию, она подвергается очищающей селекции. Отдельные регионы в пределах гена miRNA сталкиваются с разными эволюционными процессами давления, где регионы, которые жизненно важны для процесса обеспечения и функционирования, более высокие уровни сохранения. На этом этапе микроРНК редко теряется из генома животного, хотя более новые микроРНК (таким образом, предположительно нефункциональные) часто теряются. В Arabidopsis thaliana чистый поток генов miRNA, как было предсказано, составляет от 1,2 до 3,3 генов на миллион лет. Это делает ценным филогенетическим маркером, и исследует как возможное решение выдающихсяогенетических проблем, таких как их взаимоотношения членистоногих. С другой стороны, во многих случаях микроРНК плохо коррелируют с филогенезом, и возможно, их филогенетическое соответствие в степени соответствует ограниченный выбор микроРНК.
микроРНК присутствуют в геномах эукариот. организмов, от бурых водорослей до животных. Предполагается, что микроРНК независимо от растений и животных.
Если сосредоточить внимание на животных, геном Mnemiopsis leidyi, по-видимому, отсутствует узнаваемые микроРНК, а также ядерные белки Дроша и Паша, которые имеют решающее значение для биогенеза канонических микроРНК. На данный момент это единственное животное, пропавшее без вести Дроша. МикроРНК играет жизненно важную роль в регуляции экспрессии генов у всех исследованных животных без гребневиков, за исключением Trichoplax adhaerens, единственного известного представителя марты филума Placozoa.
У всех видов в кобеду 2010 г. было идентифицировано более 5000 различных микроРНК. В то время как короткие используются РНК с широко сопоставимой функцией у бактерий, у бактерий отсутствуют настоящие микроРНК.
Экспериментальное обнаружение и манипуляции
В то время как исследователи сосредоточили внимание на экспрессии miRNA в физиологических и патологических процессах, возникли различные технические переменные, связанные с выделением микроРНК. Стабильность хранимых образцов miRNA была поставлена под сомнение. микроРНК разлагаются легче, чем мРНК, из-за их длины, но также из-за повсеместного присутствия РНКаз. Это требует образцов на льду и использования оборудования, свободного от РНКазы.
экспрессию микроРНК можно количественно измерить в двухэтапном процессе полимеразной цепной реакции модифицированного ОТ-ПЦР с продуктом количественной ПЦР. Варианты метода позволяют получить абсолютную или относительную количественную оценку. miRNA также можно гибридизировать с микрочипами, слайдами или чипами с зондами для сотен или мишеней miRNA, так что относительные уровни miRNA могут быть эффективными в различных образцах. микроРНК могут быть обнаружены и профилированы методы высокопроизводительного секвенирования (секвенирование микроРНК ). Активность миР можно экспериментально ингибировать с помощью заблокированной нуклеиновой кислоты (LNA) олиго, морфолино олиго или 2′-O-метил-олиго РНК.. Конкретная miRNA может подавляться комплементарным антагомиром. Созревание микроРНК может ингибироваться в нескольких точках стерически блокирующими олигонуклеотидами. Сайт-мишень miRNA транскрипта мРНК также может быть заблокирован стерически-блокирующим олиго. Для «in situ» miRNA установка «зонды» LNA или Morpholino. Закрытая конформация LNA приводит к увеличению чувствительности и селективности, что делает ее идеальной для обнаружения коротких miRNA.
Высокопроизводительное количественное определение miRNA подвержено ошибкам из-за большей дисперсии (по сравнению с мРНК ), что связано с методологическими проблемами. мРНК -экспрессия поэтому часто анализируется, чтобы проверить эффекты miRNA на их уровнях (например, in). Базы данных Программу для установки мРНК и данные miRNA, которые предсказывают мишени miRNA на основе их применяют. Хотя это обычно делается после обнаружения представляющих интерес miRNA (например, из-за высокого уровня экспрессии), были предложены идеи инструментов анализа, которые объединяют мРНК и информацию об экспрессии miRNA.
Заболевание
Подобно тому, как миРНК участвует в нормальном функционировании эукариотических клеток, нарушение регуляции миРНК также связано с заболеванием. Созданная вручную общедоступная база данных miR2Disease документирует известную взаимосвязь между нарушением регуляции miRNA и заболеваниями человека.
Унаследованные заболевания
Мутация в области зародыша miR-96 вызывает наследственную прогрессирующую потерю слуха.
Мутация в семенной области miR-184 вызывает наследственный кератоконус с передней полярной катарактой.
Удаление кластера miR-17 ~ 92 вызывает дефекты скелета и роста.
Рак
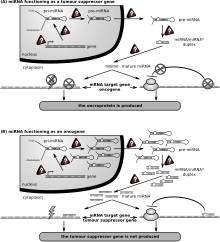
Первым заболеванием человека, которое, как известно, связано с нарушением регуляции miRNA, был хронический лимфолейкоз. Многие другие миРНК также связаны с раком и, соответственно, иногда называются «онкомиры ». В злокачественных В-клетках миРНК участвуют в путях, фундаментальных для развития В-клеток, таких как передача сигналов В-клеточного рецептора (BCR), миграция / адгезия В-клеток, межклеточные взаимодействия в иммунных нишах, а также производство и переключение классов иммуноглобулинов. MiRNA влияют на созревание В-клеток, образование пре-, маргинальной зоны, фолликулярных, В1, плазменных В-клеток и В-клеток памяти.
Другая роль miRNA при раке — использовать уровень их экспрессии для прогноза. В образцах NSCLC низкие уровни miR-324 a могут служить индикатором плохой выживаемости. Высокие уровни miR-185 или низкие уровни miR-133b могут коррелировать с метастазами и плохой выживаемостью при колоректальном раке.
Более того, специфические miRNA могут быть связаны с некоторыми гистологическими подтипами колоректального рака. Например, было показано, что уровни экспрессии miR-205 и miR-373 повышены при муцинозном колоректальном раке и связанном с муцином язвенном колите раке толстой кишки, но не при спорадической аденокарциноме толстой кишки, в которой отсутствуют муцинозные компоненты. Исследования in vitro показали, что miR-205 и miR-373 могут функционально вызывать различные признаки опухолевой прогрессии, связанной с муцином, в эпителиальных клетках кишечника.
Пролиферация клеток гепатоцеллюлярной карциномы может возникать в результате взаимодействия miR-21 с MAP2K3, a ген-репрессор опухоли. Оптимальное лечение рака включает точное определение пациентов для терапии со стратификацией риска. Те, у кого есть быстрый ответ на начальное лечение, могут получить пользу от сокращенных схем лечения, что свидетельствует о ценности точного ответа на заболевание m меры. Бесклеточные миРНК очень стабильны в крови, сверхэкспрессируются при раке и поддаются количественной оценке в диагностической лаборатории. При классической лимфоме Ходжкина плазменные miR-21, miR-494 и miR-1973 являются многообещающими биомаркерами на заболевание. Циркулирующие миРНК посредством принятия клинических решений и интерпретации результатов позитронно-эмиссионной томографии в сочетании с компьютерной томографией. Их можно проводить на каждой консультации для реакции на заболевание и имеет рецидива.
МикроРНК могут быть использованы в качестве инструментов или мишеней для лечения различных видов рака. В нескольких исследованиях было обнаружено, что специфическая микроРНК, miR-506, действует как антагонист опухоли. Было обнаружено, что в значительном количестве образцов рака шейки матки miR-506 подавлена. Кроме того, miR-506 способствует апоптозу клеток рака шейки матки через фактор транскрипции прямого пути ежа, Gli3.
Восстановление ДНК и рак
Рак вызывается накоплением мутации из-за повреждений ДНК или неисправных ошибок в репликации ДНК. Дефекты репарации ДНК вызывают накопление мутаций, которые могут привести к раку. Некоторые гены, участвующие в репарации ДНК, регулируются микроРНК.
Мутации зародышевой линии в генах репарации ДНК вызывают только 2–5% случаев рака толстой кишки. Однако измененная микроРНК, вызывающая дефицит репарации ДНК, часто используется причинным фактором. Среди 68 спорадических случаев рака толстой экспрессией белка репарации быстрого спаривания ДНК MLH1 в большинстве случаев обнаружено дефицит из-за эпигенетического метилирования CpG остров гена MLH1. Однако до 15% дефицита MLH1 при спорадических формах рака толстой кишки, по-видимому, сотрудничает со сверхэкспрессией микроРНК miR-155, которая подавляет экспрессию MLH1.
В 29–66% глиобластом репарация ДНК недостаточна из-за эпигенетического метилирования гена MGMT, которое снижает экспрессию белка MGMT. Однако для 28% глиобластом белка MGMT недостаточен, но промотор MGMT не метилирован. В глиобластомах без метилированных промоторов MGMT уровень микроРНК miR-181d обратно коррелирует с экспрессией белка MGMT, а прямой мишенью miR-181d является MGMT мРНК 3’UTR (трехпрайм нетранслируемых области мРНК МГМТ). Таким образом, в 28% глиобластом повышенная экспрессия miR-181d и пониженная экспрессия фермента репарации репарации ДНК MGMT могут быть причинными факторами ДНК.
Белки HMGA (HMGA1a, HMGA1b и HMGA2) участвуют в развитии рака, и экспрессия этих белков регулируется микроРНК. Экспрессия HMGA почти не обнаруживается в дифференцированных тканях взрослого человека, но повышена во многих случаях рака. Белки HMGA представляют собой полипептиды из ~ 100 аминокислотных остатков, характеризующиеся модульной организацией последовательностей. Эти белки имеют три высоко положительно заряженных участка, называемых АТ-крючками, которые связывают малую бороздку участков ДНК, богатых АТ, в диаграмме ДНК. Новообразования человека, включая карциномы щитовидной железы, предстательной железы, шейки матки, колоректального тракта, поджелудочной железы и яичников, демонстрируют сильное увеличение белков HMGA1a и HMGA1b. У трансгенных мышей с HMGA1, нацеленным на лимфоидные клетки, агрессивная лимфома, показывает, что высокая экспрессия HMGA1 связана с раком и что HMGA1 может действовать как онкоген. Белок HMGA2 специфически нацелен на промотор ERCC1, тем самым снижая экспресс этого гена репарации ДНК. Экспрессия белка ERCC1 была недостаточной в 100% из 47 оцененных случаев рака толстой кишки (степень участия HGMA2 неизвестна).
Болезнь сердца
Глобальная роль функции miRNA в сердце была устранена путем условного ингибирования созревания миРНК в сердце мыши. Это показало, что miRNA играет роль во время его развития. Исследования профилей экспрессии miRNA демонстрируют, что уровни miRNA проявляются в пораженном сердце человека, экспрессия их участие в кардиомиопатиях. Более специфические miRNA на животных идентифицировали роли miRNA как во время развития сердца, включая регуляцию ключевых факторов, важных для кардиогенеза, реакции гипертрофического роста и сердечной проводимости. Другая роль миРНК в сердечно-сосудистых заболеваниях — использование уровней их экспрессии для диагностики, прогноза или стратификации риска. miRNA в моделях животных также связаны с метаболизмом и регуляцией холестерина.
miRNA-712
Мышиная микроРНК-712 является потенциальным биомаркером (т.е. предиктором) атеросклероза, сердечно-сосудистые заболевания, артериальной стенки, связанного с задержкой липидов и воспалением. Неламинарный кровоток также коррелирует с развитием атеросклероза, поскольку механосеноры эндотелиальных клеток реагируют на силу сдвига нарушенного кровотока (d-поток). Ряд проатерогенных генов, включая матриксные металлопротеиназы (MMP), активируются с помощью d-потока, опосредуя провоспалительные и проангиогенные сигналы. Эти результаты наблюдались в перевязанных сонных артериях мышей, чтобы имитировать эффекты d-потока. В течение 24 часов ранее существовавшая незрелая miR-712 образовывала зрелую miR-712, что позволяет предположить, что miR-712 является чувствительной к потоку. Соответствуя этим результатам, miR-712 также активируется в эндотелиальных клетках, подвергающихся естественному d-потоку в большей кривизне дуги аорты.
Источник
Последовательность пре-мРНК miR-712 генерируется из гена рибосомных RN45 мыши в области 2 внутреннего транскрибированного спейсера (ITS2). XRN1 представляет собой экзонуклеазу, которая разрушает область ITS2 во время процессинга RN45. Таким образом, уменьшение XRN1 в условиях d-потока приводит к накоплению miR-712.
Механизм
MiR-712 нацелен на тканевой ингибитор металлопротеиназы 3 (TIMP3). ТИМП обычно регулируют активность матриксных металлопротеиназ (ММП), которые разрушают внеклеточный матрикс (ЕСМ). Артериальный ECM в основном состоит из коллагена и эластина, обеспечивающих структурную поддержку и отдачи артерий. Эти волокна играют решающую роль в регуляции воспаления и проницаемости сосудов, которые важны для развития атеросклероза. TIMP3, экспрессируемый эндотелиальными клетками, является единственным TIMP, являющимся с ECM. Снижение экспрессии TIMP3 приводит к увеличению деградации ECM в присутствии d-потока. В соответствии с данным, ингибирование до miR712 увеличивает экспрессию TIMP3 в клетках при воздействии турбулентного потока.
TIMP3 также снижает TNFα (провоспалительного регулятора) во время турбулентного потока. Активность TNFα в турбулентном потоке измеряют по экспрессии TNFα-превращающего фермента (TACE) в крови. TNFα снижается, если ингибируется miR-712 или TIMP3 сверхэкспрессируется, что позволяет предположить, что miR-712 и TIMP3 регулируют активность TACE в условиях турбулентного потока.
Анти-miR-712 эффективно подавляет экспрессию miR-712, индуцированную d-потоком, усиливает экспрессию TIMP3. Анти-miR-712 также ингибирует повышенную проницаемость сосудов, тем самым значительно снижая развитие атеросклероза и инфильтрации иммунных клеток.
Гомолог микроРНК-205 человека
Гомолог miR-712 человека был обнаружен на RN45. ген-гомолог, который поддерживает микроРНК, аналогичные мышам. МиР-205 человека имеет сходные последовательность с miR-712 мышей и охраняем у позвоночных. MiR-205 и miR-712 также разделяют более 50% клеточных сигнальных мишеней, включая TIMP3.
При тестировании d-поток снижал экспрессию XRN1 у людей, как это было в эндотелиальных клетках мышей, что указывает на распространенную роль XRN1 у людей.
Заболевание почек
Нацеленная делеция Dicer в FoxD1 -производственных почечных клетках-предшественников на мышиной модели привела к сложному почечному фенотипу, включая увеличение предшественников нефрона, меньшее количество клеток ренина, гладкомышечные артериолы, прогрессирующую мезангиальную потерю и клубочковые аневризмы. Высокопроизводительное профилирование полного транскриптома модели мыши с нокаутом FoxD1-Dicer выявило эктопическую активацию проапоптотического гена Bcl2L11 (Bim) и нарушение регуляции пути p53 с созданием эффекторных генов p53, включая Bax, Trp53inp1, Jun, Cdkn1a, Mmp2 и Arid3a. Уровни белка p53 остались неизменными, что позволяет предположить, что стромальные miRNA FoxD1 непосредственно репрессируют гены-эффекторы p53. Используя метод метода клонов с последующим сортировкой флуоресцентно-активируемых клеток, профилирование миРНК-клеток, полученных из FoxD1, не только всесторонне определенно транскрипционный ландшафт микроРНК, которые имеют решающее значение для развития сосудов, но также идентифицированы ключевые микроРНК, которые являются вероятно, модулирующий почечный фенотип в его отсутствие. Эти miRNA включают miRs-10a, 18a, 19b, 24, 30c, 92a, 106a, 130a, 152, 181a, 214, 222, 302a, 370 и 381, которые регулируют Bcl2L11 (Bim) и miRs-15b, 18a, 21, 30c, 92a, 106a, 125b-5p, 145, 214, 222, 296-5p и 302a, которые регулируют гены эффекторов р53. В соответствии с результатами профилирования, эктопический апоптоз наблюдался в клеточных производных линии предков, происходящих от FoxD1, подтвердился почечных стромальных миРНК в клеточном гомеостазе.
Нервная система
миРНК, по-видимому, регулируют развитие функции и нервной системы. Нервные miRNA участвуют на различных стадиях синаптического развития, включая дендритогенез (включая miR-132, miR-134 и miR-124 ), образование синапса и синапс созревание ( где, как полагают, участвуют miR-134 и miR-138). Некоторые исследования обнаруживают измененную экспрессию miRNA при болезни Альцгеймера, а также шизофрении, биполярном расстройстве, большой депрессии и тревожных расстройствах..
Инсульт
По данным Центра по контролю и профилактике заболеваний, инсульт причин одной из основных смерти и длительной инвалидности в Америке. 87% случаев связаны с ишемическим инсультом, который возникает в результате закупорки артерии головного мозга, по которой проходит богатая кислородом кровь. Обструкция кровотока означает, что мозг не может получать необходимые питательные вещества, такие как кислород и глюкоза, и удалять отходы, такие как углекислый газ. miRNA играют роль в посттрансляционном подавлении генов, воздействуют на гены в патогенезе церебральной ишемии, такие как воспалительный, ангиогенез и апоптотический пути.
Алкоголизм
Жизненно важная роль miRNA в экспрессии генов имеет значение для зависимости, в частности алкоголизма. Хроническое использование алкоголем приводит к стойким изменениям функции мозга, частично опосредованным изменениями экспрессии гена . Глобальная регуляция miRNA многих нижестоящих генов считается значимой в отношении реорганизации или синаптических связей или долгосрочных нейронных адаптаций, включающих изменение поведения от потребленияоля к отмене и / или. В посмертном мозге алкоголика было обнаружено изменение до 35 различных миРНК, все из которых нацелены на гены, которые включают регуляцию клеточного цикла, апоптоза, клеточная адгезия, развитие нервной системы и передача сигналов. Измененные уровни miRNA были обнаружены в медиальной префронтальной коре алкогольной зависимости мышей, что свидетельствует о роли miRNA в управлении трансляционным дисбалансом и создании дифференциально экспрессируемых белков той области мозга, где сложное когнитивное поведение и выработка решений скорее всего происходит.
миРНК могут быть либо активированы, либо подавлены в ответ на хроническое употребление алкоголя. Экспрессия miR-206 увеличивалась в префронтальной коре головного мозга алкоголезависимых крыс, воздействуя на нейротрофический фактор головного мозга (BDNF ) фактор транскрипции и в конечном итоге снижая его экспрессию. BDNF играет решающую роль в формировании и созревании новых нейронов и синапсов, предполагаемое возможное участие в росте синапсов / синаптической пластичности у лиц, злоупотребляющих алкоголем. Было обнаружено, что miR-155, важная для регуляции вызванных алкоголем факторов нейровоспаления, активируется, что свидетельствует о роли микроглии и воспалительные цитокинов в патофизиологии алкоголя. Подавление miR-382 было обнаружено в исходном ядре, структуре в базальном переднем мозге, значимой в регулировании чувства вознаграждения, которое питает мотивационные привычки. miR-382 является мишенью для дофаминового рецептора D1 (DRD1), и его сверхэкспрессия приводит к усилению регуляции DRD1 и дельта fosB, фактор транскрипции, который активирует серию событий транскрипции. в результате accumbens, что в итоге приводит к аддиктивному поведению. С другой стороны, сверхэкспрессия miR-382 приводит к ослаблению употребления алкоголя и ингибированию активации DRD1 и дельта fosB в моделях алкоголизма на крысах, демонстрируя возможность использования miRNA фармацевтических препаратов при лечении.
Ожирение
миРНК играют решающую роль в регуляции предшественников стволовых клеток, дифференцирующихся в адипоциты. Исследования для определения того, какую роль плюрипотентные стволовые клетки играют в адипогенезе, были изучены на иммортализованной линии hMSC костного мозга человека стромальных клеток. -Tert20. Снижение экспрессии miR-155, miR-221 и miR-222 было обнаружено во время адипогенного программирования как иммортализованных, так и первичных hMSC, что позволяет предположить, что они установлены как негативные регуляторы дифференцировки. Напротив, эктопическая экспрессия miРНК 155, 211 и 222 ингибировала адипогенез и подавляла индукцию главных регуляторов PPARγ и CCAAT / связывающего энхансер белка альфа (CEBPA ). Это открывает путь к возможному лечению генетического ожирения.
Другим классом miRNA, которые регулируют инсулинорезистентность, ожирение и диабет, является семейством let-7.. Let-7 накапливается в тканях человека в процессе старения. Когда let-7 эктопически сверхэкспрессировалась, чтобы имитировать ускоренное старение, мыши становились инсулинорезистентными и, таким образом, более склонными к ожирению, вызванному диетой с высоким уровнем жиров, и диабету. Напротив, когда let-7 ингибировалась инъекциями let-7-специфических антагомиров, мыши становятся более чувствительными к инсулину и значительно устойчивы к ожирению и диабету, вызванному диетой с высоким уровнем жиров. Подавление let-7 может не только предотвратить ожирение и диабет, но обратить вспять и вылечить это состояние. Эти экспериментальные данные предполагают, что ингибирование let-7 может представлять собой новую терапию для ожирения и диабета 2 типа.
Гемостаз
миРНК также играют решающую роль в регуляции сложных каскадов, включая систему гемостатического свертывания крови. Крупномасштабные исследования функционального нацеливания miRNA выявили рациональные терапевтические цели в системе гемостаза.
Некодирующие РНК
Когда проект генома человека обнаружил его первые хромосома в 1999 году было предсказано, что геном будет содержать более 100 000 генов, кодирующих белок. Однако в конечном итоге было идентифицировано только около 20 000 человек. С тех пор появление биоинформатических подходов в сочетании с исследованиями разбиения генома, изучающими транскриптом, систематическим секвенированием полноразмерных библиотек кДНК и экспериментальной проверкой (включая создание антисмысловых олигонуклеотидов, полученных из микроРНК, называемых антагомирами ), что многие транскрипты представляют собой небелковые РНК, включая несколько snoRNAs и miRNA.
Вирусы
вирусные микроРНК играют роль в регуляции генов экспрессия вирусных генов и / или генов хозяина на пользу вирусу. Следовательно, miRNA играют ключевую роль во взаимодействии хозяин-вирус и патогенезе вирусных заболеваний. Считается, что экспрессия активаторов транскрипции ДНК вируса герпеса-6 человека регулируется вирусной миРНК.
Предсказание цели
миРНК могут связываться с целевой матричной РНК (мРНК) транскрипты генов, кодирующие белок, отрицательно контролируют их трансляцию или вызывают деградацию мРНК. Ключевое значение имеет конкретное определение мишеней miRNA. Доступно сравнение прогнозирующей производительности восемнадцати алгоритмов in silico. Крупномасштабные исследования функционального таргетинга miRNA показывают, что многие функциональные miRNA могут быть упущены алгоритмами прогнозирования целей.
См. Также
Биологический портал
- Анти-miRNA олигонуклеотиды
- Экспрессия генов
- Список инструментов для прогнозирования гена miRNA
- Список инструментов для прогнозирования мишеней miRNA
- MicroDNA
- miR-324-5p
- РНК-интерференция
- Малая интерферирующая РНК
- Маленькая ядрышковая РНК-производная микроРНК
Ссылки 146>
Дополнительная литература
Внешние ссылки
- База данных miRBase
- miRTarBase, экспериментально подтвержденная база данных взаимодействий микроРНК-мишень.
- семирна, веб-приложение для поиска микроРНК в геноме растений.
- ONCO.IO : Интегрирующий ресурс для анализа микроРНК и факторов транскрипции при раке.
- MirOB : База данных MicroRNA, а также инструменты анализа и обработки данных.
- База данных ChIPBase : База данных с открытым доступом для расшифровка факторов транскрипции, которые участвовали в транскрипции микроРНК или влияли на нее, из данных ChIP-seq.
- Анимированное видео процесса биогенеза микроРНК.
- Реагенты для модуляции миРНК для включения регуляция или подавление функции эндогенных зрелых микроРНК
Pre-miRNA instead of Pri-miRNA in the first point of mechanism. Diagram of microRNA (miRNA) action with mRNA
Examples of miRNA stem-loops, with the mature miRNAs shown in red
MicroRNA (miRNA) are small, single-stranded, non-coding RNA molecules containing 21 to 23 nucleotides.[1] Found in plants, animals and some viruses, miRNAs are involved in RNA silencing and post-transcriptional regulation of gene expression.[2][3] miRNAs base-pair to complementary sequences in mRNA molecules,[4] then gene silence said mRNA molecules by one or more of the following processes: (1) cleavage of mRNA strand into two pieces, (2) destabilization of mRNA by shortening its poly(A) tail, or (3) translation of mRNA into proteins. This last method of gene silencing is the least efficient of the three, and requires the aid of ribosomes.[4][5]
miRNAs resemble the small interfering RNAs (siRNAs) of the RNA interference (RNAi) pathway, except miRNAs derive from regions of RNA transcripts that fold back on themselves to form short hairpins, whereas siRNAs derive from longer regions of double-stranded RNA.[6] The human genome may encode over 1900 miRNAs,[7] although more recent analysis suggests that the number is closer to 2,300.[8] However, only about 500 human microRNAs represent bona fide miRNA in the manually curated miRNA gene database MirGeneDB.[9]
miRNAs are abundant in many mammalian cell types[10][11] and as extracellular circulating miRNAs.[12] Circulating miRNAs are released into body fluids including blood and cerebrospinal fluid and have the potential to be available as biomarkers in a number of diseases.[12][13] MiRNAs appear to target about 60% of the genes of humans and other mammals.[14][15] Many miRNAs are evolutionarily conserved, which implies that they have important biological functions.[16][2] For example, 90 families of miRNAs have been conserved since at least the common ancestor of mammals and fish, and most of these conserved miRNAs have important functions, as shown by studies in which genes for one or more members of a family have been knocked out in mice.[2]
History[edit]
The first miRNA was discovered in the early 1990s.[17] However, miRNAs were not recognized as a distinct class of biological regulators until the early 2000s.[18][19][20][21][22] miRNA research revealed different sets of miRNAs expressed in different cell types and tissues[11][23] and multiple roles for miRNAs in plant and animal development and in many other biological processes.[24][25][26][27][28][29][30] Aberrant miRNA expression are implicated in disease states. MiRNA-based therapies are under investigation.[31][32][33][34]
The first miRNA was discovered in 1993 by a group led by Ambros and including Lee and Feinbaum. However, additional insight into its mode of action required simultaneously published work by Ruvkun’s team, including Wightman and Ha.[17][35] These groups published back-to-back papers on the lin-4 gene, which was known to control the timing of C. elegans larval development by repressing the lin-14 gene. When Lee et al. isolated the lin-4 miRNA, they found that instead of producing an mRNA encoding a protein, it produced short non-coding RNAs, one of which was a ~22-nucleotide RNA that contained sequences partially complementary to multiple sequences in the 3′ UTR of the lin-14 mRNA.[17] This complementarity was proposed to inhibit the translation of the lin-14 mRNA into the LIN-14 protein. At the time, the lin-4 small RNA was thought to be a nematode idiosyncrasy.
In 2000, a second small RNA was characterized: let-7 RNA, which represses lin-41 to promote a later developmental transition in C. elegans.[18] The let-7 RNA was found to be conserved in many species, leading to the suggestion that let-7 RNA and additional «small temporal RNAs» might regulate the timing of development in diverse animals, including humans.[19]
A year later, the lin-4 and let-7 RNAs were found to be part of a large class of small RNAs present in C. elegans, Drosophila and human cells.[20][21][22] The many RNAs of this class resembled the lin-4 and let-7 RNAs, except their expression patterns were usually inconsistent with a role in regulating the timing of development. This suggested that most might function in other types of regulatory pathways. At this point, researchers started using the term «microRNA» to refer to this class of small regulatory RNAs.[20][21][22]
The first human disease associated with deregulation of miRNAs was chronic lymphocytic leukemia. In this disorder, the miRNAs have a dual role working as both tumor suppressors and oncogenes.[36]
Nomenclature[edit]
Under a standard nomenclature system, names are assigned to experimentally confirmed miRNAs before publication.[37][38] The prefix «miR» is followed by a dash and a number, the latter often indicating order of naming. For example, miR-124 was named and likely discovered prior to miR-456. A capitalized «miR-» refers to the mature form of the miRNA, while the uncapitalized «mir-» refers to the pre-miRNA and the pri-miRNA.[39] The miRNAs encoding genes are also named using the same three-letter prefix according to the conventions of the organism gene nomenclature. For examples, the official miRNAs gene names in some organisms are «mir-1 in C. elegans and Drosophila, Mir-1 in Rattus norvegicus and MIR-25 in human.
miRNAs with nearly identical sequences except for one or two nucleotides are annotated with an additional lower case letter. For example, miR-124a is closely related to miR-124b. For example:
- hsa-miR-181a: aacauucaACgcugucggugAgu
- hsa-miR-181b: aacauucaUUgcugucggugGgu
Pre-miRNAs, pri-miRNAs and genes that lead to 100% identical mature miRNAs but that are located at different places in the genome are indicated with an additional dash-number suffix. For example, the pre-miRNAs hsa-mir-194-1 and hsa-mir-194-2 lead to an identical mature miRNA (hsa-miR-194) but are from genes located in different genome regions.
Species of origin is designated with a three-letter prefix, e.g., hsa-miR-124 is a human (Homo sapiens) miRNA and oar-miR-124 is a sheep (Ovis aries) miRNA. Other common prefixes include «v» for viral (miRNA encoded by a viral genome) and «d» for Drosophila miRNA (a fruit fly commonly studied in genetic research).
When two mature microRNAs originate from opposite arms of the same pre-miRNA and are found in roughly similar amounts, they are denoted with a -3p or -5p suffix. (In the past, this distinction was also made with «s» (sense) and «as» (antisense)). However, the mature microRNA found from one arm of the hairpin is usually much more abundant than that found from the other arm,[6] in which case, an asterisk following the name indicates the mature species found at low levels from the opposite arm of a hairpin. For example, miR-124 and miR-124* share a pre-miRNA hairpin, but much more miR-124 is found in the cell.
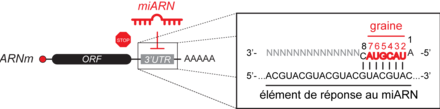
Targets[edit]
Plant miRNAs usually have near-perfect pairing with their mRNA targets, which induces gene repression through cleavage of the target transcripts.[24][40] In contrast, animal miRNAs are able to recognize their target mRNAs by using as few as 6–8 nucleotides (the seed region) at the 5′ end of the miRNA,[14][41][42] which is not enough pairing to induce cleavage of the target mRNAs.[4] Combinatorial regulation is a feature of miRNA regulation in animals.[4][43] A given miRNA may have hundreds of different mRNA targets, and a given target might be regulated by multiple miRNAs.[15][44]
Estimates of the average number of unique messenger RNAs that are targets for repression by a typical miRNA vary, depending on the estimation method,[45] but multiple approaches show that mammalian miRNAs can have many unique targets. For example, an analysis of the miRNAs highly conserved in vertebrates shows that each has, on average, roughly 400 conserved targets.[15] Likewise, experiments show that a single miRNA species can reduce the stability of hundreds of unique messenger RNAs.[46] Other experiments show that a single miRNA species may repress the production of hundreds of proteins, but that this repression often is relatively mild (much less than 2-fold).[47][48] The first human disease discovered to be associated with deregulation of miRNAs was chronic lymphocytic leukemia. Other B cell malignancies followed.
Further information on the Poly(A)-binding protein: Paip2b
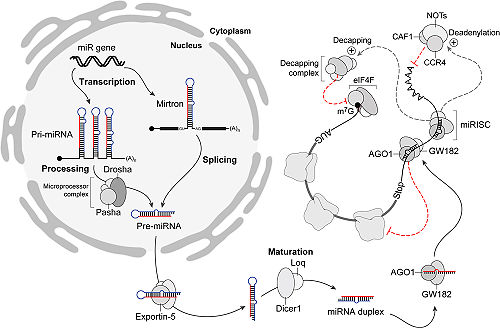
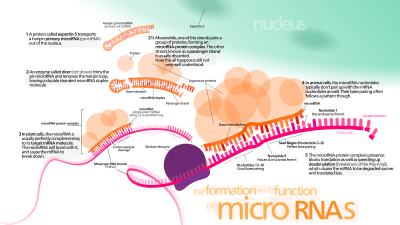
Biogenesis[edit]
As many as 40% of miRNA genes may lie in the introns or even exons of other genes.[49] These are usually, though not exclusively, found in a sense orientation,[50][51] and thus usually are regulated together with their host genes.[49][52][53]
The DNA template is not the final word on mature miRNA production: 6% of human miRNAs show RNA editing (IsomiRs), the site-specific modification of RNA sequences to yield products different from those encoded by their DNA. This increases the diversity and scope of miRNA action beyond that implicated from the genome alone.
Transcription[edit]
miRNA genes are usually transcribed by RNA polymerase II (Pol II).[54][55] The polymerase often binds to a promoter found near the DNA sequence, encoding what will become the hairpin loop of the pre-miRNA. The resulting transcript is capped with a specially modified nucleotide at the 5′ end, polyadenylated with multiple adenosines (a poly(A) tail),[54][50] and spliced. Animal miRNAs are initially transcribed as part of one arm of an ∼80 nucleotide RNA stem-loop that in turn forms part of a several hundred nucleotide-long miRNA precursor termed a pri-miRNA.[54][50] When a stem-loop precursor is found in the 3′ UTR, a transcript may serve as a pri-miRNA and a mRNA.[50] RNA polymerase III (Pol III) transcribes some miRNAs, especially those with upstream Alu sequences, transfer RNAs (tRNAs), and mammalian wide interspersed repeat (MWIR) promoter units.[56]
Nuclear processing[edit]
A single pri-miRNA may contain from one to six miRNA precursors. These hairpin loop structures are composed of about 70 nucleotides each. Each hairpin is flanked by sequences necessary for efficient processing.
The double-stranded RNA (dsRNA) structure of the hairpins in a pri-miRNA is recognized by a nuclear protein known as DiGeorge Syndrome Critical Region 8 (DGCR8 or «Pasha» in invertebrates), named for its association with DiGeorge Syndrome. DGCR8 associates with the enzyme Drosha, a protein that cuts RNA, to form the Microprocessor complex.[57][58] In this complex, DGCR8 orients the catalytic RNase III domain of Drosha to liberate hairpins from pri-miRNAs by cleaving RNA about eleven nucleotides from the hairpin base (one helical dsRNA turn into the stem).[59][60] The product resulting has a two-nucleotide overhang at its 3′ end; it has 3′ hydroxyl and 5′ phosphate groups. It is often termed as a pre-miRNA (precursor-miRNA). Sequence motifs downstream of the pre-miRNA that are important for efficient processing have been identified.[61][62][63]
Pre-miRNAs that are spliced directly out of introns, bypassing the Microprocessor complex, are known as «Mirtrons.» Originally thought to exist only in Drosophila and C. elegans, mirtrons have now been found in mammals.[64]
As many as 16% of pre-miRNAs may be altered through nuclear RNA editing.[65][66][67] Most commonly, enzymes known as adenosine deaminases acting on RNA (ADARs) catalyze adenosine to inosine (A to I) transitions. RNA editing can halt nuclear processing (for example, of pri-miR-142, leading to degradation by the ribonuclease Tudor-SN) and alter downstream processes including cytoplasmic miRNA processing and target specificity (e.g., by changing the seed region of miR-376 in the central nervous system).[65]
Nuclear export[edit]
The human exportin-5 protein (red) in complex with Ran-GTP (yellow) and a pre-microRNA (green), showing two-nucleotide overhang recognition element (orange). From PDB: 3A6P.
Pre-miRNA hairpins are exported from the nucleus in a process involving the nucleocytoplasmic shuttler Exportin-5. This protein, a member of the karyopherin family, recognizes a two-nucleotide overhang left by the RNase III enzyme Drosha at the 3′ end of the pre-miRNA hairpin. Exportin-5-mediated transport to the cytoplasm is energy-dependent, using guanosine triphosphate (GTP) bound to the Ran protein.[68]
Cytoplasmic processing[edit]
In the cytoplasm, the pre-miRNA hairpin is cleaved by the RNase III enzyme Dicer.[69] This endoribonuclease interacts with 5′ and 3′ ends of the hairpin[70] and cuts away the loop joining the 3′ and 5′ arms, yielding an imperfect miRNA:miRNA* duplex about 22 nucleotides in length.[69] Overall hairpin length and loop size influence the efficiency of Dicer processing. The imperfect nature of the miRNA:miRNA* pairing also affects cleavage.[69][71] Some of the G-rich pre-miRNAs can potentially adopt the G-quadruplex structure as an alternative to the canonical stem-loop structure. For example, human pre-miRNA 92b adopts a G-quadruplex structure which is resistant to the Dicer mediated cleavage in the cytoplasm.[72] Although either strand of the duplex may potentially act as a functional miRNA, only one strand is usually incorporated into the RNA-induced silencing complex (RISC) where the miRNA and its mRNA target interact.
While the majority of miRNAs are located within the cell, some miRNAs, commonly known as circulating miRNAs or extracellular miRNAs, have also been found in extracellular environment, including various biological fluids and cell culture media.[73][74]
Biogenesis in plants[edit]
miRNA biogenesis in plants differs from animal biogenesis mainly in the steps of nuclear processing and export. Instead of being cleaved by two different enzymes, once inside and once outside the nucleus, both cleavages of the plant miRNA are performed by a Dicer homolog, called Dicer-like1 (DL1). DL1 is expressed only in the nucleus of plant cells, which indicates that both reactions take place inside the nucleus. Before plant miRNA:miRNA* duplexes are transported out of the nucleus, its 3′ overhangs are methylated by a RNA methyltransferaseprotein called Hua-Enhancer1 (HEN1). The duplex is then transported out of the nucleus to the cytoplasm by a protein called Hasty (HST), an Exportin 5 homolog, where they disassemble and the mature miRNA is incorporated into the RISC.[75]
RNA-induced silencing complex[edit]
The mature miRNA is part of an active RNA-induced silencing complex (RISC) containing Dicer and many associated proteins.[76] RISC is also known as a microRNA ribonucleoprotein complex (miRNP);[77] A RISC with incorporated miRNA is sometimes referred to as a «miRISC.»
Dicer processing of the pre-miRNA is thought to be coupled with unwinding of the duplex. Generally, only one strand is incorporated into the miRISC, selected on the basis of its thermodynamic instability and weaker base-pairing on the 5′ end relative to the other strand.[78][79][80] The position of the stem-loop may also influence strand choice.[81] The other strand, called the passenger strand due to its lower levels in the steady state, is denoted with an asterisk (*) and is normally degraded. In some cases, both strands of the duplex are viable and become functional miRNA that target different mRNA populations.[82]
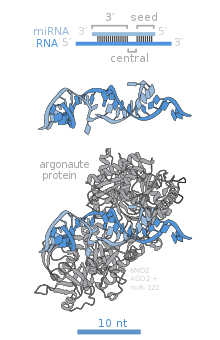
AGO2 (grey) in complex with a microRNA (light blue) and its target mRNA (dark blue)
Members of the Argonaute (Ago) protein family are central to RISC function. Argonautes are needed for miRNA-induced silencing and contain two conserved RNA binding domains: a PAZ domain that can bind the single stranded 3′ end of the mature miRNA and a PIWI domain that structurally resembles ribonuclease-H and functions to interact with the 5′ end of the guide strand. They bind the mature miRNA and orient it for interaction with a target mRNA. Some argonautes, for example human Ago2, cleave target transcripts directly; argonautes may also recruit additional proteins to achieve translational repression.[83] The human genome encodes eight argonaute proteins divided by sequence similarities into two families: AGO (with four members present in all mammalian cells and called E1F2C/hAgo in humans), and PIWI (found in the germ line and hematopoietic stem cells).[77][83]
Additional RISC components include TRBP [human immunodeficiency virus (HIV) transactivating response RNA (TAR) binding protein],[84] PACT (protein activator of the interferon-induced protein kinase), the SMN complex, fragile X mental retardation protein (FMRP), Tudor staphylococcal nuclease-domain-containing protein (Tudor-SN), the putative DNA helicase MOV10, and the RNA recognition motif containing protein TNRC6B.[68][85][86]
Mode of silencing and regulatory loops[edit]
Gene silencing may occur either via mRNA degradation or preventing mRNA from being translated. For example, miR16 contains a sequence complementary to the AU-rich element found in the 3’UTR of many unstable mRNAs, such as TNF alpha or GM-CSF.[87] It has been demonstrated that given complete complementarity between the miRNA and target mRNA sequence, Ago2 can cleave the mRNA and lead to direct mRNA degradation. In the absence of complementarity, silencing is achieved by preventing translation.[46] The relation of miRNA and its target mRNA can be based on the simple negative regulation of a target mRNA, but it seems that a common scenario is the use of a «coherent feed-forward loop», «mutual negative feedback loop» (also termed double negative loop) and «positive feedback/feed-forward loop». Some miRNAs work as buffers of random gene expression changes arising due to stochastic events in transcription, translation and protein stability. Such regulation is typically achieved by the virtue of negative feedback loops or incoherent feed-forward loop uncoupling protein output from mRNA transcription.
Turnover[edit]
Turnover of mature miRNA is needed for rapid changes in miRNA expression profiles. During miRNA maturation in the cytoplasm, uptake by the Argonaute protein is thought to stabilize the guide strand, while the opposite (* or «passenger») strand is preferentially destroyed. In what has been called a «Use it or lose it» strategy, Argonaute may preferentially retain miRNAs with many targets over miRNAs with few or no targets, leading to degradation of the non-targeting molecules.[88]
Decay of mature miRNAs in Caenorhabditis elegans is mediated by the 5′-to-3′ exoribonuclease XRN2, also known as Rat1p.[89] In plants, SDN (small RNA degrading nuclease) family members degrade miRNAs in the opposite (3′-to-5′) direction. Similar enzymes are encoded in animal genomes, but their roles have not been described.[88]
Several miRNA modifications affect miRNA stability. As indicated by work in the model organism Arabidopsis thaliana (thale cress), mature plant miRNAs appear to be stabilized by the addition of methyl moieties at the 3′ end. The 2′-O-conjugated methyl groups block the addition of uracil (U) residues by uridyltransferase enzymes, a modification that may be associated with miRNA degradation. However, uridylation may also protect some miRNAs; the consequences of this modification are incompletely understood. Uridylation of some animal miRNAs has been reported. Both plant and animal miRNAs may be altered by addition of adenine (A) residues to the 3′ end of the miRNA. An extra A added to the end of mammalian miR-122, a liver-enriched miRNA important in hepatitis C, stabilizes the molecule and plant miRNAs ending with an adenine residue have slower decay rates.[88]
Cellular functions[edit]
Interaction of microRNA with protein translation process. Several translation repression mechanisms are shown: M1) on the initiation process, preventing assembling of the initiation complex or recruiting the 40S ribosomal subunit; M2) on the ribosome assembly; M3) on the translation process; M7, M8) on the degradation of mRNA.[90] 40S and 60S are light and heavy components of the ribosome, 80S is the assembled ribosome bound to mRNA, eIF4F is a translation initiation factor, PABC1 is the Poly-A binding protein, and «cap» is the mRNA cap structure needed for mRNA circularization (which can be the normal m7G-cap or modified A-cap). The initiation of mRNA can proceed in a cap-independent manner, through recruiting 40S to IRES (Internal Ribosome Entry Site) located in 5’UTR region. The actual work of RNA silencing is performed by RISC in which the main catalytic subunit is one of the Argonaute proteins (AGO), and miRNA serves as a template for recognizing specific mRNA sequences.
The function of miRNAs appears to be in gene regulation. For that purpose, a miRNA is complementary to a part of one or more messenger RNAs (mRNAs). Animal miRNAs are usually complementary to a site in the 3′ UTR whereas plant miRNAs are usually complementary to coding regions of mRNAs.[91] Perfect or near perfect base pairing with the target RNA promotes cleavage of the RNA.[92] This is the primary mode of plant miRNAs.[93] In animals the match-ups are imperfect.
For partially complementary microRNAs to recognise their targets, nucleotides 2–7 of the miRNA (its ‘seed region’[14][41]) must be perfectly complementary.[94] Animal miRNAs inhibit protein translation of the target mRNA[95] (this is present but less common in plants).[93] Partially complementary microRNAs can also speed up deadenylation, causing mRNAs to be degraded sooner.[96] While degradation of miRNA-targeted mRNA is well documented, whether or not translational repression is accomplished through mRNA degradation, translational inhibition, or a combination of the two is hotly debated. Recent work on miR-430 in zebrafish, as well as on bantam-miRNA and miR-9 in Drosophila cultured cells, shows that translational repression is caused by the disruption of translation initiation, independent of mRNA deadenylation.[97][98]
miRNAs occasionally also cause histone modification and DNA methylation of promoter sites, which affects the expression of target genes.[99][100]
Nine mechanisms of miRNA action are described and assembled in a unified mathematical model:[90]
- Cap-40S initiation inhibition;
- 60S Ribosomal unit joining inhibition;
- Elongation inhibition;
- Ribosome drop-off (premature termination);
- Co-translational nascent protein degradation;
- Sequestration in P-bodies;
- mRNA decay (destabilisation);
- mRNA cleavage;
- Transcriptional inhibition through microRNA-mediated chromatin reorganization followed by gene silencing.
It is often impossible to discern these mechanisms using experimental data about stationary reaction rates. Nevertheless, they are differentiated in dynamics and have different kinetic signatures.[90]
Unlike plant microRNAs, the animal microRNAs target diverse genes.[41] However, genes involved in functions common to all cells, such as gene expression, have relatively fewer microRNA target sites and seem to be under selection to avoid targeting by microRNAs.[101] There is a strong correlation between ITPR gene regulations and mir-92 and mir-19.[102]
dsRNA can also activate gene expression, a mechanism that has been termed «small RNA-induced gene activation» or RNAa. dsRNAs targeting gene promoters can induce potent transcriptional activation of associated genes. This was demonstrated in human cells using synthetic dsRNAs termed small activating RNAs (saRNAs),[103] but has also been demonstrated for endogenous microRNA.[104]
Interactions between microRNAs and complementary sequences on genes and even pseudogenes that share sequence homology are thought to be a back channel of communication regulating expression levels between paralogous genes (genes having a similar structure indicating divergence from a common ancestral gene). Given the name «competing endogenous RNAs» (ceRNAs), these microRNAs bind to «microRNA response elements» on genes and pseudogenes and may provide another explanation for the persistence of non-coding DNA.[105]
Some researches show that mRNA cargo of exosomes may have a role in implantation, they can savage an adhesion between trophoblast and endometrium or support the adhesion by down regulating or up regulating expression of genes involved in adhesion/invasion.[106]
Moreover, miRNA as miR-183/96/182 seems to play a key role in circadian rhythm.[107]
Evolution[edit]
miRNAs are well conserved in both plants and animals, and are thought to be a vital and evolutionarily ancient component of gene regulation.[108][109][110][111][112] While core components of the microRNA pathway are conserved between plants and animals, miRNA repertoires in the two kingdoms appear to have emerged independently with different primary modes of action.[113][114]
microRNAs are useful phylogenetic markers because of their apparently low rate of evolution.[115] microRNAs’ origin as a regulatory mechanism developed from previous RNAi machinery that was initially used as a defense against exogenous genetic material such as viruses.[116] Their origin may have permitted the development of morphological innovation, and by making gene expression more specific and ‘fine-tunable’, permitted the genesis of complex organs[117] and perhaps, ultimately, complex life.[112] Rapid bursts of morphological innovation are generally associated with a high rate of microRNA accumulation.[115][117]
New microRNAs are created in multiple ways. Novel microRNAs can originate from the random formation of hairpins in «non-coding» sections of DNA (i.e. introns or intergene regions), but also by the duplication and modification of existing microRNAs.[118] microRNAs can also form from inverted duplications of protein-coding sequences, which allows for the creation of a foldback hairpin structure.[119] The rate of evolution (i.e. nucleotide substitution) in recently originated microRNAs is comparable to that elsewhere in the non-coding DNA, implying evolution by neutral drift; however, older microRNAs have a much lower rate of change (often less than one substitution per hundred million years),[112] suggesting that once a microRNA gains a function, it undergoes purifying selection.[118] Individual regions within an miRNA gene face different evolutionary pressures, where regions that are vital for processing and function have higher levels of conservation.[120] At this point, a microRNA is rarely lost from an animal’s genome,[112] although newer microRNAs (thus presumably non-functional) are frequently lost.[118] In Arabidopsis thaliana, the net flux of miRNA genes has been predicted to be between 1.2 and 3.3 genes per million years.[121] This makes them a valuable phylogenetic marker, and they are being looked upon as a possible solution to outstanding phylogenetic problems such as the relationships of arthropods.[122] On the other hand, in multiple cases microRNAs correlate poorly with phylogeny, and it is possible that their phylogenetic concordance largely reflects a limited sampling of microRNAs.[123]
microRNAs feature in the genomes of most eukaryotic organisms, from the brown algae[124] to the animals. However, the difference in how these microRNAs function and the way they are processed suggests that microRNAs arose independently in plants and animals.[125]
Focusing on the animals, the genome of Mnemiopsis leidyi[126] appears to lack recognizable microRNAs, as well as the nuclear proteins Drosha and Pasha, which are critical to canonical microRNA biogenesis. It is the only animal thus far reported to be missing Drosha. MicroRNAs play a vital role in the regulation of gene expression in all non-ctenophore animals investigated thus far except for Trichoplax adhaerens, the only known member of the phylum Placozoa.[127]
Across all species, in excess of 5000 different miRNAs had been identified by March 2010.[128] Whilst short RNA sequences (50 – hundreds of base pairs) of a broadly comparable function occur in bacteria, bacteria lack true microRNAs.[129]
Experimental detection and manipulation[edit]
While researchers focused on miRNA expression in physiological and pathological processes, various technical variables related to microRNA isolation emerged. The stability of stored miRNA samples has been questioned.[74] microRNAs degrade much more easily than mRNAs, partly due to their length, but also because of ubiquitously present RNases. This makes it necessary to cool samples on ice and use RNase-free equipment.[130]
microRNA expression can be quantified in a two-step polymerase chain reaction process of modified RT-PCR followed by quantitative PCR. Variations of this method achieve absolute or relative quantification.[131] miRNAs can also be hybridized to microarrays, slides or chips with probes to hundreds or thousands of miRNA targets, so that relative levels of miRNAs can be determined in different samples.[132] microRNAs can be both discovered and profiled by high-throughput sequencing methods (microRNA sequencing).[133] The activity of an miRNA can be experimentally inhibited using a locked nucleic acid (LNA) oligo, a Morpholino oligo[134][135] or a 2′-O-methyl RNA oligo.[136] A specific miRNA can be silenced by a complementary antagomir. microRNA maturation can be inhibited at several points by steric-blocking oligos.[137][138] The miRNA target site of an mRNA transcript can also be blocked by a steric-blocking oligo.[139] For the «in situ» detection of miRNA, LNA[140] or Morpholino[141] probes can be used. The locked conformation of LNA results in enhanced hybridization properties and increases sensitivity and selectivity, making it ideal for detection of short miRNA.[142]
High-throughput quantification of miRNAs is error prone, for the larger variance (compared to mRNAs) that comes with methodological problems. mRNA-expression is therefore often analyzed to check for miRNA-effects in their levels (e.g. in[143]). Databases can be used to pair mRNA- and miRNA-data that predict miRNA-targets based on their base sequence.[144][145] While this is usually done after miRNAs of interest have been detected (e. g. because of high expression levels), ideas for analysis tools that integrate mRNA- and miRNA-expression information have been proposed.[146][147]
Disease[edit]
Just as miRNA is involved in the normal functioning of eukaryotic cells, so has dysregulation of miRNA been associated with disease. A manually curated, publicly available database, miR2Disease, documents known relationships between miRNA dysregulation and human disease.[148]
Inherited diseases[edit]
A mutation in the seed region of miR-96 causes hereditary progressive hearing loss.[149]
A mutation in the seed region of miR-184 causes hereditary keratoconus with anterior polar cataract.[150]
Deletion of the miR-17~92 cluster causes skeletal and growth defects.[151]
Cancer[edit]
Role of miRNA in a cancer cell
The first human disease known to be associated with miRNA deregulation was chronic lymphocytic leukemia.[152] Many other miRNAs also have links with cancer and accordingly are sometimes referred to as «oncomirs».[153] In malignant B cells miRNAs participate in pathways fundamental to B cell development like B-cell receptor (BCR) signalling, B-cell migration/adhesion, cell-cell interactions in immune niches and the production and class-switching of immunoglobulins. MiRNAs influence B cell maturation, generation of pre-, marginal zone, follicular, B1, plasma and memory B cells.[154]
Another role for miRNA in cancers is to use their expression level for prognosis. In NSCLC samples, low miR-324a levels may serve as an indicator of poor survival.[155] Either high miR-185 or low miR-133b levels may correlate with metastasis and poor survival in colorectal cancer.[156]
Furthermore, specific miRNAs may be associated with certain histological subtypes of colorectal cancer. For instance, expression levels of miR-205 and miR-373 have been shown to be increased in mucinous colorectal cancers and mucin-producing Ulcerative Colitis-associated colon cancers, but not in sporadic colonic adenocarcinoma that lack mucinous components.[157] In-vitro studies suggested that miR-205 and miR-373 may functionally induce different features of mucinous-associated neoplastic progression in intestinal epithelial cells.[157]
Hepatocellular carcinoma cell proliferation may arise from miR-21 interaction with MAP2K3, a tumor repressor gene.[158] Optimal treatment for cancer involves accurately identifying patients for risk-stratified therapy. Those with a rapid response to initial treatment may benefit from truncated treatment regimens, showing the value of accurate disease response measures. Cell-free circulating miRNAs (cimiRNAs) are highly stable in blood, are overexpressed in cancer and are quantifiable within the diagnostic laboratory. In classical Hodgkin lymphoma, plasma miR-21, miR-494, and miR-1973 are promising disease response biomarkers.[159] Circulating miRNAs have the potential to assist clinical decision making and aid interpretation of positron emission tomography combined with computerized tomography. They can be performed at each consultation to assess disease response and detect relapse.
MicroRNAs have the potential to be used as tools or targets for treatment of different cancers.[160] The specific microRNA, miR-506 has been found to work as a tumor antagonist in several studies. A significant number of cervical cancer samples were found to have downregulated expression of miR-506. Additionally, miR-506 works to promote apoptosis of cervical cancer cells, through its direct target hedgehog pathway transcription factor, Gli3.[161][162]
DNA repair and cancer[edit]
Many miRNAs can directly target and inhibit cell cycle genes to control cell proliferation. A new strategy for tumor treatment is to inhibit tumor cell proliferation by repairing the defective miRNA pathway in tumors.[163]
Cancer is caused by the accumulation of mutations from either DNA damage or uncorrected errors in DNA replication.[164] Defects in DNA repair cause the accumulation of mutations, which can lead to cancer.[165] Several genes involved in DNA repair are regulated by microRNAs.[166]
Germ line mutations in DNA repair genes cause only 2–5% of colon cancer cases.[167] However, altered expression of microRNAs, causing DNA repair deficiencies, are frequently associated with cancers and may be an important causal factor. Among 68 sporadic colon cancers with reduced expression of the DNA mismatch repair protein MLH1, most were found to be deficient due to epigenetic methylation of the CpG island of the MLH1 gene.[168] However, up to 15% of MLH1-deficiencies in sporadic colon cancers appeared to be due to over-expression of the microRNA miR-155, which represses MLH1 expression.[169]
In 29–66%[170][171] of glioblastomas, DNA repair is deficient due to epigenetic methylation of the MGMT gene, which reduces protein expression of MGMT. However, for 28% of glioblastomas, the MGMT protein is deficient, but the MGMT promoter is not methylated.[170] In glioblastomas without methylated MGMT promoters, the level of microRNA miR-181d is inversely correlated with protein expression of MGMT and the direct target of miR-181d is the MGMT mRNA 3’UTR (the three prime untranslated region of MGMT mRNA).[170] Thus, in 28% of glioblastomas, increased expression of miR-181d and reduced expression of DNA repair enzyme MGMT may be a causal factor.
HMGA proteins (HMGA1a, HMGA1b and HMGA2) are implicated in cancer, and expression of these proteins is regulated by microRNAs. HMGA expression is almost undetectable in differentiated adult tissues, but is elevated in many cancers. HMGA proteins are polypeptides of ~100 amino acid residues characterized by a modular sequence organization. These proteins have three highly positively charged regions, termed AT hooks, that bind the minor groove of AT-rich DNA stretches in specific regions of DNA. Human neoplasias, including thyroid, prostatic, cervical, colorectal, pancreatic and ovarian carcinomas, show a strong increase of HMGA1a and HMGA1b proteins.[172] Transgenic mice with HMGA1 targeted to lymphoid cells develop aggressive lymphoma, showing that high HMGA1 expression is associated with cancers and that HMGA1 can act as an oncogene.[173] HMGA2 protein specifically targets the promoter of ERCC1, thus reducing expression of this DNA repair gene.[174] ERCC1 protein expression was deficient in 100% of 47 evaluated colon cancers (though the extent to which HGMA2 was involved is not known).[175]
Single Nucleotide polymorphisms (SNPs) can alter the binding of miRNAs on 3’UTRs for example the case of hsa-mir181a and hsa-mir181b on the CDON tumor suppressor gene.[176]
Heart disease[edit]
The global role of miRNA function in the heart has been addressed by conditionally inhibiting miRNA maturation in the murine heart. This revealed that miRNAs play an essential role during its development.[177][178] miRNA expression profiling studies demonstrate that expression levels of specific miRNAs change in diseased human hearts, pointing to their involvement in cardiomyopathies.[179][180][181] Furthermore, animal studies on specific miRNAs identified distinct roles for miRNAs both during heart development and under pathological conditions, including the regulation of key factors important for cardiogenesis, the hypertrophic growth response and cardiac conductance.[178][182][183][184][185][186] Another role for miRNA in cardiovascular diseases is to use their expression levels for diagnosis, prognosis or risk stratification.[187] miRNA’s in animal models have also been linked to cholesterol metabolism and regulation.
miRNA-712[edit]
Murine microRNA-712 is a potential biomarker (i.e. predictor) for atherosclerosis, a cardiovascular disease of the arterial wall associated with lipid retention and inflammation.[188] Non-laminar blood flow also correlates with development of atherosclerosis as mechanosenors of endothelial cells respond to the shear force of disturbed flow (d-flow).[189] A number of pro-atherogenic genes including matrix metalloproteinases (MMPs) are upregulated by d-flow,[189] mediating pro-inflammatory and pro-angiogenic signals. These findings were observed in ligated carotid arteries of mice to mimic the effects of d-flow. Within 24 hours, pre-existing immature miR-712 formed mature miR-712 suggesting that miR-712 is flow-sensitive.[189] Coinciding with these results, miR-712 is also upregulated in endothelial cells exposed to naturally occurring d-flow in the greater curvature of the aortic arch.[189]
Origin[edit]
Pre-mRNA sequence of miR-712 is generated from the murine ribosomal RN45s gene at the internal transcribed spacer region 2 (ITS2).[189] XRN1 is an exonuclease that degrades the ITS2 region during processing of RN45s.[189] Reduction of XRN1 under d-flow conditions therefore leads to the accumulation of miR-712.[189]
Mechanism[edit]
MiR-712 targets tissue inhibitor of metalloproteinases 3 (TIMP3).[189] TIMPs normally regulate activity of matrix metalloproteinases (MMPs) which degrade the extracellular matrix (ECM). Arterial ECM is mainly composed of collagen and elastin fibers, providing the structural support and recoil properties of arteries.[190] These fibers play a critical role in regulation of vascular inflammation and permeability, which are important in the development of atherosclerosis.[191] Expressed by endothelial cells, TIMP3 is the only ECM-bound TIMP.[190] A decrease in TIMP3 expression results in an increase of ECM degradation in the presence of d-flow. Consistent with these findings, inhibition of pre-miR712 increases expression of TIMP3 in cells, even when exposed to turbulent flow.[189]
TIMP3 also decreases the expression of TNFα (a pro-inflammatory regulator) during turbulent flow.[189] Activity of TNFα in turbulent flow was measured by the expression of TNFα-converting enzyme (TACE) in blood. TNFα decreased if miR-712 was inhibited or TIMP3 overexpressed,[189] suggesting that miR-712 and TIMP3 regulate TACE activity in turbulent flow conditions.
Anti-miR-712 effectively suppresses d-flow-induced miR-712 expression and increases TIMP3 expression.[189] Anti-miR-712 also inhibits vascular hyperpermeability, thereby significantly reducing atherosclerosis lesion development and immune cell infiltration.[189]
Human homolog microRNA-205[edit]
The human homolog of miR-712 was found on the RN45s homolog gene, which maintains similar miRNAs to mice.[189] MiR-205 of humans share similar sequences with miR-712 of mice and is conserved across most vertebrates.[189] MiR-205 and miR-712 also share more than 50% of the cell signaling targets, including TIMP3.[189]
When tested, d-flow decreased the expression of XRN1 in humans as it did in mice endothelial cells, indicating a potentially common role of XRN1 in humans.[189]
Kidney disease[edit]
Targeted deletion of Dicer in the FoxD1-derived renal progenitor cells in a murine model resulted in a complex renal phenotype including expansion of nephron progenitors, fewer renin cells, smooth muscle arterioles, progressive mesangial loss and glomerular aneurysms.[192] High throughput whole transcriptome profiling of the FoxD1-Dicer knockout mouse model revealed ectopic upregulation of pro-apoptotic gene, Bcl2L11 (Bim) and dysregulation of the p53 pathway with increase in p53 effector genes including Bax, Trp53inp1, Jun, Cdkn1a, Mmp2, and Arid3a. p53 protein levels remained unchanged, suggesting that FoxD1 stromal miRNAs directly repress p53-effector genes. Using a lineage tracing approach followed by Fluorescent-activated cell sorting, miRNA profiling of the FoxD1-derived cells not only comprehensively defined the transcriptional landscape of miRNAs that are critical for vascular development, but also identified key miRNAs that are likely to modulate the renal phenotype in its absence. These miRNAs include miRs‐10a, 18a, 19b, 24, 30c, 92a, 106a, 130a, 152, 181a, 214, 222, 302a, 370, and 381 that regulate Bcl2L11 (Bim) and miRs‐15b, 18a, 21, 30c, 92a, 106a, 125b‐5p, 145, 214, 222, 296‐5p and 302a that regulate p53-effector genes. Consistent with the profiling results, ectopic apoptosis was observed in the cellular derivatives of the FoxD1 derived progenitor lineage and reiterates the importance of renal stromal miRNAs in cellular homeostasis.[192]
Nervous system[edit]
miRNAs appear to regulate the development and function of the nervous system.[193] Neural miRNAs are involved at various stages of synaptic development, including dendritogenesis (involving miR-132, miR-134 and miR-124), synapse formation[194] and synapse maturation (where miR-134 and miR-138 are thought to be involved).[195] Elimination of miRNA formation in mice by experimental silencing of Dicer has led to pathological outcomes, such as reduced neuronal size and motor abnormalities when silenced in striatal neurons[196] and neurodegeneration when silenced in forebrain neurons.[197] Some studies find altered miRNA expression in Alzheimer’s disease,[198] as well as schizophrenia, bipolar disorder, major depression and anxiety disorders.[199][200][201]
Stroke[edit]
According to the Center for Disease Control and Prevention, Stroke is one of the leading causes of death and long-term disability in America. 87% of the cases are ischemic strokes, which results from blockage in the artery of the brain that carries oxygen-rich blood. The obstruction of the blood flow means the brain cannot receive necessary nutrients, such as oxygen and glucose, and remove wastes, such as carbon dioxide.[202][203] miRNAs plays a role in posttranslational gene silencing by targeting genes in the pathogenesis of cerebral ischemia, such as the inflammatory, angiogenesis, and apoptotic pathway.[204]
Alcoholism[edit]
The vital role of miRNAs in gene expression is significant to addiction, specifically alcoholism.[205] Chronic alcohol abuse results in persistent changes in brain function mediated in part by alterations in gene expression.[205] miRNA global regulation of many downstream genes deems significant regarding the reorganization or synaptic connections or long term neural adaptations involving the behavioral change from alcohol consumption to withdrawal and/or dependence.[206] Up to 35 different miRNAs have been found to be altered in the alcoholic post-mortem brain, all of which target genes that include the regulation of the cell cycle, apoptosis, cell adhesion, nervous system development and cell signaling.[205] Altered miRNA levels were found in the medial prefrontal cortex of alcohol-dependent mice, suggesting the role of miRNA in orchestrating translational imbalances and the creation of differentially expressed proteins within an area of the brain where complex cognitive behavior and decision making likely originate.[207]
miRNAs can be either upregulated or downregulated in response to chronic alcohol use. miR-206 expression increased in the prefrontal cortex of alcohol-dependent rats, targeting the transcription factor brain-derived neurotrophic factor (BDNF) and ultimately reducing its expression. BDNF plays a critical role in the formation and maturation of new neurons and synapses, suggesting a possible implication in synapse growth/synaptic plasticity in alcohol abusers.[208] miR-155, important in regulating alcohol-induced neuroinflammation responses, was found to be upregulated, suggesting the role of microglia and inflammatory cytokines in alcohol pathophysiology.[209] Downregulation of miR-382 was found in the nucleus accumbens, a structure in the basal forebrain significant in regulating feelings of reward that power motivational habits. miR-382 is the target for the dopamine receptor D1 (DRD1), and its overexpression results in the upregulation of DRD1 and delta fosB, a transcription factor that activates a series of transcription events in the nucleus accumbens that ultimately result in addictive behaviors.[210] Alternatively, overexpressing miR-382 resulted in attenuated drinking and the inhibition of DRD1 and delta fosB upregulation in rat models of alcoholism, demonstrating the possibility of using miRNA-targeted pharmaceuticals in treatments.[210]
Obesity[edit]
miRNAs play crucial roles in the regulation of stem cell progenitors differentiating into adipocytes.[211] Studies to determine what role pluripotent stem cells play in adipogenesis, were examined in the immortalized human bone marrow-derived stromal cell line hMSC-Tert20.[212] Decreased expression of miR-155, miR-221, and miR-222, have been found during the adipogenic programming of both immortalized and primary hMSCs, suggesting that they act as negative regulators of differentiation. Conversely, ectopic expression of the miRNAs 155, 221, and 222 significantly inhibited adipogenesis and repressed induction of the master regulators PPARγ and CCAAT/enhancer-binding protein alpha (CEBPA).[213] This paves the way for possible genetic obesity treatments.
Another class of miRNAs that regulate insulin resistance, obesity, and diabetes, is the let-7 family. Let-7 accumulates in human tissues during the course of aging.[214] When let-7 was ectopically overexpressed to mimic accelerated aging, mice became insulin-resistant, and thus more prone to high fat diet-induced obesity and diabetes.[215] In contrast when let-7 was inhibited by injections of let-7-specific antagomirs, mice become more insulin-sensitive and remarkably resistant to high fat diet-induced obesity and diabetes. Not only could let-7 inhibition prevent obesity and diabetes, it could also reverse and cure the condition.[216] These experimental findings suggest that let-7 inhibition could represent a new therapy for obesity and type 2 diabetes.
Hemostasis[edit]
miRNAs also play crucial roles in the regulation of complex enzymatic cascades including the hemostatic blood coagulation system.[217] Large scale studies of functional miRNA targeting have recently uncovered rationale therapeutic targets in the hemostatic system.[218][219] They have been directly linked to Calcium homeostasis in the endoplasmic reticulum, which is critical in cell differentiation in early development.[220]
Plants[edit]
miRNAs are considered to be key regulators of many developmental, homeostatic, and immune processes in plants.[221] Their roles in plant development include shoot apical meristem development, leaf growth, flower formation, seed production, or root expansion.[222][223][224][225] In addition, they play a complex role in responses to various abiotic stresses comprising heat stress, low-temperature stress, drought stress, light stress, or gamma radiation exposure.[221]
Viruses[edit]
Viral microRNAs play an important role in the regulation of gene expression of viral and/or host genes to benefit the virus. Hence, miRNAs play a key role in host–virus interactions and pathogenesis of viral diseases.[226][227] The expression of transcription activators by human herpesvirus-6 DNA is believed to be regulated by viral miRNA.[228]
Target prediction[edit]
miRNAs can bind to target messenger RNA (mRNA) transcripts of protein-coding genes and negatively control their translation or cause mRNA degradation. It is of key importance to identify the miRNA targets accurately.[229] A comparison of the predictive performance of eighteen in silico algorithms is available.[230] Large scale studies of functional miRNA targeting suggest that many functional miRNAs can be missed by target prediction algorithms.[218]
See also[edit]
- Anti-miRNA oligonucleotides
- Gene expression
- List of miRNA gene prediction tools
- List of miRNA target prediction tools
- MicroDNA
- MiRNEST
- MIR222
- miR-324-5p
- RNA interference
- Small interfering RNA
- Small nucleolar RNA-derived microRNA
- C19MC miRNA cluster
References[edit]
- ^ Green, D; Dalmay, T; Chapman, T (February 2016). «Microguards and micromessengers of the genome». Heredity. 116 (2): 125–134. doi:10.1038/hdy.2015.84. PMC 4806885. PMID 26419338.
- ^ a b c Bartel DP (March 2018). «Metazoan MicroRNAs». Cell. 173 (1): 20–51. doi:10.1016/j.cell.2018.03.006. PMC 6091663. PMID 29570994.
- ^ Qureshi A, Thakur N, Monga I, Thakur A, Kumar M (1 January 2014). «VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets». Database. 2014: bau103. doi:10.1093/database/bau103. PMC 4224276. PMID 25380780.
- ^ a b c d Bartel DP (January 2009). «MicroRNAs: target recognition and regulatory functions». Cell. 136 (2): 215–33. doi:10.1016/j.cell.2009.01.002. PMC 3794896. PMID 19167326.
- ^ Fabian MR, Sonenberg N, Filipowicz W (2010). «Regulation of mRNA translation and stability by microRNAs». Annual Review of Biochemistry. 79: 351–79. doi:10.1146/annurev-biochem-060308-103103. PMID 20533884.
- ^ a b Bartel DP (January 2004). «MicroRNAs: genomics, biogenesis, mechanism, and function». Cell. 116 (2): 281–297. doi:10.1016/S0092-8674(04)00045-5. PMID 14744438.
- ^ Homo sapiens miRNAs in the miRBase at Manchester University
- ^ Alles J, Fehlmann T, Fischer U, Backes C, Galata V, Minet M, et al. (April 2019). «An estimate of the total number of true human miRNAs». Nucleic Acids Research. 47 (7): 3353–3364. doi:10.1093/nar/gkz097. PMC 6468295. PMID 30820533.
- ^ Fromm B, Domanska D, Høye E, Ovchinnikov V, Kang W, Aparicio-Puerta E, et al. (January 2020). «MirGeneDB 2.0: the metazoan microRNA complement». Nucleic Acids Research. 48 (D1): D132–D141. doi:10.1093/nar/gkz885. PMC 6943042. PMID 31598695.
- ^ Lim LP, Lau NC, Weinstein EG, Abdelhakim A, Yekta S, Rhoades MW, Burge CB, Bartel DP (April 2003). «The microRNAs of Caenorhabditis elegans». Genes & Development. 17 (8): 991–1008. doi:10.1101/gad.1074403. PMC 196042. PMID 12672692.
- ^ a b Lagos-Quintana M, Rauhut R, Yalcin A, Meyer J, Lendeckel W, Tuschl T (April 2002). «Identification of tissue-specific microRNAs from mouse». Current Biology. 12 (9): 735–9. doi:10.1016/S0960-9822(02)00809-6. PMID 12007417.
- ^ a b Kumar S, Reddy PH (September 2016). «Are circulating microRNAs peripheral biomarkers for Alzheimer’s disease?». Biochim Biophys Acta. 1862 (9): 1617–27. doi:10.1016/j.bbadis.2016.06.001. PMC 5343750. PMID 27264337.
- ^ van den Berg MM, Krauskopf J, Ramaekers JG, et al. (February 2020). «Circulating microRNAs as potential biomarkers for psychiatric and neurodegenerative disorders». Prog Neurobiol. 185: 101732. doi:10.1016/j.pneurobio.2019.101732. PMID 31816349.
- ^ a b c Lewis BP, Burge CB, Bartel DP (January 2005). «Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets». Cell. 120 (1): 15–20. doi:10.1016/j.cell.2004.12.035. PMID 15652477.
- ^ a b c Friedman RC, Farh KK, Burge CB, Bartel DP (January 2009). «Most mammalian mRNAs are conserved targets of microRNAs». Genome Research. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ^ Fromm B, Billipp T, Peck LE, Johansen M, Tarver JE, King BL, et al. (2015). «A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome». Annual Review of Genetics. 49: 213–42. doi:10.1146/annurev-genet-120213-092023. PMC 4743252. PMID 26473382.
- ^ a b c Lee RC, Feinbaum RL, Ambros V (December 1993). «The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14». Cell. 75 (5): 843–54. doi:10.1016/0092-8674(93)90529-Y. PMID 8252621.
- ^ a b Reinhart BJ, Slack FJ, Basson M, Pasquinelli AE, Bettinger JC, Rougvie AE, Horvitz HR, Ruvkun G (February 2000). «The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans». Nature. 403 (6772): 901–6. Bibcode:2000Natur.403..901R. doi:10.1038/35002607. PMID 10706289. S2CID 4384503.
- ^ a b Pasquinelli AE, Reinhart BJ, Slack F, Martindale MQ, Kuroda MI, Maller B, Hayward DC, Ball EE, Degnan B, Müller P, Spring J, Srinivasan A, Fishman M, Finnerty J, Corbo J, Levine M, Leahy P, Davidson E, Ruvkun G (November 2000). «Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA». Nature. 408 (6808): 86–9. Bibcode:2000Natur.408…86P. doi:10.1038/35040556. PMID 11081512. S2CID 4401732.
- ^ a b c Lagos-Quintana M, Rauhut R, Lendeckel W, Tuschl T (October 2001). «Identification of novel genes coding for small expressed RNAs». Science. 294 (5543): 853–8. Bibcode:2001Sci…294..853L. doi:10.1126/science.1064921. hdl:11858/00-001M-0000-0012-F65F-2. PMID 11679670. S2CID 18101169.
- ^ a b c Lau NC, Lim LP, Weinstein EG, Bartel DP (October 2001). «An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans». Science. 294 (5543): 858–62. Bibcode:2001Sci…294..858L. doi:10.1126/science.1065062. PMID 11679671. S2CID 43262684.
- ^ a b c Lee RC, Ambros V (October 2001). «An extensive class of small RNAs in Caenorhabditis elegans». Science. 294 (5543): 862–4. Bibcode:2001Sci…294..862L. doi:10.1126/science.1065329. PMID 11679672. S2CID 33480585.
- ^ Wienholds E, Kloosterman WP, Miska E, Alvarez-Saavedra E, Berezikov E, de Bruijn E, Horvitz HR, Kauppinen S, Plasterk RH (July 2005). «MicroRNA expression in zebrafish embryonic development». Science. 309 (5732): 310–1. Bibcode:2005Sci…309..310W. doi:10.1126/science.1114519. PMID 15919954. S2CID 38939571.
- ^ a b Jones-Rhoades MW, Bartel DP, Bartel B (2006). «MicroRNAS and their regulatory roles in plants». Annual Review of Plant Biology. 57: 19–53. doi:10.1146/annurev.arplant.57.032905.105218. PMID 16669754.
- ^ Brennecke J, Hipfner DR, Stark A, Russell RB, Cohen SM (April 2003). «bantam encodes a developmentally regulated microRNA that controls cell proliferation and regulates the proapoptotic gene hid in Drosophila». Cell. 113 (1): 25–36. doi:10.1016/S0092-8674(03)00231-9. PMID 12679032.
- ^ Cuellar TL, McManus MT (December 2005). «MicroRNAs and endocrine biology». The Journal of Endocrinology. 187 (3): 327–32. doi:10.1677/joe.1.06426. PMID 16423811.
- ^ Poy MN, Eliasson L, Krutzfeldt J, Kuwajima S, Ma X, Macdonald PE, Pfeffer S, Tuschl T, Rajewsky N, Rorsman P, Stoffel M (November 2004). «A pancreatic islet-specific microRNA regulates insulin secretion». Nature. 432 (7014): 226–30. Bibcode:2004Natur.432..226P. doi:10.1038/nature03076. PMID 15538371. S2CID 4415988.
- ^ Chen CZ, Li L, Lodish HF, Bartel DP (January 2004). «MicroRNAs modulate hematopoietic lineage differentiation». Science. 303 (5654): 83–6. Bibcode:2004Sci…303…83C. doi:10.1126/science.1091903. hdl:1721.1/7483. PMID 14657504. S2CID 7044929.
- ^ Wilfred BR, Wang WX, Nelson PT (July 2007). «Energizing miRNA research: a review of the role of miRNAs in lipid metabolism, with a prediction that miR-103/107 regulates human metabolic pathways». Molecular Genetics and Metabolism. 91 (3): 209–17. doi:10.1016/j.ymgme.2007.03.011. PMC 1978064. PMID 17521938.
- ^ Harfe BD, McManus MT, Mansfield JH, Hornstein E, Tabin CJ (August 2005). «The RNaseIII enzyme Dicer is required for morphogenesis but not patterning of the vertebrate limb». Proceedings of the National Academy of Sciences of the United States of America. 102 (31): 10898–903. Bibcode:2005PNAS..10210898H. doi:10.1073/pnas.0504834102. PMC 1182454. PMID 16040801.
- ^ Trang P, Weidhaas JB, Slack FJ (December 2008). «MicroRNAs as potential cancer therapeutics». Oncogene. 27 (Suppl 2): S52–7. doi:10.1038/onc.2009.353. PMID 19956180.
- ^ Li C, Feng Y, Coukos G, Zhang L (December 2009). «Therapeutic microRNA strategies in human cancer». The AAPS Journal. 11 (4): 747–57. doi:10.1208/s12248-009-9145-9. PMC 2782079. PMID 19876744.
- ^ Fasanaro P, Greco S, Ivan M, Capogrossi MC, Martelli F (January 2010). «microRNA: emerging therapeutic targets in acute ischemic diseases». Pharmacology & Therapeutics. 125 (1): 92–104. doi:10.1016/j.pharmthera.2009.10.003. PMID 19896977.
- ^ Hydbring P, Badalian-Very G (August 2013). «Clinical applications of microRNAs». F1000Research. 2: 136. doi:10.12688/f1000research.2-136.v2. PMC 3917658. PMID 24627783.
- ^ Wightman B, Ha I, Ruvkun G (December 1993). «Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans». Cell. 75 (5): 855–62. doi:10.1016/0092-8674(93)90530-4. PMID 8252622.
- ^ Giza DE, Calin GA (2015). «microRNA and Chronic Lymphocytic Leukemia». Advances in Experimental Medicine and Biology. 889: 23–40. doi:10.1007/978-3-319-23730-5_2. ISBN 978-3-319-23729-9. PMID 26658994.
- ^ Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T (March 2003). «A uniform system for microRNA annotation». RNA. 9 (3): 277–9. doi:10.1261/rna.2183803. PMC 1370393. PMID 12592000.
- ^ Griffiths-Jones S, Grocock RJ, van Dongen S, Bateman A, Enright AJ (January 2006). «miRBase: microRNA sequences, targets and gene nomenclature». Nucleic Acids Research. 34 (Database issue): D140–4. doi:10.1093/nar/gkj112. PMC 1347474. PMID 16381832.
- ^ Wright MW, Bruford EA (January 2011). «Naming ‘junk’: human non-protein coding RNA (ncRNA) gene nomenclature». Human Genomics. 5 (2): 90–8. doi:10.1186/1479-7364-5-2-90. PMC 3051107. PMID 21296742.
- ^ Hunt M, Banerjee S, Surana P, Liu M, Fuerst G, Mathioni S, Meyers BC, Nettleton D, Wise RP (2019). «Small RNA discovery in the interaction between barley and the powdery mildew pathogen». BMC Genomics. 20 (1): 19–53. doi:10.1186/s12864-019-5947-z. PMC 6657096. PMID 31345162.
- ^ a b c Lewis BP, Shih IH, Jones-Rhoades MW, Bartel DP, Burge CB (December 2003). «Prediction of mammalian microRNA targets». Cell. 115 (7): 787–98. doi:10.1016/S0092-8674(03)01018-3. PMID 14697198.
- ^ Ellwanger DC, Büttner FA, Mewes HW, Stümpflen V (May 2011). «The sufficient minimal set of miRNA seed types». Bioinformatics. 27 (10): 1346–50. doi:10.1093/bioinformatics/btr149. PMC 3087955. PMID 21441577.
- ^ Rajewsky N (June 2006). «microRNA target predictions in animals». Nature Genetics. 38 (6s): S8–13. doi:10.1038/ng1798. PMID 16736023. S2CID 23496396.
- ^ Krek A, Grün D, Poy MN, Wolf R, Rosenberg L, Epstein EJ, MacMenamin P, da Piedade I, Gunsalus KC, Stoffel M, Rajewsky N (May 2005). «Combinatorial microRNA target predictions». Nature Genetics. 37 (5): 495–500. doi:10.1038/ng1536. PMID 15806104. S2CID 22672750.
- ^ Thomson DW, Bracken CP, Goodall GJ (September 2011). «Experimental strategies for microRNA target identification». Nucleic Acids Research. 39 (16): 6845–53. doi:10.1093/nar/gkr330. PMC 3167600. PMID 21652644.
- ^ a b Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM (February 2005). «Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs». Nature. 433 (7027): 769–73. Bibcode:2005Natur.433..769L. doi:10.1038/nature03315. PMID 15685193. S2CID 4430576.
- ^ Selbach M, Schwanhäusser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N (September 2008). «Widespread changes in protein synthesis induced by microRNAs». Nature. 455 (7209): 58–63. Bibcode:2008Natur.455…58S. doi:10.1038/nature07228. PMID 18668040. S2CID 4429008.
- ^ Baek D, Villén J, Shin C, Camargo FD, Gygi SP, Bartel DP (September 2008). «The impact of microRNAs on protein output». Nature. 455 (7209): 64–71. Bibcode:2008Natur.455…64B. doi:10.1038/nature07242. PMC 2745094. PMID 18668037.
- ^ a b Rodriguez A, Griffiths-Jones S, Ashurst JL, Bradley A (October 2004). «Identification of mammalian microRNA host genes and transcription units». Genome Research. 14 (10A): 1902–10. doi:10.1101/gr.2722704. PMC 524413. PMID 15364901.
- ^ a b c d Cai X, Hagedorn CH, Cullen BR (December 2004). «Human microRNAs are processed from capped, polyadenylated transcripts that can also function as mRNAs». RNA. 10 (12): 1957–66. doi:10.1261/rna.7135204. PMC 1370684. PMID 15525708.
- ^ Weber MJ (January 2005). «New human and mouse microRNA genes found by homology search». The FEBS Journal. 272 (1): 59–73. doi:10.1111/j.1432-1033.2004.04389.x. PMID 15634332. S2CID 32923462.
- ^ Kim YK, Kim VN (February 2007). «Processing of intronic microRNAs». The EMBO Journal. 26 (3): 775–83. doi:10.1038/sj.emboj.7601512. PMC 1794378. PMID 17255951.
- ^ Baskerville S, Bartel DP (March 2005). «Microarray profiling of microRNAs reveals frequent coexpression with neighboring miRNAs and host genes». RNA. 11 (3): 241–7. doi:10.1261/rna.7240905. PMC 1370713. PMID 15701730.
- ^ a b c Lee Y, Kim M, Han J, Yeom KH, Lee S, Baek SH, Kim VN (October 2004). «MicroRNA genes are transcribed by RNA polymerase II». The EMBO Journal. 23 (20): 4051–60. doi:10.1038/sj.emboj.7600385. PMC 524334. PMID 15372072.
- ^ Zhou X, Ruan J, Wang G, Zhang W (March 2007). «Characterization and identification of microRNA core promoters in four model species». PLOS Computational Biology. 3 (3): e37. Bibcode:2007PLSCB…3…37Z. doi:10.1371/journal.pcbi.0030037. PMC 1817659. PMID 17352530.
- ^ Faller M, Guo F (November 2008). «MicroRNA biogenesis: there’s more than one way to skin a cat». Biochimica et Biophysica Acta (BBA) — Gene Regulatory Mechanisms. 1779 (11): 663–7. doi:10.1016/j.bbagrm.2008.08.005. PMC 2633599. PMID 18778799.
- ^ Lee Y, Ahn C, Han J, Choi H, Kim J, Yim J, Lee J, Provost P, Rådmark O, Kim S, Kim VN (September 2003). «The nuclear RNase III Drosha initiates microRNA processing». Nature. 425 (6956): 415–9. Bibcode:2003Natur.425..415L. doi:10.1038/nature01957. PMID 14508493. S2CID 4421030.
- ^ Gregory RI, Chendrimada TP, Shiekhattar R (2006). «MicroRNA biogenesis: isolation and characterization of the microprocessor complex». MicroRNA Protocols. Methods in Molecular Biology. Vol. 342. pp. 33–47. doi:10.1385/1-59745-123-1:33. ISBN 978-1-59745-123-9. PMID 16957365.
- ^ Han J, Lee Y, Yeom KH, Kim YK, Jin H, Kim VN (December 2004). «The Drosha-DGCR8 complex in primary microRNA processing». Genes & Development. 18 (24): 3016–27. doi:10.1101/gad.1262504. PMC 535913. PMID 15574589.
- ^ Han J, Lee Y, Yeom KH, Nam JW, Heo I, Rhee JK, Sohn SY, Cho Y, Zhang BT, Kim VN (June 2006). «Molecular basis for the recognition of primary microRNAs by the Drosha-DGCR8 complex». Cell. 125 (5): 887–901. doi:10.1016/j.cell.2006.03.043. PMID 16751099.
- ^ Conrad T, Marsico A, Gehre M, Orom UA (October 2014). «Microprocessor activity controls differential miRNA biogenesis in Vivo». Cell Reports. 9 (2): 542–54. doi:10.1016/j.celrep.2014.09.007. PMID 25310978.
- ^ Auyeung VC, Ulitsky I, McGeary SE, Bartel DP (February 2013). «Beyond secondary structure: primary-sequence determinants license pri-miRNA hairpins for processing». Cell. 152 (4): 844–58. doi:10.1016/j.cell.2013.01.031. PMC 3707628. PMID 23415231.
- ^ Ali PS, Ghoshdastider U, Hoffmann J, Brutschy B, Filipek S (November 2012). «Recognition of the let-7g miRNA precursor by human Lin28B». FEBS Letters. 586 (22): 3986–90. doi:10.1016/j.febslet.2012.09.034. PMID 23063642. S2CID 28899778.
- ^ Berezikov E, Chung WJ, Willis J, Cuppen E, Lai EC (October 2007). «Mammalian mirtron genes». Molecular Cell. 28 (2): 328–36. doi:10.1016/j.molcel.2007.09.028. PMC 2763384. PMID 17964270.
- ^ a b Kawahara Y, Megraw M, Kreider E, Iizasa H, Valente L, Hatzigeorgiou AG, Nishikura K (September 2008). «Frequency and fate of microRNA editing in human brain». Nucleic Acids Research. 36 (16): 5270–80. doi:10.1093/nar/gkn479. PMC 2532740. PMID 18684997.
- ^ Winter J, Jung S, Keller S, Gregory RI, Diederichs S (March 2009). «Many roads to maturity: microRNA biogenesis pathways and their regulation». Nature Cell Biology. 11 (3): 228–34. doi:10.1038/ncb0309-228. PMID 19255566. S2CID 205286318.
- ^ Ohman M (October 2007). «A-to-I editing challenger or ally to the microRNA process». Biochimie. 89 (10): 1171–6. doi:10.1016/j.biochi.2007.06.002. PMID 17628290.
- ^ a b Murchison EP, Hannon GJ (June 2004). «miRNAs on the move: miRNA biogenesis and the RNAi machinery». Current Opinion in Cell Biology. 16 (3): 223–9. doi:10.1016/j.ceb.2004.04.003. PMID 15145345.
- ^ a b c Lund E, Dahlberg JE (2006). «Substrate selectivity of exportin 5 and Dicer in the biogenesis of microRNAs». Cold Spring Harbor Symposia on Quantitative Biology. 71: 59–66. doi:10.1101/sqb.2006.71.050. PMID 17381281.
- ^ Park JE, Heo I, Tian Y, Simanshu DK, Chang H, Jee D, Patel DJ, Kim VN (July 2011). «Dicer recognizes the 5′ end of RNA for efficient and accurate processing». Nature. 475 (7355): 201–5. doi:10.1038/nature10198. PMC 4693635. PMID 21753850.
- ^ Ji X (2008). «The mechanism of RNase III action: how dicer dices». RNA Interference. Current Topics in Microbiology and Immunology. Vol. 320. pp. 99–116. doi:10.1007/978-3-540-75157-1_5. ISBN 978-3-540-75156-4. PMID 18268841.
- ^ Mirihana Arachchilage G, Dassanayake AC, Basu S (February 2015). «A potassium ion-dependent RNA structural switch regulates human pre-miRNA 92b maturation». Chemistry & Biology. 22 (2): 262–72. doi:10.1016/j.chembiol.2014.12.013. PMID 25641166.
- ^ Sohel MH (2016). «Extracellular/Circulating MicroRNAs: Release Mechanisms, Functions and Challenges». Achievements in the Life Sciences. 10 (2): 175–186. doi:10.1016/j.als.2016.11.007.
- ^ a b Boeckel JN, Reis SM, Leistner D, Thomé CE, Zeiher AM, Fichtlscherer S, Keller T (April 2014). «From heart to toe: heart’s contribution on peripheral microRNA levels». International Journal of Cardiology. 172 (3): 616–7. doi:10.1016/j.ijcard.2014.01.082. PMID 24508494.
- ^ Lelandais-Brière C, Sorin C, Declerck M, Benslimane A, Crespi M, Hartmann C (March 2010). «Small RNA diversity in plants and its impact in development». Current Genomics. 11 (1): 14–23. doi:10.2174/138920210790217918. PMC 2851111. PMID 20808519.
- ^ Rana TM (January 2007). «Illuminating the silence: understanding the structure and function of small RNAs». Nature Reviews Molecular Cell Biology. 8 (1): 23–36. doi:10.1038/nrm2085. PMID 17183358. S2CID 8966239.
- ^ a b Schwarz DS, Zamore PD (May 2002). «Why do miRNAs live in the miRNP?». Genes & Development. 16 (9): 1025–31. doi:10.1101/gad.992502. PMID 12000786.
- ^ Krol J, Sobczak K, Wilczynska U, Drath M, Jasinska A, Kaczynska D, Krzyzosiak WJ (October 2004). «Structural features of microRNA (miRNA) precursors and their relevance to miRNA biogenesis and small interfering RNA/short hairpin RNA design». The Journal of Biological Chemistry. 279 (40): 42230–9. doi:10.1074/jbc.M404931200. PMID 15292246.
- ^ Khvorova A, Reynolds A, Jayasena SD (October 2003). «Functional siRNAs and miRNAs exhibit strand bias». Cell. 115 (2): 209–16. doi:10.1016/S0092-8674(03)00801-8. PMID 14567918.
- ^ Schwarz DS, Hutvágner G, Du T, Xu Z, Aronin N, Zamore PD (October 2003). «Asymmetry in the assembly of the RNAi enzyme complex». Cell. 115 (2): 199–208. doi:10.1016/S0092-8674(03)00759-1. PMID 14567917.
- ^ Lin SL, Chang D, Ying SY (August 2005). «Asymmetry of intronic pre-miRNA structures in functional RISC assembly». Gene. 356: 32–8. doi:10.1016/j.gene.2005.04.036. PMC 1788082. PMID 16005165.
- ^ Okamura K, Chung WJ, Lai EC (September 2008). «The long and short of inverted repeat genes in animals: microRNAs, mirtrons and hairpin RNAs». Cell Cycle. 7 (18): 2840–5. doi:10.4161/cc.7.18.6734. PMC 2697033. PMID 18769156.
- ^ a b Pratt AJ, MacRae IJ (July 2009). «The RNA-induced silencing complex: a versatile gene-silencing machine». The Journal of Biological Chemistry. 284 (27): 17897–901. doi:10.1074/jbc.R900012200. PMC 2709356. PMID 19342379.
- ^ MacRae IJ, Ma E, Zhou M, Robinson CV, Doudna JA (January 2008). «In vitro reconstitution of the human RISC-loading complex». Proceedings of the National Academy of Sciences of the United States of America. 105 (2): 512–7. Bibcode:2008PNAS..105..512M. doi:10.1073/pnas.0710869105. PMC 2206567. PMID 18178619.
- ^ Mourelatos Z, Dostie J, Paushkin S, Sharma A, Charroux B, Abel L, Rappsilber J, Mann M, Dreyfuss G (March 2002). «miRNPs: a novel class of ribonucleoproteins containing numerous microRNAs». Genes & Development. 16 (6): 720–8. doi:10.1101/gad.974702. PMC 155365. PMID 11914277.
- ^ Meister G, Landthaler M, Peters L, Chen PY, Urlaub H, Lührmann R, Tuschl T (December 2005). «Identification of novel argonaute-associated proteins». Current Biology. 15 (23): 2149–55. doi:10.1016/j.cub.2005.10.048. PMID 16289642.
- ^ Jing Q, Huang S, Guth S, Zarubin T, Motoyama A, Chen J, Di Padova F, Lin SC, Gram H, Han J (March 2005). «Involvement of microRNA in AU-rich element-mediated mRNA instability». Cell. 120 (5): 623–34. doi:10.1016/j.cell.2004.12.038. PMID 15766526.
- ^ a b c Kai ZS, Pasquinelli AE (January 2010). «MicroRNA assassins: factors that regulate the disappearance of miRNAs». Nature Structural & Molecular Biology. 17 (1): 5–10. doi:10.1038/nsmb.1762. PMC 6417416. PMID 20051982.
- ^ Chatterjee S, Grosshans H (September 2009). «Active turnover modulates mature microRNA activity in Caenorhabditis elegans». Nature. 461 (7263): 546–9. Bibcode:2009Natur.461..546C. doi:10.1038/nature08349. PMID 19734881. S2CID 4414841.
- ^ a b c Morozova N, Zinovyev A, Nonne N, Pritchard LL, Gorban AN, Harel-Bellan A (September 2012). «Kinetic signatures of microRNA modes of action». RNA. 18 (9): 1635–55. doi:10.1261/rna.032284.112. PMC 3425779. PMID 22850425.
- ^ Wang XJ, Reyes JL, Chua NH, Gaasterland T (2004). «Prediction and identification of Arabidopsis thaliana microRNAs and their mRNA targets». Genome Biology. 5 (9): R65. doi:10.1186/gb-2004-5-9-r65. PMC 522872. PMID 15345049.
- ^ Kawasaki H, Taira K (2004). «MicroRNA-196 inhibits HOXB8 expression in myeloid differentiation of HL60 cells». Nucleic Acids Symposium Series. 48 (1): 211–2. doi:10.1093/nass/48.1.211. PMID 17150553.
- ^ a b Moxon S, Jing R, Szittya G, Schwach F, Rusholme Pilcher RL, Moulton V, Dalmay T (October 2008). «Deep sequencing of tomato short RNAs identifies microRNAs targeting genes involved in fruit ripening». Genome Research. 18 (10): 1602–9. doi:10.1101/gr.080127.108. PMC 2556272. PMID 18653800.
- ^ Mazière P, Enright AJ (June 2007). «Prediction of microRNA targets». Drug Discovery Today. 12 (11–12): 452–8. doi:10.1016/j.drudis.2007.04.002. PMID 17532529.
- ^ Williams AE (February 2008). «Functional aspects of animal microRNAs». Cellular and Molecular Life Sciences. 65 (4): 545–62. doi:10.1007/s00018-007-7355-9. PMID 17965831. S2CID 5708394.
- ^ Eulalio A, Huntzinger E, Nishihara T, Rehwinkel J, Fauser M, Izaurralde E (January 2009). «Deadenylation is a widespread effect of miRNA regulation». RNA. 15 (1): 21–32. doi:10.1261/rna.1399509. PMC 2612776. PMID 19029310.
- ^ Bazzini AA, Lee MT, Giraldez AJ (April 2012). «Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish». Science. 336 (6078): 233–7. Bibcode:2012Sci…336..233B. doi:10.1126/science.1215704. PMC 3547538. PMID 22422859.
- ^ Djuranovic S, Nahvi A, Green R (April 2012). «miRNA-mediated gene silencing by translational repression followed by mRNA deadenylation and decay». Science. 336 (6078): 237–40. Bibcode:2012Sci…336..237D. doi:10.1126/science.1215691. PMC 3971879. PMID 22499947.
- ^ Tan Y, Zhang B, Wu T, Skogerbø G, Zhu X, Guo X, He S, Chen R (February 2009). «Transcriptional inhibiton of Hoxd4 expression by miRNA-10a in human breast cancer cells». BMC Molecular Biology. 10 (1): 12. doi:10.1186/1471-2199-10-12. PMC 2680403. PMID 19232136.
- ^ Hawkins PG, Morris KV (March 2008). «RNA and transcriptional modulation of gene expression». Cell Cycle. 7 (5): 602–7. doi:10.4161/cc.7.5.5522. PMC 2877389. PMID 18256543.
- ^ Stark A, Brennecke J, Bushati N, Russell RB, Cohen SM (December 2005). «Animal MicroRNAs confer robustness to gene expression and have a significant impact on 3’UTR evolution». Cell. 123 (6): 1133–46. doi:10.1016/j.cell.2005.11.023. PMID 16337999.
- ^ He L, Hannon GJ (July 2004). «MicroRNAs: small RNAs with a big role in gene regulation». Nature Reviews. Genetics. 5 (7): 522–531. doi:10.1038/nrg1379. PMID 15211354. S2CID 5270062.
- ^ Li LC (2008). «Small RNA-Mediated Gene Activation». In Morris KV (ed.). RNA and the Regulation of Gene Expression: A Hidden Layer of Complexity. Horizon Scientific Press. ISBN 978-1-904455-25-7.
- ^ Place RF, Li LC, Pookot D, Noonan EJ, Dahiya R (February 2008). «MicroRNA-373 induces expression of genes with complementary promoter sequences». Proceedings of the National Academy of Sciences of the United States of America. 105 (5): 1608–13. Bibcode:2008PNAS..105.1608P. doi:10.1073/pnas.0707594105. PMC 2234192. PMID 18227514.
- ^ Salmena L, Poliseno L, Tay Y, Kats L, Pandolfi PP (August 2011). «A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language?». Cell. 146 (3): 353–8. doi:10.1016/j.cell.2011.07.014. PMC 3235919. PMID 21802130.
- ^ Cuman C, Van Sinderen M, Gantier MP, Rainczuk K, Sorby K, Rombauts L, et al. (October 2015). «Human Blastocyst Secreted microRNA Regulate Endometrial Epithelial Cell Adhesion». EBioMedicine. 2 (10): 1528–1535. doi:10.1016/j.ebiom.2015.09.003. PMC 4634783. PMID 26629549.
- ^ Zhou L, Miller C, Miraglia LJ, Romero A, Mure LS, Panda S, Kay SA (January 2021). «A genome-wide microRNA screen identifies the microRNA-183/96/182 cluster as a modulator of circadian rhythms». Proceedings of the National Academy of Sciences of the United States of America. 118 (1): e2020454118. Bibcode:2021PNAS..11820454Z. doi:10.1073/pnas.2020454118. PMC 7817116. PMID 33443164. S2CID 230713808.
- «MicroRNAs Play Key Role in Regulation of Circadian Rhythms». Science News. 6 January 2021.
- ^ Axtell MJ, Bartel DP (June 2005). «Antiquity of microRNAs and their targets in land plants». The Plant Cell. 17 (6): 1658–73. doi:10.1105/tpc.105.032185. PMC 1143068. PMID 15849273.
- ^ Tanzer A, Stadler PF (May 2004). «Molecular evolution of a microRNA cluster». Journal of Molecular Biology. 339 (2): 327–35. CiteSeerX 10.1.1.194.1598. doi:10.1016/j.jmb.2004.03.065. PMID 15136036.
- ^ Chen K, Rajewsky N (February 2007). «The evolution of gene regulation by transcription factors and microRNAs». Nature Reviews Genetics. 8 (2): 93–103. doi:10.1038/nrg1990. PMID 17230196. S2CID 174231.
- ^ Lee CT, Risom T, Strauss WM (April 2007). «Evolutionary conservation of microRNA regulatory circuits: an examination of microRNA gene complexity and conserved microRNA-target interactions through metazoan phylogeny». DNA and Cell Biology. 26 (4): 209–18. doi:10.1089/dna.2006.0545. PMID 17465887.
- ^ a b c d Peterson KJ, Dietrich MR, McPeek MA (July 2009). «MicroRNAs and metazoan macroevolution: insights into canalization, complexity, and the Cambrian explosion». BioEssays. 31 (7): 736–47. doi:10.1002/bies.200900033. PMID 19472371. S2CID 15364875.
- ^ Shabalina SA, Koonin EV (October 2008). «Origins and evolution of eukaryotic RNA interference». Trends in Ecology & Evolution. 23 (10): 578–87. doi:10.1016/j.tree.2008.06.005. PMC 2695246. PMID 18715673.
- ^ Axtell MJ, Westholm JO, Lai EC (2011). «Vive la différence: biogenesis and evolution of microRNAs in plants and animals». Genome Biology. 12 (4): 221. doi:10.1186/gb-2011-12-4-221. PMC 3218855. PMID 21554756.
- ^ a b Wheeler BM, Heimberg AM, Moy VN, Sperling EA, Holstein TW, Heber S, Peterson KJ (2009). «The deep evolution of metazoan microRNAs». Evolution & Development. 11 (1): 50–68. doi:10.1111/j.1525-142X.2008.00302.x. PMID 19196333. S2CID 14924603.
- ^ Pashkovskiy PP, Ryazansky SS (June 2013). «Biogenesis, evolution, and functions of plant microRNAs». Biochemistry. Biokhimiia. 78 (6): 627–37. doi:10.1134/S0006297913060084. PMID 23980889. S2CID 12025420.
- ^ a b Heimberg AM, Sempere LF, Moy VN, Donoghue PC, Peterson KJ (February 2008). «MicroRNAs and the advent of vertebrate morphological complexity». Proceedings of the National Academy of Sciences of the United States of America. 105 (8): 2946–50. Bibcode:2008PNAS..105.2946H. doi:10.1073/pnas.0712259105. PMC 2268565. PMID 18287013.
- ^ a b c Nozawa M, Miura S, Nei M (July 2010). «Origins and evolution of microRNA genes in Drosophila species». Genome Biology and Evolution. 2: 180–9. doi:10.1093/gbe/evq009. PMC 2942034. PMID 20624724.
- ^ Allen E, Xie Z, Gustafson AM, Sung GH, Spatafora JW, Carrington JC (December 2004). «Evolution of microRNA genes by inverted duplication of target gene sequences in Arabidopsis thaliana». Nature Genetics. 36 (12): 1282–90. doi:10.1038/ng1478. PMID 15565108. S2CID 11997028.
- ^ Warthmann N, Das S, Lanz C, Weigel D (May 2008). «Comparative analysis of the MIR319a microRNA locus in Arabidopsis and related Brassicaceae». Molecular Biology and Evolution. 25 (5): 892–902. doi:10.1093/molbev/msn029. PMID 18296705.
- ^ Fahlgren N, Jogdeo S, Kasschau KD, Sullivan CM, Chapman EJ, Laubinger S, Smith LM, Dasenko M, Givan SA, Weigel D, Carrington JC (April 2010). «MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana». The Plant Cell. 22 (4): 1074–89. doi:10.1105/tpc.110.073999. PMC 2879733. PMID 20407027.
- ^ Caravas J, Friedrich M (June 2010). «Of mites and millipedes: recent progress in resolving the base of the arthropod tree». BioEssays. 32 (6): 488–95. doi:10.1002/bies.201000005. PMID 20486135. S2CID 20548122.
- ^ Kenny NJ, Namigai EK, Marlétaz F, Hui JH, Shimeld SM (December 2015). «Draft genome assemblies and predicted microRNA complements of the intertidal lophotrochozoans Patella vulgata (Mollusca, Patellogastropoda) and Spirobranchus (Pomatoceros) lamarcki (Annelida, Serpulida)». Marine Genomics. 24 (2): 139–46. doi:10.1016/j.margen.2015.07.004. PMID 26319627.
- ^ Cock JM, Sterck L, Rouzé P, Scornet D, Allen AE, Amoutzias G, et al. (June 2010). «The Ectocarpus genome and the independent evolution of multicellularity in brown algae». Nature. 465 (7298): 617–21. Bibcode:2010Natur.465..617C. doi:10.1038/nature09016. PMID 20520714.
- ^ Cuperus JT, Fahlgren N, Carrington JC (February 2011). «Evolution and functional diversification of MIRNA genes». The Plant Cell. 23 (2): 431–42. doi:10.1105/tpc.110.082784. PMC 3077775. PMID 21317375.
- ^ Ryan JF, Pang K, Schnitzler CE, Nguyen AD, Moreland RT, Simmons DK, Koch BJ, Francis WR, Havlak P, Smith SA, Putnam NH, Haddock SH, Dunn CW, Wolfsberg TG, Mullikin JC, Martindale MQ, Baxevanis AD (December 2013). «The genome of the ctenophore Mnemiopsis leidyi and its implications for cell type evolution». Science. 342 (6164): 1242592. doi:10.1126/science.1242592. PMC 3920664. PMID 24337300.
- ^ Maxwell EK, Ryan JF, Schnitzler CE, Browne WE, Baxevanis AD (December 2012). «MicroRNAs and essential components of the microRNA processing machinery are not encoded in the genome of the ctenophore Mnemiopsis leidyi». BMC Genomics. 13 (1): 714. doi:10.1186/1471-2164-13-714. PMC 3563456. PMID 23256903.
- ^ Dimond PF (15 March 2010). «miRNAs’ Therapeutic Potential». Genetic Engineering & Biotechnology News. 30 (6): 1. Archived from the original on 19 July 2010. Retrieved 10 July 2010.
- ^ Tjaden B, Goodwin SS, Opdyke JA, Guillier M, Fu DX, Gottesman S, Storz G (2006). «Target prediction for small, noncoding RNAs in bacteria». Nucleic Acids Research. 34 (9): 2791–802. doi:10.1093/nar/gkl356. PMC 1464411. PMID 16717284.
- ^ Liu CG, Calin GA, Volinia S, Croce CM (2008). «MicroRNA expression profiling using microarrays». Nature Protocols. 3 (4): 563–78. doi:10.1038/nprot.2008.14. PMID 18388938. S2CID 2441105.
- ^ Chen C, Ridzon DA, Broomer AJ, Zhou Z, Lee DH, Nguyen JT, Barbisin M, Xu NL, Mahuvakar VR, Andersen MR, Lao KQ, Livak KJ, Guegler KJ (November 2005). «Real-time quantification of microRNAs by stem-loop RT-PCR». Nucleic Acids Research. 33 (20): e179. doi:10.1093/nar/gni178. PMC 1292995. PMID 16314309.
- ^ Shingara J, Keiger K, Shelton J, Laosinchai-Wolf W, Powers P, Conrad R, Brown D, Labourier E (September 2005). «An optimized isolation and labeling platform for accurate microRNA expression profiling». RNA. 11 (9): 1461–70. doi:10.1261/rna.2610405. PMC 1370829. PMID 16043497.
- ^ Buermans HP, Ariyurek Y, van Ommen G, den Dunnen JT, ‘t Hoen PA (December 2010). «New methods for next generation sequencing based microRNA expression profiling». BMC Genomics. 11: 716. doi:10.1186/1471-2164-11-716. PMC 3022920. PMID 21171994.
- ^ Kloosterman WP, Wienholds E, Ketting RF, Plasterk RH (2004). «Substrate requirements for let-7 function in the developing zebrafish embryo». Nucleic Acids Research. 32 (21): 6284–91. doi:10.1093/nar/gkh968. PMC 535676. PMID 15585662.
- ^ Flynt AS, Li N, Thatcher EJ, Solnica-Krezel L, Patton JG (February 2007). «Zebrafish miR-214 modulates Hedgehog signaling to specify muscle cell fate». Nature Genetics. 39 (2): 259–63. doi:10.1038/ng1953. PMC 3982799. PMID 17220889.
- ^ Meister G, Landthaler M, Dorsett Y, Tuschl T (March 2004). «Sequence-specific inhibition of microRNA- and siRNA-induced RNA silencing». RNA. 10 (3): 544–50. doi:10.1261/rna.5235104. PMC 1370948. PMID 14970398.
- ^ Kloosterman WP, Lagendijk AK, Ketting RF, Moulton JD, Plasterk RH (August 2007). «Targeted inhibition of miRNA maturation with morpholinos reveals a role for miR-375 in pancreatic islet development». PLOS Biology. 5 (8): e203. doi:10.1371/journal.pbio.0050203. PMC 1925136. PMID 17676975.
- ^ Gebert LF, Rebhan MA, Crivelli SE, Denzler R, Stoffel M, Hall J (January 2014). «Miravirsen (SPC3649) can inhibit the biogenesis of miR-122». Nucleic Acids Research. 42 (1): 609–21. doi:10.1093/nar/gkt852. PMC 3874169. PMID 24068553.
- ^ Choi WY, Giraldez AJ, Schier AF (October 2007). «Target protectors reveal dampening and balancing of Nodal agonist and antagonist by miR-430». Science. 318 (5848): 271–4. Bibcode:2007Sci…318..271C. doi:10.1126/science.1147535. PMID 17761850. S2CID 30461594.
- ^ You Y, Moreira BG, Behlke MA, Owczarzy R (May 2006). «Design of LNA probes that improve mismatch discrimination». Nucleic Acids Research. 34 (8): e60. doi:10.1093/nar/gkl175. PMC 1456327. PMID 16670427.
- ^ Lagendijk AK, Moulton JD, Bakkers J (June 2012). «Revealing details: whole mount microRNA in situ hybridization protocol for zebrafish embryos and adult tissues». Biology Open. 1 (6): 566–9. doi:10.1242/bio.2012810. PMC 3509442. PMID 23213449.
- ^ Kaur H, Arora A, Wengel J, Maiti S (June 2006). «Thermodynamic, counterion, and hydration effects for the incorporation of locked nucleic acid nucleotides into DNA duplexes». Biochemistry. 45 (23): 7347–55. doi:10.1021/bi060307w. PMID 16752924.
- ^ Nielsen JA, Lau P, Maric D, Barker JL, Hudson LD (August 2009). «Integrating microRNA and mRNA expression profiles of neuronal progenitors to identify regulatory networks underlying the onset of cortical neurogenesis». BMC Neuroscience. 10: 98. doi:10.1186/1471-2202-10-98. PMC 2736963. PMID 19689821.
- ^ Grimson A, Farh KK, Johnston WK, Garrett-Engele P, Lim LP, Bartel DP (July 2007). «MicroRNA targeting specificity in mammals: determinants beyond seed pairing». Molecular Cell. 27 (1): 91–105. doi:10.1016/j.molcel.2007.06.017. PMC 3800283. PMID 17612493.
- ^ Griffiths-Jones S, Saini HK, van Dongen S, Enright AJ (January 2008). «miRBase: tools for microRNA genomics». Nucleic Acids Research. 36 (Database issue): D154–8. doi:10.1093/nar/gkm952. PMC 2238936. PMID 17991681.
- ^ Nam S, Li M, Choi K, Balch C, Kim S, Nephew KP (July 2009). «MicroRNA and mRNA integrated analysis (MMIA): a web tool for examining biological functions of microRNA expression». Nucleic Acids Research. 37 (Web Server issue): W356–62. doi:10.1093/nar/gkp294. PMC 2703907. PMID 19420067.
- ^ Artmann S, Jung K, Bleckmann A, Beissbarth T (2012). Provero P (ed.). «Detection of simultaneous group effects in microRNA expression and related target gene sets». PLOS ONE. 7 (6): e38365. Bibcode:2012PLoSO…738365A. doi:10.1371/journal.pone.0038365. PMC 3378551. PMID 22723856.
- ^ Jiang Q, Wang Y, Hao Y, Juan L, Teng M, Zhang X, Li M, Wang G, Liu Y (January 2009). «miR2Disease: a manually curated database for microRNA deregulation in human disease». Nucleic Acids Research. 37. 37 (Database issue): D98–104. doi:10.1093/nar/gkn714. PMC 2686559. PMID 18927107.
- ^ Mencía A, Modamio-Høybjør S, Redshaw N, Morín M, Mayo-Merino F, Olavarrieta L, Aguirre LA, del Castillo I, Steel KP, Dalmay T, Moreno F, Moreno-Pelayo MA (May 2009). «Mutations in the seed region of human miR-96 are responsible for nonsyndromic progressive hearing loss». Nature Genetics. 41 (5): 609–13. doi:10.1038/ng.355. PMID 19363479. S2CID 11113852.
- ^ Hughes AE, Bradley DT, Campbell M, Lechner J, Dash DP, Simpson DA, Willoughby CE (November 2011). «Mutation altering the miR-184 seed region causes familial keratoconus with cataract». American Journal of Human Genetics. 89 (5): 628–33. doi:10.1016/j.ajhg.2011.09.014. PMC 3213395. PMID 21996275.
- ^ de Pontual L, Yao E, Callier P, Faivre L, Drouin V, Cariou S, Van Haeringen A, Geneviève D, Goldenberg A, Oufadem M, Manouvrier S, Munnich A, Vidigal JA, Vekemans M, Lyonnet S, Henrion-Caude A, Ventura A, Amiel J (September 2011). «Germline deletion of the miR-17∼92 cluster causes skeletal and growth defects in humans». Nature Genetics. 43 (10): 1026–30. doi:10.1038/ng.915. PMC 3184212. PMID 21892160.
- ^ Calin GA, Liu CG, Sevignani C, Ferracin M, Felli N, Dumitru CD, Shimizu M, Cimmino A, Zupo S, Dono M, Dell’Aquila ML, Alder H, Rassenti L, Kipps TJ, Bullrich F, Negrini M, Croce CM (August 2004). «MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias». Proc Natl Acad Sci U S A. 101 (32): 11755–60. Bibcode:2004PNAS..10111755C. doi:10.1073/pnas.0404432101. PMC 511048. PMID 15284443.
- ^ Velle, Angelo; Pesenti, Chiara; Grassi, Tommaso; Beltrame, Luca; Martini, Paolo; Jaconi, Marta; Agostinis, Federico; Calura, Enrica; Katsaros, Dionyssios; Borella, Fulvio; Fruscio, Robert; D’Incalci, Maurizio; Marchini, Sergio; Romualdi, Chiara (17 January 2023). «A comprehensive investigation of histotype‐specific microRNA and their variants in Stage I epithelial ovarian cancers». International Journal of Cancer: ijc.34408. doi:10.1002/ijc.34408. ISSN 0020-7136. PMID 36541726. S2CID 255034585.
- ^ Kyriakidis I, Kyriakidis K, Tsezou A (August 2022). «MicroRNAs and the Diagnosis of Childhood Acute Lymphoblastic Leukemia: Systematic Review, Meta-Analysis and Re-Analysis with Novel Small RNA-Seq Tools». Cancers. 14 (16): 3976. doi:10.3390/cancers14163976. PMC 9406077. PMID 36010971.
- ^ Võsa U, Vooder T, Kolde R, Fischer K, Välk K, Tõnisson N, Roosipuu R, Vilo J, Metspalu A, Annilo T (October 2011). «Identification of miR-374a as a prognostic marker for survival in patients with early-stage nonsmall cell lung cancer». Genes, Chromosomes & Cancer. 50 (10): 812–22. doi:10.1002/gcc.20902. PMID 21748820. S2CID 9746594.
- ^ Akçakaya P, Ekelund S, Kolosenko I, Caramuta S, Ozata DM, Xie H, Lindforss U, Olivecrona H, Lui WO (August 2011). «miR-185 and miR-133b deregulation is associated with overall survival and metastasis in colorectal cancer». International Journal of Oncology. 39 (2): 311–8. doi:10.3892/ijo.2011.1043. PMID 21573504.
- ^ a b Eyking A, Reis H, Frank M, Gerken G, Schmid KW, Cario E (2016). «MiR-205 and MiR-373 Are Associated with Aggressive Human Mucinous Colorectal Cancer». PLOS ONE. 11 (6): e0156871. Bibcode:2016PLoSO..1156871E. doi:10.1371/journal.pone.0156871. PMC 4894642. PMID 27271572.
- ^ MicroRNA-21 promotes hepatocellular carcinoma HepG2 cell proliferation through repression of mitogen-activated protein kinase-kinase 3. Guangxian Xu et al., 2013
- ^ Jones K, Nourse JP, Keane C, Bhatnagar A, Gandhi MK (January 2014). «Plasma microRNA are disease response biomarkers in classical Hodgkin lymphoma». Clinical Cancer Research. 20 (1): 253–64. doi:10.1158/1078-0432.CCR-13-1024. PMID 24222179.
- ^ Hosseinahli N, Aghapour M, Duijf PH, Baradaran B (August 2018). «Treating cancer with microRNA replacement therapy: A literature review». Journal of Cellular Physiology. 233 (8): 5574–5588. doi:10.1002/jcp.26514. PMID 29521426. S2CID 3766576.
- ^ Liu G, Sun Y, Ji P, Li X, Cogdell D, Yang D, Parker Kerrigan BC, Shmulevich I, Chen K, Sood AK, Xue F, Zhang W (July 2014). «MiR-506 suppresses proliferation and induces senescence by directly targeting the CDK4/6-FOXM1 axis in ovarian cancer». The Journal of Pathology. 233 (3): 308–18. doi:10.1002/path.4348. PMC 4144705. PMID 24604117.
- ^ Wen SY, Lin Y, Yu YQ, Cao SJ, Zhang R, Yang XM, Li J, Zhang YL, Wang YH, Ma MZ, Sun WW, Lou XL, Wang JH, Teng YC, Zhang ZG (February 2015). «miR-506 acts as a tumor suppressor by directly targeting the hedgehog pathway transcription factor Gli3 in human cervical cancer». Oncogene. 34 (6): 717–25. doi:10.1038/onc.2014.9. PMID 24608427. S2CID 20603801.
- ^ Peking University (13 September 2022). «Scientists Develop a New, Powerful Cancer-Fighting Weapon». Cell. SciTech Daily. 185 (11): 1888–1904.e24. doi:10.1016/j.cell.2022.04.030. PMID 35623329. S2CID 249070106. Retrieved 15 September 2022.
- ^ Loeb KR, Loeb LA (March 2000). «Significance of multiple mutations in cancer». Carcinogenesis. 21 (3): 379–385. doi:10.1093/carcin/21.3.379. PMID 10688858.
- ^ Lodish H, Berk A, Kaiser CA, Krieger M, Bretscher A, Ploegh H, Amon A, Martin KC (2016). Molecular Cell Biology (8th ed.). New York: W. H. Freeman and Company. p. 203. ISBN 978-1-4641-8339-3.
- ^ Hu H, Gatti RA (June 2011). «MicroRNAs: new players in the DNA damage response». Journal of Molecular Cell Biology. 3 (3): 151–158. doi:10.1093/jmcb/mjq042. PMC 3104011. PMID 21183529.
- ^ Jasperson KW, Tuohy TM, Neklason DW, Burt RW (June 2010). «Hereditary and familial colon cancer». Gastroenterology. 138 (6): 2044–58. doi:10.1053/j.gastro.2010.01.054. PMC 3057468. PMID 20420945.
- ^ Truninger K, Menigatti M, Luz J, Russell A, Haider R, Gebbers JO, Bannwart F, Yurtsever H, Neuweiler J, Riehle HM, Cattaruzza MS, Heinimann K, Schär P, Jiricny J, Marra G (May 2005). «Immunohistochemical analysis reveals high frequency of PMS2 defects in colorectal cancer». Gastroenterology. 128 (5): 1160–71. doi:10.1053/j.gastro.2005.01.056. PMID 15887099.
- ^ Valeri N, Gasparini P, Fabbri M, Braconi C, Veronese A, Lovat F, Adair B, Vannini I, Fanini F, Bottoni A, Costinean S, Sandhu SK, Nuovo GJ, Alder H, Gafa R, Calore F, Ferracin M, Lanza G, Volinia S, Negrini M, McIlhatton MA, Amadori D, Fishel R, Croce CM (April 2010). «Modulation of mismatch repair and genomic stability by miR-155». Proceedings of the National Academy of Sciences of the United States of America. 107 (15): 6982–7. Bibcode:2010PNAS..107.6982V. doi:10.1073/pnas.1002472107. PMC 2872463. PMID 20351277.
- ^ a b c Zhang W, Zhang J, Hoadley K, Kushwaha D, Ramakrishnan V, Li S, Kang C, You Y, Jiang C, Song SW, Jiang T, Chen CC (June 2012). «miR-181d: a predictive glioblastoma biomarker that downregulates MGMT expression». Neuro-Oncology. 14 (6): 712–9. doi:10.1093/neuonc/nos089. PMC 3367855. PMID 22570426.
- ^ Spiegl-Kreinecker S, Pirker C, Filipits M, Lötsch D, Buchroithner J, Pichler J, Silye R, Weis S, Micksche M, Fischer J, Berger W (January 2010). «O6-Methylguanine DNA methyltransferase protein expression in tumor cells predicts outcome of temozolomide therapy in glioblastoma patients». Neuro-Oncology. 12 (1): 28–36. doi:10.1093/neuonc/nop003. PMC 2940563. PMID 20150365.
- ^ Sgarra R, Rustighi A, Tessari MA, Di Bernardo J, Altamura S, Fusco A, Manfioletti G, Giancotti V (September 2004). «Nuclear phosphoproteins HMGA and their relationship with chromatin structure and cancer». FEBS Letters. 574 (1–3): 1–8. doi:10.1016/j.febslet.2004.08.013. PMID 15358530. S2CID 28903539.
- ^ Xu Y, Sumter TF, Bhattacharya R, Tesfaye A, Fuchs EJ, Wood LJ, Huso DL, Resar LM (May 2004). «The HMG-I oncogene causes highly penetrant, aggressive lymphoid malignancy in transgenic mice and is overexpressed in human leukemia». Cancer Research. 64 (10): 3371–5. doi:10.1158/0008-5472.CAN-04-0044. PMID 15150086.
- ^ Borrmann L, Schwanbeck R, Heyduk T, Seebeck B, Rogalla P, Bullerdiek J, Wisniewski JR (December 2003). «High mobility group A2 protein and its derivatives bind a specific region of the promoter of DNA repair gene ERCC1 and modulate its activity». Nucleic Acids Research. 31 (23): 6841–51. doi:10.1093/nar/gkg884. PMC 290254. PMID 14627817.
- ^ Facista A, Nguyen H, Lewis C, Prasad AR, Ramsey L, Zaitlin B, Nfonsam V, Krouse RS, Bernstein H, Payne CM, Stern S, Oatman N, Banerjee B, Bernstein C (April 2012). «Deficient expression of DNA repair enzymes in early progression to sporadic colon cancer». Genome Integrity. 3 (1): 3. doi:10.1186/2041-9414-3-3. PMC 3351028. PMID 22494821.
- ^ Gibert B, Delloye-Bourgeois C, Gattolliat CH, Meurette O, Le Guernevel S, Fombonne J, et al. (November 2014). «Regulation by miR181 family of the dependence receptor CDON tumor suppressive activity in neuroblastoma». Journal of the National Cancer Institute. 106 (11). doi:10.1093/jnci/dju318. PMID 25313246.
- ^ Chen JF, Murchison EP, Tang R, Callis TE, Tatsuguchi M, Deng Z, Rojas M, Hammond SM, Schneider MD, Selzman CH, Meissner G, Patterson C, Hannon GJ, Wang DZ (February 2008). «Targeted deletion of Dicer in the heart leads to dilated cardiomyopathy and heart failure». Proceedings of the National Academy of Sciences of the United States of America. 105 (6): 2111–6. Bibcode:2008PNAS..105.2111C. doi:10.1073/pnas.0710228105. PMC 2542870. PMID 18256189.
- ^ a b Zhao Y, Ransom JF, Li A, Vedantham V, von Drehle M, Muth AN, Tsuchihashi T, McManus MT, Schwartz RJ, Srivastava D (April 2007). «Dysregulation of cardiogenesis, cardiac conduction, and cell cycle in mice lacking miRNA-1-2». Cell. 129 (2): 303–17. doi:10.1016/j.cell.2007.03.030. PMID 17397913.
- ^ Thum T, Galuppo P, Wolf C, Fiedler J, Kneitz S, van Laake LW, Doevendans PA, Mummery CL, Borlak J, Haverich A, Gross C, Engelhardt S, Ertl G, Bauersachs J (July 2007). «MicroRNAs in the human heart: a clue to fetal gene reprogramming in heart failure». Circulation. 116 (3): 258–67. doi:10.1161/CIRCULATIONAHA.107.687947. PMID 17606841.
- ^ van Rooij E, Sutherland LB, Liu N, Williams AH, McAnally J, Gerard RD, Richardson JA, Olson EN (November 2006). «A signature pattern of stress-responsive microRNAs that can evoke cardiac hypertrophy and heart failure». Proceedings of the National Academy of Sciences of the United States of America. 103 (48): 18255–60. Bibcode:2006PNAS..10318255V. doi:10.1073/pnas.0608791103. PMC 1838739. PMID 17108080.
- ^ Tatsuguchi M, Seok HY, Callis TE, Thomson JM, Chen JF, Newman M, Rojas M, Hammond SM, Wang DZ (June 2007). «Expression of microRNAs is dynamically regulated during cardiomyocyte hypertrophy». Journal of Molecular and Cellular Cardiology. 42 (6): 1137–41. doi:10.1016/j.yjmcc.2007.04.004. PMC 1934409. PMID 17498736.
- ^ Zhao Y, Samal E, Srivastava D (July 2005). «Serum response factor regulates a muscle-specific microRNA that targets Hand2 during cardiogenesis». Nature. 436 (7048): 214–20. Bibcode:2005Natur.436..214Z. doi:10.1038/nature03817. PMID 15951802. S2CID 4340449.
- ^ Xiao J, Luo X, Lin H, Zhang Y, Lu Y, Wang N, Zhang Y, Yang B, Wang Z (April 2007). «MicroRNA miR-133 represses HERG K+ channel expression contributing to QT prolongation in diabetic hearts». The Journal of Biological Chemistry. 282 (17): 12363–7. doi:10.1074/jbc.C700015200. PMID 17344217.
- ^ Yang B, Lin H, Xiao J, Lu Y, Luo X, Li B, Zhang Y, Xu C, Bai Y, Wang H, Chen G, Wang Z (April 2007). «The muscle-specific microRNA miR-1 regulates cardiac arrhythmogenic potential by targeting GJA1 and KCNJ2». Nature Medicine. 13 (4): 486–91. doi:10.1038/nm1569. PMID 17401374. S2CID 1935811.
- ^ Carè A, Catalucci D, Felicetti F, Bonci D, Addario A, Gallo P, Bang ML, Segnalini P, Gu Y, Dalton ND, Elia L, Latronico MV, Høydal M, Autore C, Russo MA, Dorn GW, Ellingsen O, Ruiz-Lozano P, Peterson KL, Croce CM, Peschle C, Condorelli G (May 2007). «MicroRNA-133 controls cardiac hypertrophy». Nature Medicine. 13 (5): 613–8. doi:10.1038/nm1582. PMID 17468766. S2CID 10097893.
- ^ van Rooij E, Sutherland LB, Qi X, Richardson JA, Hill J, Olson EN (April 2007). «Control of stress-dependent cardiac growth and gene expression by a microRNA». Science. 316 (5824): 575–9. Bibcode:2007Sci…316..575V. doi:10.1126/science.1139089. PMID 17379774. S2CID 1927839.
- ^ Keller T, Boeckel JN, Groß S, Klotsche J, Palapies L, Leistner D, et al. (July 2017). «Improved risk stratification in prevention by use of a panel of selected circulating microRNAs». Scientific Reports. 7 (1): 4511. Bibcode:2017NatSR…7.4511K. doi:10.1038/s41598-017-04040-w. PMC 5495799. PMID 28674420.
- ^ Insull W (January 2009). «The pathology of atherosclerosis: plaque development and plaque responses to medical treatment». The American Journal of Medicine. 122 (1 Suppl): S3–S14. doi:10.1016/j.amjmed.2008.10.013. PMID 19110086.
- ^ a b c d e f g h i j k l m n o p q Son DJ, Kumar S, Takabe W, Kim CW, Ni CW, Alberts-Grill N, Jang IH, Kim S, Kim W, Won Kang S, Baker AH, Woong Seo J, Ferrara KW, Jo H (2013). «The atypical mechanosensitive microRNA-712 derived from pre-ribosomal RNA induces endothelial inflammation and atherosclerosis». Nature Communications. 4: 3000. Bibcode:2013NatCo…4.3000S. doi:10.1038/ncomms4000. PMC 3923891. PMID 24346612.
- ^ a b Basu R, Fan D, Kandalam V, Lee J, Das SK, Wang X, Baldwin TA, Oudit GY, Kassiri Z (December 2012). «Loss of Timp3 gene leads to abdominal aortic aneurysm formation in response to angiotensin II». The Journal of Biological Chemistry. 287 (53): 44083–96. doi:10.1074/jbc.M112.425652. PMC 3531724. PMID 23144462.
- ^ Libby P (2002). «Inflammation in atherosclerosis». Nature. 420 (6917): 868–74. Bibcode:2002Natur.420..868L. doi:10.1038/nature01323. PMID 12490960. S2CID 407449.
- ^ a b Phua YL, Chu JY, Marrone AK, Bodnar AJ, Sims-Lucas S, Ho J (October 2015). «Renal stromal miRNAs are required for normal nephrogenesis and glomerular mesangial survival». Physiological Reports. 3 (10): e12537. doi:10.14814/phy2.12537. PMC 4632944. PMID 26438731.
- ^ Maes OC, Chertkow HM, Wang E, Schipper HM (May 2009). «MicroRNA: Implications for Alzheimer Disease and other Human CNS Disorders». Current Genomics. 10 (3): 154–68. doi:10.2174/138920209788185252. PMC 2705849. PMID 19881909.
- ^ Amin ND, Bai G, Klug JR, Bonanomi D, Pankratz MT, Gifford WD, Hinckley CA, Sternfeld MJ, Driscoll SP, Dominguez B, Lee KF, Jin X, Pfaff SL (December 2015). «Loss of motoneuron-specific microRNA-218 causes systemic neuromuscular failure». Science. 350 (6267): 1525–9. Bibcode:2015Sci…350.1525A. doi:10.1126/science.aad2509. PMC 4913787. PMID 26680198.
- ^ Schratt G (December 2009). «microRNAs at the synapse». Nature Reviews. Neuroscience. 10 (12): 842–9. doi:10.1038/nrn2763. PMID 19888283. S2CID 3507952.
- ^ Cuellar TL, Davis TH, Nelson PT, Loeb GB, Harfe BD, Ullian E, McManus MT (April 2008). «Dicer loss in striatal neurons produces behavioral and neuroanatomical phenotypes in the absence of neurodegeneration». Proceedings of the National Academy of Sciences. 105 (14): 5614–19. Bibcode:2008PNAS..105.5614C. doi:10.1073/pnas.0801689105. PMC 2291142. PMID 18385371.
- ^ Hébert SS, Papadopoulou AS, Smith P, Galas M, Planel E, Silahtaroglu AN, Sergeant N, Buée L, De Strooper B (October 2010). «Genetic ablation of Dicer in adult forebrain neurons results in abnormal tau hyperphosphorylation and neurodegeneration». Human Molecular Genetics. 19 (20): 3959–69. doi:10.1093/hmg/ddq311. PMID 20660113.
- ^ Hosseinian S, Arefian A, Rakhsh-Khorshid H (2020). «A meta-analysis of gene expression data highlights synaptic dysfunction in the hippocampus of brains with Alzheimer’s disease». Scientific Reports. 10 (1): 8384. Bibcode:2020NatSR..10.8384H. doi:10.1038/s41598-020-64452-z. PMC 7239885. PMID 32433480.
- ^ Hommers LG, Domschke K, Deckert J (January 2015). «Heterogeneity and individuality: microRNAs in mental disorders». Journal of Neural Transmission. 122 (1): 79–97. doi:10.1007/s00702-014-1338-4. PMID 25395183. S2CID 25088900.
- ^ Feng J, Sun G, Yan J, Noltner K, Li W, Buzin CH, Longmate J, Heston LL, Rossi J, Sommer SS (July 2009). Reif A (ed.). «Evidence for X-chromosomal schizophrenia associated with microRNA alterations». PLOS ONE. 4 (7): e6121. Bibcode:2009PLoSO…4.6121F. doi:10.1371/journal.pone.0006121. PMC 2699475. PMID 19568434.
- ^ Beveridge NJ, Gardiner E, Carroll AP, Tooney PA, Cairns MJ (December 2010). «Schizophrenia is associated with an increase in cortical microRNA biogenesis». Molecular Psychiatry. 15 (12): 1176–89. doi:10.1038/mp.2009.84. PMC 2990188. PMID 19721432.
- ^ «Stroke Facts | cdc.gov». www.cdc.gov. 15 March 2019. Retrieved 5 December 2019.
- ^ Rink C, Khanna S (May 2011). «MicroRNA in ischemic stroke etiology and pathology». Physiological Genomics. 43 (10): 521–528. doi:10.1152/physiolgenomics.00158.2010. PMC 3110894. PMID 20841499.
- ^ Ouyang YB, Stary CM, Yang GY, Giffard R (January 2013). «microRNAs: innovative targets for cerebral ischemia and stroke». Current Drug Targets. 14 (1): 90–101. doi:10.2174/138945013804806424. PMC 3673881. PMID 23170800.
- ^ a b c Lewohl JM, Nunez YO, Dodd PR, Tiwari GR, Harris RA, Mayfield RD (November 2011). «Up-regulation of microRNAs in brain of human alcoholics». Alcoholism: Clinical and Experimental Research. 35 (11): 1928–37. doi:10.1111/j.1530-0277.2011.01544.x. PMC 3170679. PMID 21651580.
- ^ Tapocik JD, Solomon M, Flanigan M, Meinhardt M, Barbier E, Schank JR, Schwandt M, Sommer WH, Heilig M (June 2013). «Coordinated dysregulation of mRNAs and microRNAs in the rat medial prefrontal cortex following a history of alcohol dependence». The Pharmacogenomics Journal. 13 (3): 286–96. doi:10.1038/tpj.2012.17. PMC 3546132. PMID 22614244.
- ^ Gorini G, Nunez YO, Mayfield RD (2013). «Integration of miRNA and protein profiling reveals coordinated neuroadaptations in the alcohol-dependent mouse brain». PLOS ONE. 8 (12): e82565. Bibcode:2013PLoSO…882565G. doi:10.1371/journal.pone.0082565. PMC 3865091. PMID 24358208.
- ^ Tapocik JD, Barbier E, Flanigan M, Solomon M, Pincus A, Pilling A, Sun H, Schank JR, King C, Heilig M (March 2014). «microRNA-206 in rat medial prefrontal cortex regulates BDNF expression and alcohol drinking». The Journal of Neuroscience. 34 (13): 4581–8. doi:10.1523/JNEUROSCI.0445-14.2014. PMC 3965783. PMID 24672003.
- ^ Lippai D, Bala S, Csak T, Kurt-Jones EA, Szabo G (2013). «Chronic alcohol-induced microRNA-155 contributes to neuroinflammation in a TLR4-dependent manner in mice». PLOS ONE. 8 (8): e70945. Bibcode:2013PLoSO…870945L. doi:10.1371/journal.pone.0070945. PMC 3739772. PMID 23951048.
- ^ a b Li J, Li J, Liu X, Qin S, Guan Y, Liu Y, Cheng Y, Chen X, Li W, Wang S, Xiong M, Kuzhikandathil EV, Ye JH, Zhang C (September 2013). «MicroRNA expression profile and functional analysis reveal that miR-382 is a critical novel gene of alcohol addiction». EMBO Molecular Medicine. 5 (9): 1402–14. doi:10.1002/emmm.201201900. PMC 3799494. PMID 23873704.
- ^ Romao JM, Jin W, Dodson MV, Hausman GJ, Moore SS, Guan LL (September 2011). «MicroRNA regulation in mammalian adipogenesis». Experimental Biology and Medicine. 236 (9): 997–1004. doi:10.1258/ebm.2011.011101. PMID 21844119. S2CID 30646787.
- ^ Skårn M, Namløs HM, Noordhuis P, Wang MY, Meza-Zepeda LA, Myklebost O (April 2012). «Adipocyte differentiation of human bone marrow-derived stromal cells is modulated by microRNA-155, microRNA-221, and microRNA-222». Stem Cells and Development. 21 (6): 873–83. doi:10.1089/scd.2010.0503. hdl:10852/40423. PMID 21756067.
- ^ Zuo Y, Qiang L, Farmer SR (March 2006). «Activation of CCAAT/enhancer-binding protein (C/EBP) alpha expression by C/EBP beta during adipogenesis requires a peroxisome proliferator-activated receptor-gamma-associated repression of HDAC1 at the C/ebp alpha gene promoter». The Journal of Biological Chemistry. 281 (12): 7960–7. doi:10.1074/jbc.M510682200. PMID 16431920.
- ^ Jun-Hao ET, Gupta RR, Shyh-Chang N (March 2016). «Lin28 and let-7 in the Metabolic Physiology of Aging». Trends in Endocrinology and Metabolism. 27 (3): 132–141. doi:10.1016/j.tem.2015.12.006. PMID 26811207. S2CID 3614126.
- ^ Zhu H, Shyh-Chang N, Segrè AV, Shinoda G, Shah SP, Einhorn WS, Takeuchi A, Engreitz JM, Hagan JP, Kharas MG, Urbach A, Thornton JE, Triboulet R, Gregory RI, Altshuler D, Daley GQ (September 2011). «The Lin28/let-7 axis regulates glucose metabolism». Cell. 147 (1): 81–94. doi:10.1016/j.cell.2011.08.033. PMC 3353524. PMID 21962509.
- ^ Frost RJ, Olson EN (December 2011). «Control of glucose homeostasis and insulin sensitivity by the Let-7 family of microRNAs». Proceedings of the National Academy of Sciences of the United States of America. 108 (52): 21075–80. Bibcode:2011PNAS..10821075F. doi:10.1073/pnas.1118922109. PMC 3248488. PMID 22160727.
- ^ Teruel-Montoya R, Rosendaal FR, Martínez C (February 2015). «MicroRNAs in hemostasis». Journal of Thrombosis and Haemostasis. 13 (2): 170–181. doi:10.1111/jth.12788. PMID 25400249.
- ^ a b Nourse J, Braun J, Lackner K, Hüttelmaier S, Danckwardt S (November 2018). «Large-scale identification of functional microRNA targeting reveals cooperative regulation of the hemostatic system». Journal of Thrombosis and Haemostasis. 16 (11): 2233–2245. doi:10.1111/jth.14290. PMID 30207063.
- ^ Nourse J, Danckwardt S (February 2021). «A novel rationale for targeting FXI: Insights from the hemostatic microRNA targetome for emerging anticoagulant strategies». Pharmacology & Therapeutics. 218: 107676. doi:10.1016/j.pharmthera.2020.107676. PMID 32898547.
- ^ Berardi E, Pues M, Thorrez L, Sampaolesi M (October 2012). «miRNAs in ESC differentiation». American Journal of Physiology. Heart and Circulatory Physiology. 303 (8): H931–H939. doi:10.1152/ajpheart.00338.2012. PMID 22886416. S2CID 6402014.
- ^ a b Borchers A, Pieler T (November 2010). «Programming pluripotent precursor cells derived from Xenopus embryos to generate specific tissues and organs». Genes. 1 (3): 413–426. doi:10.3390/ijms232314755. PMC 9740008. PMID 36499082.
- ^ Curaba J, Spriggs A, Taylor J, Li Z, Helliwell C (July 2012). «miRNA regulation in the early development of barley seed». BMC Plant Biology. 12 (1): 120. doi:10.1186/1471-2229-12-120. PMC 3443071. PMID 22838835.
- ^ Choudhary A, Kumar A, Kaur H, Kaur N (May 2021). «MiRNA: the taskmaster of plant world». Biologia. 76 (5): 1551–1567. doi:10.1007/s11756-021-00720-1. ISSN 1336-9563. S2CID 233685660.
- ^ Waheed S, Zeng L (March 2020). «The Critical Role of miRNAs in Regulation of Flowering Time and Flower Development». Genes. 11 (3): 319. doi:10.3390/genes11030319. PMC 7140873. PMID 32192095.
- ^ Wong CE, Zhao YT, Wang XJ, Croft L, Wang ZH, Haerizadeh F, et al. (May 2011). «MicroRNAs in the shoot apical meristem of soybean». Journal of Experimental Botany. 62 (8): 2495–2506. doi:10.1093/jxb/erq437. hdl:10536/DRO/DU:30106047. PMID 21504877.
- ^ Qureshi A, Thakur N, Monga I, Thakur A, Kumar M (1 January 2014). «VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets». Database. 2014: bau103. doi:10.1093/database/bau103. PMC 4224276. PMID 25380780.
- ^ Kumar M. «VIRmiRNA». Resource for experimental viral miRNA and their targets. Bioinformatics center, CSIR-IMTECH.
- ^ Tuddenham L, Jung JS, Chane-Woon-Ming B, Dölken L, Pfeffer S (February 2012). «Small RNA deep sequencing identifies microRNAs and other small noncoding RNAs from human herpesvirus 6B». Journal of Virology. 86 (3): 1638–49. doi:10.1128/JVI.05911-11. PMC 3264354. PMID 22114334.
- ^ Zheng H, Fu R, Wang JT, Liu Q, Chen H, Jiang SW (April 2013). «Advances in the techniques for the prediction of microRNA targets». International Journal of Molecular Sciences. 14 (4): 8179–87. doi:10.3390/ijms14048179. PMC 3645737. PMID 23591837.
- ^ Agarwal V, Bell GW, Nam JW, Bartel DP (August 2015). «Predicting effective microRNA target sites in mammalian mRNAs». eLife. 4: e05005. doi:10.7554/eLife.05005. PMC 4532895. PMID 26267216.
Further reading[edit]
- miRNA definition and classification: Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T (March 2003). «A uniform system for microRNA annotation». RNA. 9 (3): 277–9. doi:10.1261/rna.2183803. PMC 1370393. PMID 12592000.
- Science review of small RNA: Baulcombe D (September 2002). «DNA events. An RNA microcosm». Science. 297 (5589): 2002–3. doi:10.1126/science.1077906. PMID 12242426. S2CID 82531727.
- Discovery of lin-4, the first miRNA to be discovered: Lee RC, Feinbaum RL, Ambros V (December 1993). «The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14». Cell. 75 (5): 843–54. doi:10.1016/0092-8674(93)90529-Y. PMID 8252621.
External links[edit]
- The miRBase database
- miRTarBase, the experimentally validated microRNA-target interactions database.
- semirna, Web application to search for microRNAs in a plant genome.
- ONCO.IO: Integrative resource for microRNA and transcription factors analysis in cancer.
- MirOB: MicroRNA targets database and data analysis and dataviz tool.
- ChIPBase database: An open access database for decoding the transcription factors that were involved in or affected the transcription of microRNAs from ChIP-seq data.
- An animated video of the microRNA biogenesis process.
- miRNA modulation reagents to enable up-regulation or suppression of endogenous mature microRNA function
Pre-miRNA instead of Pri-miRNA in the first point of mechanism. Diagram of microRNA (miRNA) action with mRNA
Examples of miRNA stem-loops, with the mature miRNAs shown in red
MicroRNA (miRNA) are small, single-stranded, non-coding RNA molecules containing 21 to 23 nucleotides.[1] Found in plants, animals and some viruses, miRNAs are involved in RNA silencing and post-transcriptional regulation of gene expression.[2][3] miRNAs base-pair to complementary sequences in mRNA molecules,[4] then gene silence said mRNA molecules by one or more of the following processes: (1) cleavage of mRNA strand into two pieces, (2) destabilization of mRNA by shortening its poly(A) tail, or (3) translation of mRNA into proteins. This last method of gene silencing is the least efficient of the three, and requires the aid of ribosomes.[4][5]
miRNAs resemble the small interfering RNAs (siRNAs) of the RNA interference (RNAi) pathway, except miRNAs derive from regions of RNA transcripts that fold back on themselves to form short hairpins, whereas siRNAs derive from longer regions of double-stranded RNA.[6] The human genome may encode over 1900 miRNAs,[7] although more recent analysis suggests that the number is closer to 2,300.[8] However, only about 500 human microRNAs represent bona fide miRNA in the manually curated miRNA gene database MirGeneDB.[9]
miRNAs are abundant in many mammalian cell types[10][11] and as extracellular circulating miRNAs.[12] Circulating miRNAs are released into body fluids including blood and cerebrospinal fluid and have the potential to be available as biomarkers in a number of diseases.[12][13] MiRNAs appear to target about 60% of the genes of humans and other mammals.[14][15] Many miRNAs are evolutionarily conserved, which implies that they have important biological functions.[16][2] For example, 90 families of miRNAs have been conserved since at least the common ancestor of mammals and fish, and most of these conserved miRNAs have important functions, as shown by studies in which genes for one or more members of a family have been knocked out in mice.[2]
History[edit]
The first miRNA was discovered in the early 1990s.[17] However, miRNAs were not recognized as a distinct class of biological regulators until the early 2000s.[18][19][20][21][22] miRNA research revealed different sets of miRNAs expressed in different cell types and tissues[11][23] and multiple roles for miRNAs in plant and animal development and in many other biological processes.[24][25][26][27][28][29][30] Aberrant miRNA expression are implicated in disease states. MiRNA-based therapies are under investigation.[31][32][33][34]
The first miRNA was discovered in 1993 by a group led by Ambros and including Lee and Feinbaum. However, additional insight into its mode of action required simultaneously published work by Ruvkun’s team, including Wightman and Ha.[17][35] These groups published back-to-back papers on the lin-4 gene, which was known to control the timing of C. elegans larval development by repressing the lin-14 gene. When Lee et al. isolated the lin-4 miRNA, they found that instead of producing an mRNA encoding a protein, it produced short non-coding RNAs, one of which was a ~22-nucleotide RNA that contained sequences partially complementary to multiple sequences in the 3′ UTR of the lin-14 mRNA.[17] This complementarity was proposed to inhibit the translation of the lin-14 mRNA into the LIN-14 protein. At the time, the lin-4 small RNA was thought to be a nematode idiosyncrasy.
In 2000, a second small RNA was characterized: let-7 RNA, which represses lin-41 to promote a later developmental transition in C. elegans.[18] The let-7 RNA was found to be conserved in many species, leading to the suggestion that let-7 RNA and additional «small temporal RNAs» might regulate the timing of development in diverse animals, including humans.[19]
A year later, the lin-4 and let-7 RNAs were found to be part of a large class of small RNAs present in C. elegans, Drosophila and human cells.[20][21][22] The many RNAs of this class resembled the lin-4 and let-7 RNAs, except their expression patterns were usually inconsistent with a role in regulating the timing of development. This suggested that most might function in other types of regulatory pathways. At this point, researchers started using the term «microRNA» to refer to this class of small regulatory RNAs.[20][21][22]
The first human disease associated with deregulation of miRNAs was chronic lymphocytic leukemia. In this disorder, the miRNAs have a dual role working as both tumor suppressors and oncogenes.[36]
Nomenclature[edit]
Under a standard nomenclature system, names are assigned to experimentally confirmed miRNAs before publication.[37][38] The prefix «miR» is followed by a dash and a number, the latter often indicating order of naming. For example, miR-124 was named and likely discovered prior to miR-456. A capitalized «miR-» refers to the mature form of the miRNA, while the uncapitalized «mir-» refers to the pre-miRNA and the pri-miRNA.[39] The miRNAs encoding genes are also named using the same three-letter prefix according to the conventions of the organism gene nomenclature. For examples, the official miRNAs gene names in some organisms are «mir-1 in C. elegans and Drosophila, Mir-1 in Rattus norvegicus and MIR-25 in human.
miRNAs with nearly identical sequences except for one or two nucleotides are annotated with an additional lower case letter. For example, miR-124a is closely related to miR-124b. For example:
- hsa-miR-181a: aacauucaACgcugucggugAgu
- hsa-miR-181b: aacauucaUUgcugucggugGgu
Pre-miRNAs, pri-miRNAs and genes that lead to 100% identical mature miRNAs but that are located at different places in the genome are indicated with an additional dash-number suffix. For example, the pre-miRNAs hsa-mir-194-1 and hsa-mir-194-2 lead to an identical mature miRNA (hsa-miR-194) but are from genes located in different genome regions.
Species of origin is designated with a three-letter prefix, e.g., hsa-miR-124 is a human (Homo sapiens) miRNA and oar-miR-124 is a sheep (Ovis aries) miRNA. Other common prefixes include «v» for viral (miRNA encoded by a viral genome) and «d» for Drosophila miRNA (a fruit fly commonly studied in genetic research).
When two mature microRNAs originate from opposite arms of the same pre-miRNA and are found in roughly similar amounts, they are denoted with a -3p or -5p suffix. (In the past, this distinction was also made with «s» (sense) and «as» (antisense)). However, the mature microRNA found from one arm of the hairpin is usually much more abundant than that found from the other arm,[6] in which case, an asterisk following the name indicates the mature species found at low levels from the opposite arm of a hairpin. For example, miR-124 and miR-124* share a pre-miRNA hairpin, but much more miR-124 is found in the cell.
Targets[edit]
Plant miRNAs usually have near-perfect pairing with their mRNA targets, which induces gene repression through cleavage of the target transcripts.[24][40] In contrast, animal miRNAs are able to recognize their target mRNAs by using as few as 6–8 nucleotides (the seed region) at the 5′ end of the miRNA,[14][41][42] which is not enough pairing to induce cleavage of the target mRNAs.[4] Combinatorial regulation is a feature of miRNA regulation in animals.[4][43] A given miRNA may have hundreds of different mRNA targets, and a given target might be regulated by multiple miRNAs.[15][44]
Estimates of the average number of unique messenger RNAs that are targets for repression by a typical miRNA vary, depending on the estimation method,[45] but multiple approaches show that mammalian miRNAs can have many unique targets. For example, an analysis of the miRNAs highly conserved in vertebrates shows that each has, on average, roughly 400 conserved targets.[15] Likewise, experiments show that a single miRNA species can reduce the stability of hundreds of unique messenger RNAs.[46] Other experiments show that a single miRNA species may repress the production of hundreds of proteins, but that this repression often is relatively mild (much less than 2-fold).[47][48] The first human disease discovered to be associated with deregulation of miRNAs was chronic lymphocytic leukemia. Other B cell malignancies followed.
Further information on the Poly(A)-binding protein: Paip2b
Biogenesis[edit]
As many as 40% of miRNA genes may lie in the introns or even exons of other genes.[49] These are usually, though not exclusively, found in a sense orientation,[50][51] and thus usually are regulated together with their host genes.[49][52][53]
The DNA template is not the final word on mature miRNA production: 6% of human miRNAs show RNA editing (IsomiRs), the site-specific modification of RNA sequences to yield products different from those encoded by their DNA. This increases the diversity and scope of miRNA action beyond that implicated from the genome alone.
Transcription[edit]
miRNA genes are usually transcribed by RNA polymerase II (Pol II).[54][55] The polymerase often binds to a promoter found near the DNA sequence, encoding what will become the hairpin loop of the pre-miRNA. The resulting transcript is capped with a specially modified nucleotide at the 5′ end, polyadenylated with multiple adenosines (a poly(A) tail),[54][50] and spliced. Animal miRNAs are initially transcribed as part of one arm of an ∼80 nucleotide RNA stem-loop that in turn forms part of a several hundred nucleotide-long miRNA precursor termed a pri-miRNA.[54][50] When a stem-loop precursor is found in the 3′ UTR, a transcript may serve as a pri-miRNA and a mRNA.[50] RNA polymerase III (Pol III) transcribes some miRNAs, especially those with upstream Alu sequences, transfer RNAs (tRNAs), and mammalian wide interspersed repeat (MWIR) promoter units.[56]
Nuclear processing[edit]
A single pri-miRNA may contain from one to six miRNA precursors. These hairpin loop structures are composed of about 70 nucleotides each. Each hairpin is flanked by sequences necessary for efficient processing.
The double-stranded RNA (dsRNA) structure of the hairpins in a pri-miRNA is recognized by a nuclear protein known as DiGeorge Syndrome Critical Region 8 (DGCR8 or «Pasha» in invertebrates), named for its association with DiGeorge Syndrome. DGCR8 associates with the enzyme Drosha, a protein that cuts RNA, to form the Microprocessor complex.[57][58] In this complex, DGCR8 orients the catalytic RNase III domain of Drosha to liberate hairpins from pri-miRNAs by cleaving RNA about eleven nucleotides from the hairpin base (one helical dsRNA turn into the stem).[59][60] The product resulting has a two-nucleotide overhang at its 3′ end; it has 3′ hydroxyl and 5′ phosphate groups. It is often termed as a pre-miRNA (precursor-miRNA). Sequence motifs downstream of the pre-miRNA that are important for efficient processing have been identified.[61][62][63]
Pre-miRNAs that are spliced directly out of introns, bypassing the Microprocessor complex, are known as «Mirtrons.» Originally thought to exist only in Drosophila and C. elegans, mirtrons have now been found in mammals.[64]
As many as 16% of pre-miRNAs may be altered through nuclear RNA editing.[65][66][67] Most commonly, enzymes known as adenosine deaminases acting on RNA (ADARs) catalyze adenosine to inosine (A to I) transitions. RNA editing can halt nuclear processing (for example, of pri-miR-142, leading to degradation by the ribonuclease Tudor-SN) and alter downstream processes including cytoplasmic miRNA processing and target specificity (e.g., by changing the seed region of miR-376 in the central nervous system).[65]
Nuclear export[edit]
The human exportin-5 protein (red) in complex with Ran-GTP (yellow) and a pre-microRNA (green), showing two-nucleotide overhang recognition element (orange). From PDB: 3A6P.
Pre-miRNA hairpins are exported from the nucleus in a process involving the nucleocytoplasmic shuttler Exportin-5. This protein, a member of the karyopherin family, recognizes a two-nucleotide overhang left by the RNase III enzyme Drosha at the 3′ end of the pre-miRNA hairpin. Exportin-5-mediated transport to the cytoplasm is energy-dependent, using guanosine triphosphate (GTP) bound to the Ran protein.[68]
Cytoplasmic processing[edit]
In the cytoplasm, the pre-miRNA hairpin is cleaved by the RNase III enzyme Dicer.[69] This endoribonuclease interacts with 5′ and 3′ ends of the hairpin[70] and cuts away the loop joining the 3′ and 5′ arms, yielding an imperfect miRNA:miRNA* duplex about 22 nucleotides in length.[69] Overall hairpin length and loop size influence the efficiency of Dicer processing. The imperfect nature of the miRNA:miRNA* pairing also affects cleavage.[69][71] Some of the G-rich pre-miRNAs can potentially adopt the G-quadruplex structure as an alternative to the canonical stem-loop structure. For example, human pre-miRNA 92b adopts a G-quadruplex structure which is resistant to the Dicer mediated cleavage in the cytoplasm.[72] Although either strand of the duplex may potentially act as a functional miRNA, only one strand is usually incorporated into the RNA-induced silencing complex (RISC) where the miRNA and its mRNA target interact.
While the majority of miRNAs are located within the cell, some miRNAs, commonly known as circulating miRNAs or extracellular miRNAs, have also been found in extracellular environment, including various biological fluids and cell culture media.[73][74]
Biogenesis in plants[edit]
miRNA biogenesis in plants differs from animal biogenesis mainly in the steps of nuclear processing and export. Instead of being cleaved by two different enzymes, once inside and once outside the nucleus, both cleavages of the plant miRNA are performed by a Dicer homolog, called Dicer-like1 (DL1). DL1 is expressed only in the nucleus of plant cells, which indicates that both reactions take place inside the nucleus. Before plant miRNA:miRNA* duplexes are transported out of the nucleus, its 3′ overhangs are methylated by a RNA methyltransferaseprotein called Hua-Enhancer1 (HEN1). The duplex is then transported out of the nucleus to the cytoplasm by a protein called Hasty (HST), an Exportin 5 homolog, where they disassemble and the mature miRNA is incorporated into the RISC.[75]
RNA-induced silencing complex[edit]
The mature miRNA is part of an active RNA-induced silencing complex (RISC) containing Dicer and many associated proteins.[76] RISC is also known as a microRNA ribonucleoprotein complex (miRNP);[77] A RISC with incorporated miRNA is sometimes referred to as a «miRISC.»
Dicer processing of the pre-miRNA is thought to be coupled with unwinding of the duplex. Generally, only one strand is incorporated into the miRISC, selected on the basis of its thermodynamic instability and weaker base-pairing on the 5′ end relative to the other strand.[78][79][80] The position of the stem-loop may also influence strand choice.[81] The other strand, called the passenger strand due to its lower levels in the steady state, is denoted with an asterisk (*) and is normally degraded. In some cases, both strands of the duplex are viable and become functional miRNA that target different mRNA populations.[82]
AGO2 (grey) in complex with a microRNA (light blue) and its target mRNA (dark blue)
Members of the Argonaute (Ago) protein family are central to RISC function. Argonautes are needed for miRNA-induced silencing and contain two conserved RNA binding domains: a PAZ domain that can bind the single stranded 3′ end of the mature miRNA and a PIWI domain that structurally resembles ribonuclease-H and functions to interact with the 5′ end of the guide strand. They bind the mature miRNA and orient it for interaction with a target mRNA. Some argonautes, for example human Ago2, cleave target transcripts directly; argonautes may also recruit additional proteins to achieve translational repression.[83] The human genome encodes eight argonaute proteins divided by sequence similarities into two families: AGO (with four members present in all mammalian cells and called E1F2C/hAgo in humans), and PIWI (found in the germ line and hematopoietic stem cells).[77][83]
Additional RISC components include TRBP [human immunodeficiency virus (HIV) transactivating response RNA (TAR) binding protein],[84] PACT (protein activator of the interferon-induced protein kinase), the SMN complex, fragile X mental retardation protein (FMRP), Tudor staphylococcal nuclease-domain-containing protein (Tudor-SN), the putative DNA helicase MOV10, and the RNA recognition motif containing protein TNRC6B.[68][85][86]
Mode of silencing and regulatory loops[edit]
Gene silencing may occur either via mRNA degradation or preventing mRNA from being translated. For example, miR16 contains a sequence complementary to the AU-rich element found in the 3’UTR of many unstable mRNAs, such as TNF alpha or GM-CSF.[87] It has been demonstrated that given complete complementarity between the miRNA and target mRNA sequence, Ago2 can cleave the mRNA and lead to direct mRNA degradation. In the absence of complementarity, silencing is achieved by preventing translation.[46] The relation of miRNA and its target mRNA can be based on the simple negative regulation of a target mRNA, but it seems that a common scenario is the use of a «coherent feed-forward loop», «mutual negative feedback loop» (also termed double negative loop) and «positive feedback/feed-forward loop». Some miRNAs work as buffers of random gene expression changes arising due to stochastic events in transcription, translation and protein stability. Such regulation is typically achieved by the virtue of negative feedback loops or incoherent feed-forward loop uncoupling protein output from mRNA transcription.
Turnover[edit]
Turnover of mature miRNA is needed for rapid changes in miRNA expression profiles. During miRNA maturation in the cytoplasm, uptake by the Argonaute protein is thought to stabilize the guide strand, while the opposite (* or «passenger») strand is preferentially destroyed. In what has been called a «Use it or lose it» strategy, Argonaute may preferentially retain miRNAs with many targets over miRNAs with few or no targets, leading to degradation of the non-targeting molecules.[88]
Decay of mature miRNAs in Caenorhabditis elegans is mediated by the 5′-to-3′ exoribonuclease XRN2, also known as Rat1p.[89] In plants, SDN (small RNA degrading nuclease) family members degrade miRNAs in the opposite (3′-to-5′) direction. Similar enzymes are encoded in animal genomes, but their roles have not been described.[88]
Several miRNA modifications affect miRNA stability. As indicated by work in the model organism Arabidopsis thaliana (thale cress), mature plant miRNAs appear to be stabilized by the addition of methyl moieties at the 3′ end. The 2′-O-conjugated methyl groups block the addition of uracil (U) residues by uridyltransferase enzymes, a modification that may be associated with miRNA degradation. However, uridylation may also protect some miRNAs; the consequences of this modification are incompletely understood. Uridylation of some animal miRNAs has been reported. Both plant and animal miRNAs may be altered by addition of adenine (A) residues to the 3′ end of the miRNA. An extra A added to the end of mammalian miR-122, a liver-enriched miRNA important in hepatitis C, stabilizes the molecule and plant miRNAs ending with an adenine residue have slower decay rates.[88]
Cellular functions[edit]
Interaction of microRNA with protein translation process. Several translation repression mechanisms are shown: M1) on the initiation process, preventing assembling of the initiation complex or recruiting the 40S ribosomal subunit; M2) on the ribosome assembly; M3) on the translation process; M7, M8) on the degradation of mRNA.[90] 40S and 60S are light and heavy components of the ribosome, 80S is the assembled ribosome bound to mRNA, eIF4F is a translation initiation factor, PABC1 is the Poly-A binding protein, and «cap» is the mRNA cap structure needed for mRNA circularization (which can be the normal m7G-cap or modified A-cap). The initiation of mRNA can proceed in a cap-independent manner, through recruiting 40S to IRES (Internal Ribosome Entry Site) located in 5’UTR region. The actual work of RNA silencing is performed by RISC in which the main catalytic subunit is one of the Argonaute proteins (AGO), and miRNA serves as a template for recognizing specific mRNA sequences.
The function of miRNAs appears to be in gene regulation. For that purpose, a miRNA is complementary to a part of one or more messenger RNAs (mRNAs). Animal miRNAs are usually complementary to a site in the 3′ UTR whereas plant miRNAs are usually complementary to coding regions of mRNAs.[91] Perfect or near perfect base pairing with the target RNA promotes cleavage of the RNA.[92] This is the primary mode of plant miRNAs.[93] In animals the match-ups are imperfect.
For partially complementary microRNAs to recognise their targets, nucleotides 2–7 of the miRNA (its ‘seed region’[14][41]) must be perfectly complementary.[94] Animal miRNAs inhibit protein translation of the target mRNA[95] (this is present but less common in plants).[93] Partially complementary microRNAs can also speed up deadenylation, causing mRNAs to be degraded sooner.[96] While degradation of miRNA-targeted mRNA is well documented, whether or not translational repression is accomplished through mRNA degradation, translational inhibition, or a combination of the two is hotly debated. Recent work on miR-430 in zebrafish, as well as on bantam-miRNA and miR-9 in Drosophila cultured cells, shows that translational repression is caused by the disruption of translation initiation, independent of mRNA deadenylation.[97][98]
miRNAs occasionally also cause histone modification and DNA methylation of promoter sites, which affects the expression of target genes.[99][100]
Nine mechanisms of miRNA action are described and assembled in a unified mathematical model:[90]
- Cap-40S initiation inhibition;
- 60S Ribosomal unit joining inhibition;
- Elongation inhibition;
- Ribosome drop-off (premature termination);
- Co-translational nascent protein degradation;
- Sequestration in P-bodies;
- mRNA decay (destabilisation);
- mRNA cleavage;
- Transcriptional inhibition through microRNA-mediated chromatin reorganization followed by gene silencing.
It is often impossible to discern these mechanisms using experimental data about stationary reaction rates. Nevertheless, they are differentiated in dynamics and have different kinetic signatures.[90]
Unlike plant microRNAs, the animal microRNAs target diverse genes.[41] However, genes involved in functions common to all cells, such as gene expression, have relatively fewer microRNA target sites and seem to be under selection to avoid targeting by microRNAs.[101] There is a strong correlation between ITPR gene regulations and mir-92 and mir-19.[102]
dsRNA can also activate gene expression, a mechanism that has been termed «small RNA-induced gene activation» or RNAa. dsRNAs targeting gene promoters can induce potent transcriptional activation of associated genes. This was demonstrated in human cells using synthetic dsRNAs termed small activating RNAs (saRNAs),[103] but has also been demonstrated for endogenous microRNA.[104]
Interactions between microRNAs and complementary sequences on genes and even pseudogenes that share sequence homology are thought to be a back channel of communication regulating expression levels between paralogous genes (genes having a similar structure indicating divergence from a common ancestral gene). Given the name «competing endogenous RNAs» (ceRNAs), these microRNAs bind to «microRNA response elements» on genes and pseudogenes and may provide another explanation for the persistence of non-coding DNA.[105]
Some researches show that mRNA cargo of exosomes may have a role in implantation, they can savage an adhesion between trophoblast and endometrium or support the adhesion by down regulating or up regulating expression of genes involved in adhesion/invasion.[106]
Moreover, miRNA as miR-183/96/182 seems to play a key role in circadian rhythm.[107]
Evolution[edit]
miRNAs are well conserved in both plants and animals, and are thought to be a vital and evolutionarily ancient component of gene regulation.[108][109][110][111][112] While core components of the microRNA pathway are conserved between plants and animals, miRNA repertoires in the two kingdoms appear to have emerged independently with different primary modes of action.[113][114]
microRNAs are useful phylogenetic markers because of their apparently low rate of evolution.[115] microRNAs’ origin as a regulatory mechanism developed from previous RNAi machinery that was initially used as a defense against exogenous genetic material such as viruses.[116] Their origin may have permitted the development of morphological innovation, and by making gene expression more specific and ‘fine-tunable’, permitted the genesis of complex organs[117] and perhaps, ultimately, complex life.[112] Rapid bursts of morphological innovation are generally associated with a high rate of microRNA accumulation.[115][117]
New microRNAs are created in multiple ways. Novel microRNAs can originate from the random formation of hairpins in «non-coding» sections of DNA (i.e. introns or intergene regions), but also by the duplication and modification of existing microRNAs.[118] microRNAs can also form from inverted duplications of protein-coding sequences, which allows for the creation of a foldback hairpin structure.[119] The rate of evolution (i.e. nucleotide substitution) in recently originated microRNAs is comparable to that elsewhere in the non-coding DNA, implying evolution by neutral drift; however, older microRNAs have a much lower rate of change (often less than one substitution per hundred million years),[112] suggesting that once a microRNA gains a function, it undergoes purifying selection.[118] Individual regions within an miRNA gene face different evolutionary pressures, where regions that are vital for processing and function have higher levels of conservation.[120] At this point, a microRNA is rarely lost from an animal’s genome,[112] although newer microRNAs (thus presumably non-functional) are frequently lost.[118] In Arabidopsis thaliana, the net flux of miRNA genes has been predicted to be between 1.2 and 3.3 genes per million years.[121] This makes them a valuable phylogenetic marker, and they are being looked upon as a possible solution to outstanding phylogenetic problems such as the relationships of arthropods.[122] On the other hand, in multiple cases microRNAs correlate poorly with phylogeny, and it is possible that their phylogenetic concordance largely reflects a limited sampling of microRNAs.[123]
microRNAs feature in the genomes of most eukaryotic organisms, from the brown algae[124] to the animals. However, the difference in how these microRNAs function and the way they are processed suggests that microRNAs arose independently in plants and animals.[125]
Focusing on the animals, the genome of Mnemiopsis leidyi[126] appears to lack recognizable microRNAs, as well as the nuclear proteins Drosha and Pasha, which are critical to canonical microRNA biogenesis. It is the only animal thus far reported to be missing Drosha. MicroRNAs play a vital role in the regulation of gene expression in all non-ctenophore animals investigated thus far except for Trichoplax adhaerens, the only known member of the phylum Placozoa.[127]
Across all species, in excess of 5000 different miRNAs had been identified by March 2010.[128] Whilst short RNA sequences (50 – hundreds of base pairs) of a broadly comparable function occur in bacteria, bacteria lack true microRNAs.[129]
Experimental detection and manipulation[edit]
While researchers focused on miRNA expression in physiological and pathological processes, various technical variables related to microRNA isolation emerged. The stability of stored miRNA samples has been questioned.[74] microRNAs degrade much more easily than mRNAs, partly due to their length, but also because of ubiquitously present RNases. This makes it necessary to cool samples on ice and use RNase-free equipment.[130]
microRNA expression can be quantified in a two-step polymerase chain reaction process of modified RT-PCR followed by quantitative PCR. Variations of this method achieve absolute or relative quantification.[131] miRNAs can also be hybridized to microarrays, slides or chips with probes to hundreds or thousands of miRNA targets, so that relative levels of miRNAs can be determined in different samples.[132] microRNAs can be both discovered and profiled by high-throughput sequencing methods (microRNA sequencing).[133] The activity of an miRNA can be experimentally inhibited using a locked nucleic acid (LNA) oligo, a Morpholino oligo[134][135] or a 2′-O-methyl RNA oligo.[136] A specific miRNA can be silenced by a complementary antagomir. microRNA maturation can be inhibited at several points by steric-blocking oligos.[137][138] The miRNA target site of an mRNA transcript can also be blocked by a steric-blocking oligo.[139] For the «in situ» detection of miRNA, LNA[140] or Morpholino[141] probes can be used. The locked conformation of LNA results in enhanced hybridization properties and increases sensitivity and selectivity, making it ideal for detection of short miRNA.[142]
High-throughput quantification of miRNAs is error prone, for the larger variance (compared to mRNAs) that comes with methodological problems. mRNA-expression is therefore often analyzed to check for miRNA-effects in their levels (e.g. in[143]). Databases can be used to pair mRNA- and miRNA-data that predict miRNA-targets based on their base sequence.[144][145] While this is usually done after miRNAs of interest have been detected (e. g. because of high expression levels), ideas for analysis tools that integrate mRNA- and miRNA-expression information have been proposed.[146][147]
Disease[edit]
Just as miRNA is involved in the normal functioning of eukaryotic cells, so has dysregulation of miRNA been associated with disease. A manually curated, publicly available database, miR2Disease, documents known relationships between miRNA dysregulation and human disease.[148]
Inherited diseases[edit]
A mutation in the seed region of miR-96 causes hereditary progressive hearing loss.[149]
A mutation in the seed region of miR-184 causes hereditary keratoconus with anterior polar cataract.[150]
Deletion of the miR-17~92 cluster causes skeletal and growth defects.[151]
Cancer[edit]
Role of miRNA in a cancer cell
The first human disease known to be associated with miRNA deregulation was chronic lymphocytic leukemia.[152] Many other miRNAs also have links with cancer and accordingly are sometimes referred to as «oncomirs».[153] In malignant B cells miRNAs participate in pathways fundamental to B cell development like B-cell receptor (BCR) signalling, B-cell migration/adhesion, cell-cell interactions in immune niches and the production and class-switching of immunoglobulins. MiRNAs influence B cell maturation, generation of pre-, marginal zone, follicular, B1, plasma and memory B cells.[154]
Another role for miRNA in cancers is to use their expression level for prognosis. In NSCLC samples, low miR-324a levels may serve as an indicator of poor survival.[155] Either high miR-185 or low miR-133b levels may correlate with metastasis and poor survival in colorectal cancer.[156]
Furthermore, specific miRNAs may be associated with certain histological subtypes of colorectal cancer. For instance, expression levels of miR-205 and miR-373 have been shown to be increased in mucinous colorectal cancers and mucin-producing Ulcerative Colitis-associated colon cancers, but not in sporadic colonic adenocarcinoma that lack mucinous components.[157] In-vitro studies suggested that miR-205 and miR-373 may functionally induce different features of mucinous-associated neoplastic progression in intestinal epithelial cells.[157]
Hepatocellular carcinoma cell proliferation may arise from miR-21 interaction with MAP2K3, a tumor repressor gene.[158] Optimal treatment for cancer involves accurately identifying patients for risk-stratified therapy. Those with a rapid response to initial treatment may benefit from truncated treatment regimens, showing the value of accurate disease response measures. Cell-free circulating miRNAs (cimiRNAs) are highly stable in blood, are overexpressed in cancer and are quantifiable within the diagnostic laboratory. In classical Hodgkin lymphoma, plasma miR-21, miR-494, and miR-1973 are promising disease response biomarkers.[159] Circulating miRNAs have the potential to assist clinical decision making and aid interpretation of positron emission tomography combined with computerized tomography. They can be performed at each consultation to assess disease response and detect relapse.
MicroRNAs have the potential to be used as tools or targets for treatment of different cancers.[160] The specific microRNA, miR-506 has been found to work as a tumor antagonist in several studies. A significant number of cervical cancer samples were found to have downregulated expression of miR-506. Additionally, miR-506 works to promote apoptosis of cervical cancer cells, through its direct target hedgehog pathway transcription factor, Gli3.[161][162]
DNA repair and cancer[edit]
Many miRNAs can directly target and inhibit cell cycle genes to control cell proliferation. A new strategy for tumor treatment is to inhibit tumor cell proliferation by repairing the defective miRNA pathway in tumors.[163]
Cancer is caused by the accumulation of mutations from either DNA damage or uncorrected errors in DNA replication.[164] Defects in DNA repair cause the accumulation of mutations, which can lead to cancer.[165] Several genes involved in DNA repair are regulated by microRNAs.[166]
Germ line mutations in DNA repair genes cause only 2–5% of colon cancer cases.[167] However, altered expression of microRNAs, causing DNA repair deficiencies, are frequently associated with cancers and may be an important causal factor. Among 68 sporadic colon cancers with reduced expression of the DNA mismatch repair protein MLH1, most were found to be deficient due to epigenetic methylation of the CpG island of the MLH1 gene.[168] However, up to 15% of MLH1-deficiencies in sporadic colon cancers appeared to be due to over-expression of the microRNA miR-155, which represses MLH1 expression.[169]
In 29–66%[170][171] of glioblastomas, DNA repair is deficient due to epigenetic methylation of the MGMT gene, which reduces protein expression of MGMT. However, for 28% of glioblastomas, the MGMT protein is deficient, but the MGMT promoter is not methylated.[170] In glioblastomas without methylated MGMT promoters, the level of microRNA miR-181d is inversely correlated with protein expression of MGMT and the direct target of miR-181d is the MGMT mRNA 3’UTR (the three prime untranslated region of MGMT mRNA).[170] Thus, in 28% of glioblastomas, increased expression of miR-181d and reduced expression of DNA repair enzyme MGMT may be a causal factor.
HMGA proteins (HMGA1a, HMGA1b and HMGA2) are implicated in cancer, and expression of these proteins is regulated by microRNAs. HMGA expression is almost undetectable in differentiated adult tissues, but is elevated in many cancers. HMGA proteins are polypeptides of ~100 amino acid residues characterized by a modular sequence organization. These proteins have three highly positively charged regions, termed AT hooks, that bind the minor groove of AT-rich DNA stretches in specific regions of DNA. Human neoplasias, including thyroid, prostatic, cervical, colorectal, pancreatic and ovarian carcinomas, show a strong increase of HMGA1a and HMGA1b proteins.[172] Transgenic mice with HMGA1 targeted to lymphoid cells develop aggressive lymphoma, showing that high HMGA1 expression is associated with cancers and that HMGA1 can act as an oncogene.[173] HMGA2 protein specifically targets the promoter of ERCC1, thus reducing expression of this DNA repair gene.[174] ERCC1 protein expression was deficient in 100% of 47 evaluated colon cancers (though the extent to which HGMA2 was involved is not known).[175]
Single Nucleotide polymorphisms (SNPs) can alter the binding of miRNAs on 3’UTRs for example the case of hsa-mir181a and hsa-mir181b on the CDON tumor suppressor gene.[176]
Heart disease[edit]
The global role of miRNA function in the heart has been addressed by conditionally inhibiting miRNA maturation in the murine heart. This revealed that miRNAs play an essential role during its development.[177][178] miRNA expression profiling studies demonstrate that expression levels of specific miRNAs change in diseased human hearts, pointing to their involvement in cardiomyopathies.[179][180][181] Furthermore, animal studies on specific miRNAs identified distinct roles for miRNAs both during heart development and under pathological conditions, including the regulation of key factors important for cardiogenesis, the hypertrophic growth response and cardiac conductance.[178][182][183][184][185][186] Another role for miRNA in cardiovascular diseases is to use their expression levels for diagnosis, prognosis or risk stratification.[187] miRNA’s in animal models have also been linked to cholesterol metabolism and regulation.
miRNA-712[edit]
Murine microRNA-712 is a potential biomarker (i.e. predictor) for atherosclerosis, a cardiovascular disease of the arterial wall associated with lipid retention and inflammation.[188] Non-laminar blood flow also correlates with development of atherosclerosis as mechanosenors of endothelial cells respond to the shear force of disturbed flow (d-flow).[189] A number of pro-atherogenic genes including matrix metalloproteinases (MMPs) are upregulated by d-flow,[189] mediating pro-inflammatory and pro-angiogenic signals. These findings were observed in ligated carotid arteries of mice to mimic the effects of d-flow. Within 24 hours, pre-existing immature miR-712 formed mature miR-712 suggesting that miR-712 is flow-sensitive.[189] Coinciding with these results, miR-712 is also upregulated in endothelial cells exposed to naturally occurring d-flow in the greater curvature of the aortic arch.[189]
Origin[edit]
Pre-mRNA sequence of miR-712 is generated from the murine ribosomal RN45s gene at the internal transcribed spacer region 2 (ITS2).[189] XRN1 is an exonuclease that degrades the ITS2 region during processing of RN45s.[189] Reduction of XRN1 under d-flow conditions therefore leads to the accumulation of miR-712.[189]
Mechanism[edit]
MiR-712 targets tissue inhibitor of metalloproteinases 3 (TIMP3).[189] TIMPs normally regulate activity of matrix metalloproteinases (MMPs) which degrade the extracellular matrix (ECM). Arterial ECM is mainly composed of collagen and elastin fibers, providing the structural support and recoil properties of arteries.[190] These fibers play a critical role in regulation of vascular inflammation and permeability, which are important in the development of atherosclerosis.[191] Expressed by endothelial cells, TIMP3 is the only ECM-bound TIMP.[190] A decrease in TIMP3 expression results in an increase of ECM degradation in the presence of d-flow. Consistent with these findings, inhibition of pre-miR712 increases expression of TIMP3 in cells, even when exposed to turbulent flow.[189]
TIMP3 also decreases the expression of TNFα (a pro-inflammatory regulator) during turbulent flow.[189] Activity of TNFα in turbulent flow was measured by the expression of TNFα-converting enzyme (TACE) in blood. TNFα decreased if miR-712 was inhibited or TIMP3 overexpressed,[189] suggesting that miR-712 and TIMP3 regulate TACE activity in turbulent flow conditions.
Anti-miR-712 effectively suppresses d-flow-induced miR-712 expression and increases TIMP3 expression.[189] Anti-miR-712 also inhibits vascular hyperpermeability, thereby significantly reducing atherosclerosis lesion development and immune cell infiltration.[189]
Human homolog microRNA-205[edit]
The human homolog of miR-712 was found on the RN45s homolog gene, which maintains similar miRNAs to mice.[189] MiR-205 of humans share similar sequences with miR-712 of mice and is conserved across most vertebrates.[189] MiR-205 and miR-712 also share more than 50% of the cell signaling targets, including TIMP3.[189]
When tested, d-flow decreased the expression of XRN1 in humans as it did in mice endothelial cells, indicating a potentially common role of XRN1 in humans.[189]
Kidney disease[edit]
Targeted deletion of Dicer in the FoxD1-derived renal progenitor cells in a murine model resulted in a complex renal phenotype including expansion of nephron progenitors, fewer renin cells, smooth muscle arterioles, progressive mesangial loss and glomerular aneurysms.[192] High throughput whole transcriptome profiling of the FoxD1-Dicer knockout mouse model revealed ectopic upregulation of pro-apoptotic gene, Bcl2L11 (Bim) and dysregulation of the p53 pathway with increase in p53 effector genes including Bax, Trp53inp1, Jun, Cdkn1a, Mmp2, and Arid3a. p53 protein levels remained unchanged, suggesting that FoxD1 stromal miRNAs directly repress p53-effector genes. Using a lineage tracing approach followed by Fluorescent-activated cell sorting, miRNA profiling of the FoxD1-derived cells not only comprehensively defined the transcriptional landscape of miRNAs that are critical for vascular development, but also identified key miRNAs that are likely to modulate the renal phenotype in its absence. These miRNAs include miRs‐10a, 18a, 19b, 24, 30c, 92a, 106a, 130a, 152, 181a, 214, 222, 302a, 370, and 381 that regulate Bcl2L11 (Bim) and miRs‐15b, 18a, 21, 30c, 92a, 106a, 125b‐5p, 145, 214, 222, 296‐5p and 302a that regulate p53-effector genes. Consistent with the profiling results, ectopic apoptosis was observed in the cellular derivatives of the FoxD1 derived progenitor lineage and reiterates the importance of renal stromal miRNAs in cellular homeostasis.[192]
Nervous system[edit]
miRNAs appear to regulate the development and function of the nervous system.[193] Neural miRNAs are involved at various stages of synaptic development, including dendritogenesis (involving miR-132, miR-134 and miR-124), synapse formation[194] and synapse maturation (where miR-134 and miR-138 are thought to be involved).[195] Elimination of miRNA formation in mice by experimental silencing of Dicer has led to pathological outcomes, such as reduced neuronal size and motor abnormalities when silenced in striatal neurons[196] and neurodegeneration when silenced in forebrain neurons.[197] Some studies find altered miRNA expression in Alzheimer’s disease,[198] as well as schizophrenia, bipolar disorder, major depression and anxiety disorders.[199][200][201]
Stroke[edit]
According to the Center for Disease Control and Prevention, Stroke is one of the leading causes of death and long-term disability in America. 87% of the cases are ischemic strokes, which results from blockage in the artery of the brain that carries oxygen-rich blood. The obstruction of the blood flow means the brain cannot receive necessary nutrients, such as oxygen and glucose, and remove wastes, such as carbon dioxide.[202][203] miRNAs plays a role in posttranslational gene silencing by targeting genes in the pathogenesis of cerebral ischemia, such as the inflammatory, angiogenesis, and apoptotic pathway.[204]
Alcoholism[edit]
The vital role of miRNAs in gene expression is significant to addiction, specifically alcoholism.[205] Chronic alcohol abuse results in persistent changes in brain function mediated in part by alterations in gene expression.[205] miRNA global regulation of many downstream genes deems significant regarding the reorganization or synaptic connections or long term neural adaptations involving the behavioral change from alcohol consumption to withdrawal and/or dependence.[206] Up to 35 different miRNAs have been found to be altered in the alcoholic post-mortem brain, all of which target genes that include the regulation of the cell cycle, apoptosis, cell adhesion, nervous system development and cell signaling.[205] Altered miRNA levels were found in the medial prefrontal cortex of alcohol-dependent mice, suggesting the role of miRNA in orchestrating translational imbalances and the creation of differentially expressed proteins within an area of the brain where complex cognitive behavior and decision making likely originate.[207]
miRNAs can be either upregulated or downregulated in response to chronic alcohol use. miR-206 expression increased in the prefrontal cortex of alcohol-dependent rats, targeting the transcription factor brain-derived neurotrophic factor (BDNF) and ultimately reducing its expression. BDNF plays a critical role in the formation and maturation of new neurons and synapses, suggesting a possible implication in synapse growth/synaptic plasticity in alcohol abusers.[208] miR-155, important in regulating alcohol-induced neuroinflammation responses, was found to be upregulated, suggesting the role of microglia and inflammatory cytokines in alcohol pathophysiology.[209] Downregulation of miR-382 was found in the nucleus accumbens, a structure in the basal forebrain significant in regulating feelings of reward that power motivational habits. miR-382 is the target for the dopamine receptor D1 (DRD1), and its overexpression results in the upregulation of DRD1 and delta fosB, a transcription factor that activates a series of transcription events in the nucleus accumbens that ultimately result in addictive behaviors.[210] Alternatively, overexpressing miR-382 resulted in attenuated drinking and the inhibition of DRD1 and delta fosB upregulation in rat models of alcoholism, demonstrating the possibility of using miRNA-targeted pharmaceuticals in treatments.[210]
Obesity[edit]
miRNAs play crucial roles in the regulation of stem cell progenitors differentiating into adipocytes.[211] Studies to determine what role pluripotent stem cells play in adipogenesis, were examined in the immortalized human bone marrow-derived stromal cell line hMSC-Tert20.[212] Decreased expression of miR-155, miR-221, and miR-222, have been found during the adipogenic programming of both immortalized and primary hMSCs, suggesting that they act as negative regulators of differentiation. Conversely, ectopic expression of the miRNAs 155, 221, and 222 significantly inhibited adipogenesis and repressed induction of the master regulators PPARγ and CCAAT/enhancer-binding protein alpha (CEBPA).[213] This paves the way for possible genetic obesity treatments.
Another class of miRNAs that regulate insulin resistance, obesity, and diabetes, is the let-7 family. Let-7 accumulates in human tissues during the course of aging.[214] When let-7 was ectopically overexpressed to mimic accelerated aging, mice became insulin-resistant, and thus more prone to high fat diet-induced obesity and diabetes.[215] In contrast when let-7 was inhibited by injections of let-7-specific antagomirs, mice become more insulin-sensitive and remarkably resistant to high fat diet-induced obesity and diabetes. Not only could let-7 inhibition prevent obesity and diabetes, it could also reverse and cure the condition.[216] These experimental findings suggest that let-7 inhibition could represent a new therapy for obesity and type 2 diabetes.
Hemostasis[edit]
miRNAs also play crucial roles in the regulation of complex enzymatic cascades including the hemostatic blood coagulation system.[217] Large scale studies of functional miRNA targeting have recently uncovered rationale therapeutic targets in the hemostatic system.[218][219] They have been directly linked to Calcium homeostasis in the endoplasmic reticulum, which is critical in cell differentiation in early development.[220]
Plants[edit]
miRNAs are considered to be key regulators of many developmental, homeostatic, and immune processes in plants.[221] Their roles in plant development include shoot apical meristem development, leaf growth, flower formation, seed production, or root expansion.[222][223][224][225] In addition, they play a complex role in responses to various abiotic stresses comprising heat stress, low-temperature stress, drought stress, light stress, or gamma radiation exposure.[221]
Viruses[edit]
Viral microRNAs play an important role in the regulation of gene expression of viral and/or host genes to benefit the virus. Hence, miRNAs play a key role in host–virus interactions and pathogenesis of viral diseases.[226][227] The expression of transcription activators by human herpesvirus-6 DNA is believed to be regulated by viral miRNA.[228]
Target prediction[edit]
miRNAs can bind to target messenger RNA (mRNA) transcripts of protein-coding genes and negatively control their translation or cause mRNA degradation. It is of key importance to identify the miRNA targets accurately.[229] A comparison of the predictive performance of eighteen in silico algorithms is available.[230] Large scale studies of functional miRNA targeting suggest that many functional miRNAs can be missed by target prediction algorithms.[218]
See also[edit]
- Anti-miRNA oligonucleotides
- Gene expression
- List of miRNA gene prediction tools
- List of miRNA target prediction tools
- MicroDNA
- MiRNEST
- MIR222
- miR-324-5p
- RNA interference
- Small interfering RNA
- Small nucleolar RNA-derived microRNA
- C19MC miRNA cluster
References[edit]
- ^ Green, D; Dalmay, T; Chapman, T (February 2016). «Microguards and micromessengers of the genome». Heredity. 116 (2): 125–134. doi:10.1038/hdy.2015.84. PMC 4806885. PMID 26419338.
- ^ a b c Bartel DP (March 2018). «Metazoan MicroRNAs». Cell. 173 (1): 20–51. doi:10.1016/j.cell.2018.03.006. PMC 6091663. PMID 29570994.
- ^ Qureshi A, Thakur N, Monga I, Thakur A, Kumar M (1 January 2014). «VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets». Database. 2014: bau103. doi:10.1093/database/bau103. PMC 4224276. PMID 25380780.
- ^ a b c d Bartel DP (January 2009). «MicroRNAs: target recognition and regulatory functions». Cell. 136 (2): 215–33. doi:10.1016/j.cell.2009.01.002. PMC 3794896. PMID 19167326.
- ^ Fabian MR, Sonenberg N, Filipowicz W (2010). «Regulation of mRNA translation and stability by microRNAs». Annual Review of Biochemistry. 79: 351–79. doi:10.1146/annurev-biochem-060308-103103. PMID 20533884.
- ^ a b Bartel DP (January 2004). «MicroRNAs: genomics, biogenesis, mechanism, and function». Cell. 116 (2): 281–297. doi:10.1016/S0092-8674(04)00045-5. PMID 14744438.
- ^ Homo sapiens miRNAs in the miRBase at Manchester University
- ^ Alles J, Fehlmann T, Fischer U, Backes C, Galata V, Minet M, et al. (April 2019). «An estimate of the total number of true human miRNAs». Nucleic Acids Research. 47 (7): 3353–3364. doi:10.1093/nar/gkz097. PMC 6468295. PMID 30820533.
- ^ Fromm B, Domanska D, Høye E, Ovchinnikov V, Kang W, Aparicio-Puerta E, et al. (January 2020). «MirGeneDB 2.0: the metazoan microRNA complement». Nucleic Acids Research. 48 (D1): D132–D141. doi:10.1093/nar/gkz885. PMC 6943042. PMID 31598695.
- ^ Lim LP, Lau NC, Weinstein EG, Abdelhakim A, Yekta S, Rhoades MW, Burge CB, Bartel DP (April 2003). «The microRNAs of Caenorhabditis elegans». Genes & Development. 17 (8): 991–1008. doi:10.1101/gad.1074403. PMC 196042. PMID 12672692.
- ^ a b Lagos-Quintana M, Rauhut R, Yalcin A, Meyer J, Lendeckel W, Tuschl T (April 2002). «Identification of tissue-specific microRNAs from mouse». Current Biology. 12 (9): 735–9. doi:10.1016/S0960-9822(02)00809-6. PMID 12007417.
- ^ a b Kumar S, Reddy PH (September 2016). «Are circulating microRNAs peripheral biomarkers for Alzheimer’s disease?». Biochim Biophys Acta. 1862 (9): 1617–27. doi:10.1016/j.bbadis.2016.06.001. PMC 5343750. PMID 27264337.
- ^ van den Berg MM, Krauskopf J, Ramaekers JG, et al. (February 2020). «Circulating microRNAs as potential biomarkers for psychiatric and neurodegenerative disorders». Prog Neurobiol. 185: 101732. doi:10.1016/j.pneurobio.2019.101732. PMID 31816349.
- ^ a b c Lewis BP, Burge CB, Bartel DP (January 2005). «Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets». Cell. 120 (1): 15–20. doi:10.1016/j.cell.2004.12.035. PMID 15652477.
- ^ a b c Friedman RC, Farh KK, Burge CB, Bartel DP (January 2009). «Most mammalian mRNAs are conserved targets of microRNAs». Genome Research. 19 (1): 92–105. doi:10.1101/gr.082701.108. PMC 2612969. PMID 18955434.
- ^ Fromm B, Billipp T, Peck LE, Johansen M, Tarver JE, King BL, et al. (2015). «A Uniform System for the Annotation of Vertebrate microRNA Genes and the Evolution of the Human microRNAome». Annual Review of Genetics. 49: 213–42. doi:10.1146/annurev-genet-120213-092023. PMC 4743252. PMID 26473382.
- ^ a b c Lee RC, Feinbaum RL, Ambros V (December 1993). «The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14». Cell. 75 (5): 843–54. doi:10.1016/0092-8674(93)90529-Y. PMID 8252621.
- ^ a b Reinhart BJ, Slack FJ, Basson M, Pasquinelli AE, Bettinger JC, Rougvie AE, Horvitz HR, Ruvkun G (February 2000). «The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans». Nature. 403 (6772): 901–6. Bibcode:2000Natur.403..901R. doi:10.1038/35002607. PMID 10706289. S2CID 4384503.
- ^ a b Pasquinelli AE, Reinhart BJ, Slack F, Martindale MQ, Kuroda MI, Maller B, Hayward DC, Ball EE, Degnan B, Müller P, Spring J, Srinivasan A, Fishman M, Finnerty J, Corbo J, Levine M, Leahy P, Davidson E, Ruvkun G (November 2000). «Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA». Nature. 408 (6808): 86–9. Bibcode:2000Natur.408…86P. doi:10.1038/35040556. PMID 11081512. S2CID 4401732.
- ^ a b c Lagos-Quintana M, Rauhut R, Lendeckel W, Tuschl T (October 2001). «Identification of novel genes coding for small expressed RNAs». Science. 294 (5543): 853–8. Bibcode:2001Sci…294..853L. doi:10.1126/science.1064921. hdl:11858/00-001M-0000-0012-F65F-2. PMID 11679670. S2CID 18101169.
- ^ a b c Lau NC, Lim LP, Weinstein EG, Bartel DP (October 2001). «An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans». Science. 294 (5543): 858–62. Bibcode:2001Sci…294..858L. doi:10.1126/science.1065062. PMID 11679671. S2CID 43262684.
- ^ a b c Lee RC, Ambros V (October 2001). «An extensive class of small RNAs in Caenorhabditis elegans». Science. 294 (5543): 862–4. Bibcode:2001Sci…294..862L. doi:10.1126/science.1065329. PMID 11679672. S2CID 33480585.
- ^ Wienholds E, Kloosterman WP, Miska E, Alvarez-Saavedra E, Berezikov E, de Bruijn E, Horvitz HR, Kauppinen S, Plasterk RH (July 2005). «MicroRNA expression in zebrafish embryonic development». Science. 309 (5732): 310–1. Bibcode:2005Sci…309..310W. doi:10.1126/science.1114519. PMID 15919954. S2CID 38939571.
- ^ a b Jones-Rhoades MW, Bartel DP, Bartel B (2006). «MicroRNAS and their regulatory roles in plants». Annual Review of Plant Biology. 57: 19–53. doi:10.1146/annurev.arplant.57.032905.105218. PMID 16669754.
- ^ Brennecke J, Hipfner DR, Stark A, Russell RB, Cohen SM (April 2003). «bantam encodes a developmentally regulated microRNA that controls cell proliferation and regulates the proapoptotic gene hid in Drosophila». Cell. 113 (1): 25–36. doi:10.1016/S0092-8674(03)00231-9. PMID 12679032.
- ^ Cuellar TL, McManus MT (December 2005). «MicroRNAs and endocrine biology». The Journal of Endocrinology. 187 (3): 327–32. doi:10.1677/joe.1.06426. PMID 16423811.
- ^ Poy MN, Eliasson L, Krutzfeldt J, Kuwajima S, Ma X, Macdonald PE, Pfeffer S, Tuschl T, Rajewsky N, Rorsman P, Stoffel M (November 2004). «A pancreatic islet-specific microRNA regulates insulin secretion». Nature. 432 (7014): 226–30. Bibcode:2004Natur.432..226P. doi:10.1038/nature03076. PMID 15538371. S2CID 4415988.
- ^ Chen CZ, Li L, Lodish HF, Bartel DP (January 2004). «MicroRNAs modulate hematopoietic lineage differentiation». Science. 303 (5654): 83–6. Bibcode:2004Sci…303…83C. doi:10.1126/science.1091903. hdl:1721.1/7483. PMID 14657504. S2CID 7044929.
- ^ Wilfred BR, Wang WX, Nelson PT (July 2007). «Energizing miRNA research: a review of the role of miRNAs in lipid metabolism, with a prediction that miR-103/107 regulates human metabolic pathways». Molecular Genetics and Metabolism. 91 (3): 209–17. doi:10.1016/j.ymgme.2007.03.011. PMC 1978064. PMID 17521938.
- ^ Harfe BD, McManus MT, Mansfield JH, Hornstein E, Tabin CJ (August 2005). «The RNaseIII enzyme Dicer is required for morphogenesis but not patterning of the vertebrate limb». Proceedings of the National Academy of Sciences of the United States of America. 102 (31): 10898–903. Bibcode:2005PNAS..10210898H. doi:10.1073/pnas.0504834102. PMC 1182454. PMID 16040801.
- ^ Trang P, Weidhaas JB, Slack FJ (December 2008). «MicroRNAs as potential cancer therapeutics». Oncogene. 27 (Suppl 2): S52–7. doi:10.1038/onc.2009.353. PMID 19956180.
- ^ Li C, Feng Y, Coukos G, Zhang L (December 2009). «Therapeutic microRNA strategies in human cancer». The AAPS Journal. 11 (4): 747–57. doi:10.1208/s12248-009-9145-9. PMC 2782079. PMID 19876744.
- ^ Fasanaro P, Greco S, Ivan M, Capogrossi MC, Martelli F (January 2010). «microRNA: emerging therapeutic targets in acute ischemic diseases». Pharmacology & Therapeutics. 125 (1): 92–104. doi:10.1016/j.pharmthera.2009.10.003. PMID 19896977.
- ^ Hydbring P, Badalian-Very G (August 2013). «Clinical applications of microRNAs». F1000Research. 2: 136. doi:10.12688/f1000research.2-136.v2. PMC 3917658. PMID 24627783.
- ^ Wightman B, Ha I, Ruvkun G (December 1993). «Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans». Cell. 75 (5): 855–62. doi:10.1016/0092-8674(93)90530-4. PMID 8252622.
- ^ Giza DE, Calin GA (2015). «microRNA and Chronic Lymphocytic Leukemia». Advances in Experimental Medicine and Biology. 889: 23–40. doi:10.1007/978-3-319-23730-5_2. ISBN 978-3-319-23729-9. PMID 26658994.
- ^ Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T (March 2003). «A uniform system for microRNA annotation». RNA. 9 (3): 277–9. doi:10.1261/rna.2183803. PMC 1370393. PMID 12592000.
- ^ Griffiths-Jones S, Grocock RJ, van Dongen S, Bateman A, Enright AJ (January 2006). «miRBase: microRNA sequences, targets and gene nomenclature». Nucleic Acids Research. 34 (Database issue): D140–4. doi:10.1093/nar/gkj112. PMC 1347474. PMID 16381832.
- ^ Wright MW, Bruford EA (January 2011). «Naming ‘junk’: human non-protein coding RNA (ncRNA) gene nomenclature». Human Genomics. 5 (2): 90–8. doi:10.1186/1479-7364-5-2-90. PMC 3051107. PMID 21296742.
- ^ Hunt M, Banerjee S, Surana P, Liu M, Fuerst G, Mathioni S, Meyers BC, Nettleton D, Wise RP (2019). «Small RNA discovery in the interaction between barley and the powdery mildew pathogen». BMC Genomics. 20 (1): 19–53. doi:10.1186/s12864-019-5947-z. PMC 6657096. PMID 31345162.
- ^ a b c Lewis BP, Shih IH, Jones-Rhoades MW, Bartel DP, Burge CB (December 2003). «Prediction of mammalian microRNA targets». Cell. 115 (7): 787–98. doi:10.1016/S0092-8674(03)01018-3. PMID 14697198.
- ^ Ellwanger DC, Büttner FA, Mewes HW, Stümpflen V (May 2011). «The sufficient minimal set of miRNA seed types». Bioinformatics. 27 (10): 1346–50. doi:10.1093/bioinformatics/btr149. PMC 3087955. PMID 21441577.
- ^ Rajewsky N (June 2006). «microRNA target predictions in animals». Nature Genetics. 38 (6s): S8–13. doi:10.1038/ng1798. PMID 16736023. S2CID 23496396.
- ^ Krek A, Grün D, Poy MN, Wolf R, Rosenberg L, Epstein EJ, MacMenamin P, da Piedade I, Gunsalus KC, Stoffel M, Rajewsky N (May 2005). «Combinatorial microRNA target predictions». Nature Genetics. 37 (5): 495–500. doi:10.1038/ng1536. PMID 15806104. S2CID 22672750.
- ^ Thomson DW, Bracken CP, Goodall GJ (September 2011). «Experimental strategies for microRNA target identification». Nucleic Acids Research. 39 (16): 6845–53. doi:10.1093/nar/gkr330. PMC 3167600. PMID 21652644.
- ^ a b Lim LP, Lau NC, Garrett-Engele P, Grimson A, Schelter JM, Castle J, Bartel DP, Linsley PS, Johnson JM (February 2005). «Microarray analysis shows that some microRNAs downregulate large numbers of target mRNAs». Nature. 433 (7027): 769–73. Bibcode:2005Natur.433..769L. doi:10.1038/nature03315. PMID 15685193. S2CID 4430576.
- ^ Selbach M, Schwanhäusser B, Thierfelder N, Fang Z, Khanin R, Rajewsky N (September 2008). «Widespread changes in protein synthesis induced by microRNAs». Nature. 455 (7209): 58–63. Bibcode:2008Natur.455…58S. doi:10.1038/nature07228. PMID 18668040. S2CID 4429008.
- ^ Baek D, Villén J, Shin C, Camargo FD, Gygi SP, Bartel DP (September 2008). «The impact of microRNAs on protein output». Nature. 455 (7209): 64–71. Bibcode:2008Natur.455…64B. doi:10.1038/nature07242. PMC 2745094. PMID 18668037.
- ^ a b Rodriguez A, Griffiths-Jones S, Ashurst JL, Bradley A (October 2004). «Identification of mammalian microRNA host genes and transcription units». Genome Research. 14 (10A): 1902–10. doi:10.1101/gr.2722704. PMC 524413. PMID 15364901.
- ^ a b c d Cai X, Hagedorn CH, Cullen BR (December 2004). «Human microRNAs are processed from capped, polyadenylated transcripts that can also function as mRNAs». RNA. 10 (12): 1957–66. doi:10.1261/rna.7135204. PMC 1370684. PMID 15525708.
- ^ Weber MJ (January 2005). «New human and mouse microRNA genes found by homology search». The FEBS Journal. 272 (1): 59–73. doi:10.1111/j.1432-1033.2004.04389.x. PMID 15634332. S2CID 32923462.
- ^ Kim YK, Kim VN (February 2007). «Processing of intronic microRNAs». The EMBO Journal. 26 (3): 775–83. doi:10.1038/sj.emboj.7601512. PMC 1794378. PMID 17255951.
- ^ Baskerville S, Bartel DP (March 2005). «Microarray profiling of microRNAs reveals frequent coexpression with neighboring miRNAs and host genes». RNA. 11 (3): 241–7. doi:10.1261/rna.7240905. PMC 1370713. PMID 15701730.
- ^ a b c Lee Y, Kim M, Han J, Yeom KH, Lee S, Baek SH, Kim VN (October 2004). «MicroRNA genes are transcribed by RNA polymerase II». The EMBO Journal. 23 (20): 4051–60. doi:10.1038/sj.emboj.7600385. PMC 524334. PMID 15372072.
- ^ Zhou X, Ruan J, Wang G, Zhang W (March 2007). «Characterization and identification of microRNA core promoters in four model species». PLOS Computational Biology. 3 (3): e37. Bibcode:2007PLSCB…3…37Z. doi:10.1371/journal.pcbi.0030037. PMC 1817659. PMID 17352530.
- ^ Faller M, Guo F (November 2008). «MicroRNA biogenesis: there’s more than one way to skin a cat». Biochimica et Biophysica Acta (BBA) — Gene Regulatory Mechanisms. 1779 (11): 663–7. doi:10.1016/j.bbagrm.2008.08.005. PMC 2633599. PMID 18778799.
- ^ Lee Y, Ahn C, Han J, Choi H, Kim J, Yim J, Lee J, Provost P, Rådmark O, Kim S, Kim VN (September 2003). «The nuclear RNase III Drosha initiates microRNA processing». Nature. 425 (6956): 415–9. Bibcode:2003Natur.425..415L. doi:10.1038/nature01957. PMID 14508493. S2CID 4421030.
- ^ Gregory RI, Chendrimada TP, Shiekhattar R (2006). «MicroRNA biogenesis: isolation and characterization of the microprocessor complex». MicroRNA Protocols. Methods in Molecular Biology. Vol. 342. pp. 33–47. doi:10.1385/1-59745-123-1:33. ISBN 978-1-59745-123-9. PMID 16957365.
- ^ Han J, Lee Y, Yeom KH, Kim YK, Jin H, Kim VN (December 2004). «The Drosha-DGCR8 complex in primary microRNA processing». Genes & Development. 18 (24): 3016–27. doi:10.1101/gad.1262504. PMC 535913. PMID 15574589.
- ^ Han J, Lee Y, Yeom KH, Nam JW, Heo I, Rhee JK, Sohn SY, Cho Y, Zhang BT, Kim VN (June 2006). «Molecular basis for the recognition of primary microRNAs by the Drosha-DGCR8 complex». Cell. 125 (5): 887–901. doi:10.1016/j.cell.2006.03.043. PMID 16751099.
- ^ Conrad T, Marsico A, Gehre M, Orom UA (October 2014). «Microprocessor activity controls differential miRNA biogenesis in Vivo». Cell Reports. 9 (2): 542–54. doi:10.1016/j.celrep.2014.09.007. PMID 25310978.
- ^ Auyeung VC, Ulitsky I, McGeary SE, Bartel DP (February 2013). «Beyond secondary structure: primary-sequence determinants license pri-miRNA hairpins for processing». Cell. 152 (4): 844–58. doi:10.1016/j.cell.2013.01.031. PMC 3707628. PMID 23415231.
- ^ Ali PS, Ghoshdastider U, Hoffmann J, Brutschy B, Filipek S (November 2012). «Recognition of the let-7g miRNA precursor by human Lin28B». FEBS Letters. 586 (22): 3986–90. doi:10.1016/j.febslet.2012.09.034. PMID 23063642. S2CID 28899778.
- ^ Berezikov E, Chung WJ, Willis J, Cuppen E, Lai EC (October 2007). «Mammalian mirtron genes». Molecular Cell. 28 (2): 328–36. doi:10.1016/j.molcel.2007.09.028. PMC 2763384. PMID 17964270.
- ^ a b Kawahara Y, Megraw M, Kreider E, Iizasa H, Valente L, Hatzigeorgiou AG, Nishikura K (September 2008). «Frequency and fate of microRNA editing in human brain». Nucleic Acids Research. 36 (16): 5270–80. doi:10.1093/nar/gkn479. PMC 2532740. PMID 18684997.
- ^ Winter J, Jung S, Keller S, Gregory RI, Diederichs S (March 2009). «Many roads to maturity: microRNA biogenesis pathways and their regulation». Nature Cell Biology. 11 (3): 228–34. doi:10.1038/ncb0309-228. PMID 19255566. S2CID 205286318.
- ^ Ohman M (October 2007). «A-to-I editing challenger or ally to the microRNA process». Biochimie. 89 (10): 1171–6. doi:10.1016/j.biochi.2007.06.002. PMID 17628290.
- ^ a b Murchison EP, Hannon GJ (June 2004). «miRNAs on the move: miRNA biogenesis and the RNAi machinery». Current Opinion in Cell Biology. 16 (3): 223–9. doi:10.1016/j.ceb.2004.04.003. PMID 15145345.
- ^ a b c Lund E, Dahlberg JE (2006). «Substrate selectivity of exportin 5 and Dicer in the biogenesis of microRNAs». Cold Spring Harbor Symposia on Quantitative Biology. 71: 59–66. doi:10.1101/sqb.2006.71.050. PMID 17381281.
- ^ Park JE, Heo I, Tian Y, Simanshu DK, Chang H, Jee D, Patel DJ, Kim VN (July 2011). «Dicer recognizes the 5′ end of RNA for efficient and accurate processing». Nature. 475 (7355): 201–5. doi:10.1038/nature10198. PMC 4693635. PMID 21753850.
- ^ Ji X (2008). «The mechanism of RNase III action: how dicer dices». RNA Interference. Current Topics in Microbiology and Immunology. Vol. 320. pp. 99–116. doi:10.1007/978-3-540-75157-1_5. ISBN 978-3-540-75156-4. PMID 18268841.
- ^ Mirihana Arachchilage G, Dassanayake AC, Basu S (February 2015). «A potassium ion-dependent RNA structural switch regulates human pre-miRNA 92b maturation». Chemistry & Biology. 22 (2): 262–72. doi:10.1016/j.chembiol.2014.12.013. PMID 25641166.
- ^ Sohel MH (2016). «Extracellular/Circulating MicroRNAs: Release Mechanisms, Functions and Challenges». Achievements in the Life Sciences. 10 (2): 175–186. doi:10.1016/j.als.2016.11.007.
- ^ a b Boeckel JN, Reis SM, Leistner D, Thomé CE, Zeiher AM, Fichtlscherer S, Keller T (April 2014). «From heart to toe: heart’s contribution on peripheral microRNA levels». International Journal of Cardiology. 172 (3): 616–7. doi:10.1016/j.ijcard.2014.01.082. PMID 24508494.
- ^ Lelandais-Brière C, Sorin C, Declerck M, Benslimane A, Crespi M, Hartmann C (March 2010). «Small RNA diversity in plants and its impact in development». Current Genomics. 11 (1): 14–23. doi:10.2174/138920210790217918. PMC 2851111. PMID 20808519.
- ^ Rana TM (January 2007). «Illuminating the silence: understanding the structure and function of small RNAs». Nature Reviews Molecular Cell Biology. 8 (1): 23–36. doi:10.1038/nrm2085. PMID 17183358. S2CID 8966239.
- ^ a b Schwarz DS, Zamore PD (May 2002). «Why do miRNAs live in the miRNP?». Genes & Development. 16 (9): 1025–31. doi:10.1101/gad.992502. PMID 12000786.
- ^ Krol J, Sobczak K, Wilczynska U, Drath M, Jasinska A, Kaczynska D, Krzyzosiak WJ (October 2004). «Structural features of microRNA (miRNA) precursors and their relevance to miRNA biogenesis and small interfering RNA/short hairpin RNA design». The Journal of Biological Chemistry. 279 (40): 42230–9. doi:10.1074/jbc.M404931200. PMID 15292246.
- ^ Khvorova A, Reynolds A, Jayasena SD (October 2003). «Functional siRNAs and miRNAs exhibit strand bias». Cell. 115 (2): 209–16. doi:10.1016/S0092-8674(03)00801-8. PMID 14567918.
- ^ Schwarz DS, Hutvágner G, Du T, Xu Z, Aronin N, Zamore PD (October 2003). «Asymmetry in the assembly of the RNAi enzyme complex». Cell. 115 (2): 199–208. doi:10.1016/S0092-8674(03)00759-1. PMID 14567917.
- ^ Lin SL, Chang D, Ying SY (August 2005). «Asymmetry of intronic pre-miRNA structures in functional RISC assembly». Gene. 356: 32–8. doi:10.1016/j.gene.2005.04.036. PMC 1788082. PMID 16005165.
- ^ Okamura K, Chung WJ, Lai EC (September 2008). «The long and short of inverted repeat genes in animals: microRNAs, mirtrons and hairpin RNAs». Cell Cycle. 7 (18): 2840–5. doi:10.4161/cc.7.18.6734. PMC 2697033. PMID 18769156.
- ^ a b Pratt AJ, MacRae IJ (July 2009). «The RNA-induced silencing complex: a versatile gene-silencing machine». The Journal of Biological Chemistry. 284 (27): 17897–901. doi:10.1074/jbc.R900012200. PMC 2709356. PMID 19342379.
- ^ MacRae IJ, Ma E, Zhou M, Robinson CV, Doudna JA (January 2008). «In vitro reconstitution of the human RISC-loading complex». Proceedings of the National Academy of Sciences of the United States of America. 105 (2): 512–7. Bibcode:2008PNAS..105..512M. doi:10.1073/pnas.0710869105. PMC 2206567. PMID 18178619.
- ^ Mourelatos Z, Dostie J, Paushkin S, Sharma A, Charroux B, Abel L, Rappsilber J, Mann M, Dreyfuss G (March 2002). «miRNPs: a novel class of ribonucleoproteins containing numerous microRNAs». Genes & Development. 16 (6): 720–8. doi:10.1101/gad.974702. PMC 155365. PMID 11914277.
- ^ Meister G, Landthaler M, Peters L, Chen PY, Urlaub H, Lührmann R, Tuschl T (December 2005). «Identification of novel argonaute-associated proteins». Current Biology. 15 (23): 2149–55. doi:10.1016/j.cub.2005.10.048. PMID 16289642.
- ^ Jing Q, Huang S, Guth S, Zarubin T, Motoyama A, Chen J, Di Padova F, Lin SC, Gram H, Han J (March 2005). «Involvement of microRNA in AU-rich element-mediated mRNA instability». Cell. 120 (5): 623–34. doi:10.1016/j.cell.2004.12.038. PMID 15766526.
- ^ a b c Kai ZS, Pasquinelli AE (January 2010). «MicroRNA assassins: factors that regulate the disappearance of miRNAs». Nature Structural & Molecular Biology. 17 (1): 5–10. doi:10.1038/nsmb.1762. PMC 6417416. PMID 20051982.
- ^ Chatterjee S, Grosshans H (September 2009). «Active turnover modulates mature microRNA activity in Caenorhabditis elegans». Nature. 461 (7263): 546–9. Bibcode:2009Natur.461..546C. doi:10.1038/nature08349. PMID 19734881. S2CID 4414841.
- ^ a b c Morozova N, Zinovyev A, Nonne N, Pritchard LL, Gorban AN, Harel-Bellan A (September 2012). «Kinetic signatures of microRNA modes of action». RNA. 18 (9): 1635–55. doi:10.1261/rna.032284.112. PMC 3425779. PMID 22850425.
- ^ Wang XJ, Reyes JL, Chua NH, Gaasterland T (2004). «Prediction and identification of Arabidopsis thaliana microRNAs and their mRNA targets». Genome Biology. 5 (9): R65. doi:10.1186/gb-2004-5-9-r65. PMC 522872. PMID 15345049.
- ^ Kawasaki H, Taira K (2004). «MicroRNA-196 inhibits HOXB8 expression in myeloid differentiation of HL60 cells». Nucleic Acids Symposium Series. 48 (1): 211–2. doi:10.1093/nass/48.1.211. PMID 17150553.
- ^ a b Moxon S, Jing R, Szittya G, Schwach F, Rusholme Pilcher RL, Moulton V, Dalmay T (October 2008). «Deep sequencing of tomato short RNAs identifies microRNAs targeting genes involved in fruit ripening». Genome Research. 18 (10): 1602–9. doi:10.1101/gr.080127.108. PMC 2556272. PMID 18653800.
- ^ Mazière P, Enright AJ (June 2007). «Prediction of microRNA targets». Drug Discovery Today. 12 (11–12): 452–8. doi:10.1016/j.drudis.2007.04.002. PMID 17532529.
- ^ Williams AE (February 2008). «Functional aspects of animal microRNAs». Cellular and Molecular Life Sciences. 65 (4): 545–62. doi:10.1007/s00018-007-7355-9. PMID 17965831. S2CID 5708394.
- ^ Eulalio A, Huntzinger E, Nishihara T, Rehwinkel J, Fauser M, Izaurralde E (January 2009). «Deadenylation is a widespread effect of miRNA regulation». RNA. 15 (1): 21–32. doi:10.1261/rna.1399509. PMC 2612776. PMID 19029310.
- ^ Bazzini AA, Lee MT, Giraldez AJ (April 2012). «Ribosome profiling shows that miR-430 reduces translation before causing mRNA decay in zebrafish». Science. 336 (6078): 233–7. Bibcode:2012Sci…336..233B. doi:10.1126/science.1215704. PMC 3547538. PMID 22422859.
- ^ Djuranovic S, Nahvi A, Green R (April 2012). «miRNA-mediated gene silencing by translational repression followed by mRNA deadenylation and decay». Science. 336 (6078): 237–40. Bibcode:2012Sci…336..237D. doi:10.1126/science.1215691. PMC 3971879. PMID 22499947.
- ^ Tan Y, Zhang B, Wu T, Skogerbø G, Zhu X, Guo X, He S, Chen R (February 2009). «Transcriptional inhibiton of Hoxd4 expression by miRNA-10a in human breast cancer cells». BMC Molecular Biology. 10 (1): 12. doi:10.1186/1471-2199-10-12. PMC 2680403. PMID 19232136.
- ^ Hawkins PG, Morris KV (March 2008). «RNA and transcriptional modulation of gene expression». Cell Cycle. 7 (5): 602–7. doi:10.4161/cc.7.5.5522. PMC 2877389. PMID 18256543.
- ^ Stark A, Brennecke J, Bushati N, Russell RB, Cohen SM (December 2005). «Animal MicroRNAs confer robustness to gene expression and have a significant impact on 3’UTR evolution». Cell. 123 (6): 1133–46. doi:10.1016/j.cell.2005.11.023. PMID 16337999.
- ^ He L, Hannon GJ (July 2004). «MicroRNAs: small RNAs with a big role in gene regulation». Nature Reviews. Genetics. 5 (7): 522–531. doi:10.1038/nrg1379. PMID 15211354. S2CID 5270062.
- ^ Li LC (2008). «Small RNA-Mediated Gene Activation». In Morris KV (ed.). RNA and the Regulation of Gene Expression: A Hidden Layer of Complexity. Horizon Scientific Press. ISBN 978-1-904455-25-7.
- ^ Place RF, Li LC, Pookot D, Noonan EJ, Dahiya R (February 2008). «MicroRNA-373 induces expression of genes with complementary promoter sequences». Proceedings of the National Academy of Sciences of the United States of America. 105 (5): 1608–13. Bibcode:2008PNAS..105.1608P. doi:10.1073/pnas.0707594105. PMC 2234192. PMID 18227514.
- ^ Salmena L, Poliseno L, Tay Y, Kats L, Pandolfi PP (August 2011). «A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language?». Cell. 146 (3): 353–8. doi:10.1016/j.cell.2011.07.014. PMC 3235919. PMID 21802130.
- ^ Cuman C, Van Sinderen M, Gantier MP, Rainczuk K, Sorby K, Rombauts L, et al. (October 2015). «Human Blastocyst Secreted microRNA Regulate Endometrial Epithelial Cell Adhesion». EBioMedicine. 2 (10): 1528–1535. doi:10.1016/j.ebiom.2015.09.003. PMC 4634783. PMID 26629549.
- ^ Zhou L, Miller C, Miraglia LJ, Romero A, Mure LS, Panda S, Kay SA (January 2021). «A genome-wide microRNA screen identifies the microRNA-183/96/182 cluster as a modulator of circadian rhythms». Proceedings of the National Academy of Sciences of the United States of America. 118 (1): e2020454118. Bibcode:2021PNAS..11820454Z. doi:10.1073/pnas.2020454118. PMC 7817116. PMID 33443164. S2CID 230713808.
- «MicroRNAs Play Key Role in Regulation of Circadian Rhythms». Science News. 6 January 2021.
- ^ Axtell MJ, Bartel DP (June 2005). «Antiquity of microRNAs and their targets in land plants». The Plant Cell. 17 (6): 1658–73. doi:10.1105/tpc.105.032185. PMC 1143068. PMID 15849273.
- ^ Tanzer A, Stadler PF (May 2004). «Molecular evolution of a microRNA cluster». Journal of Molecular Biology. 339 (2): 327–35. CiteSeerX 10.1.1.194.1598. doi:10.1016/j.jmb.2004.03.065. PMID 15136036.
- ^ Chen K, Rajewsky N (February 2007). «The evolution of gene regulation by transcription factors and microRNAs». Nature Reviews Genetics. 8 (2): 93–103. doi:10.1038/nrg1990. PMID 17230196. S2CID 174231.
- ^ Lee CT, Risom T, Strauss WM (April 2007). «Evolutionary conservation of microRNA regulatory circuits: an examination of microRNA gene complexity and conserved microRNA-target interactions through metazoan phylogeny». DNA and Cell Biology. 26 (4): 209–18. doi:10.1089/dna.2006.0545. PMID 17465887.
- ^ a b c d Peterson KJ, Dietrich MR, McPeek MA (July 2009). «MicroRNAs and metazoan macroevolution: insights into canalization, complexity, and the Cambrian explosion». BioEssays. 31 (7): 736–47. doi:10.1002/bies.200900033. PMID 19472371. S2CID 15364875.
- ^ Shabalina SA, Koonin EV (October 2008). «Origins and evolution of eukaryotic RNA interference». Trends in Ecology & Evolution. 23 (10): 578–87. doi:10.1016/j.tree.2008.06.005. PMC 2695246. PMID 18715673.
- ^ Axtell MJ, Westholm JO, Lai EC (2011). «Vive la différence: biogenesis and evolution of microRNAs in plants and animals». Genome Biology. 12 (4): 221. doi:10.1186/gb-2011-12-4-221. PMC 3218855. PMID 21554756.
- ^ a b Wheeler BM, Heimberg AM, Moy VN, Sperling EA, Holstein TW, Heber S, Peterson KJ (2009). «The deep evolution of metazoan microRNAs». Evolution & Development. 11 (1): 50–68. doi:10.1111/j.1525-142X.2008.00302.x. PMID 19196333. S2CID 14924603.
- ^ Pashkovskiy PP, Ryazansky SS (June 2013). «Biogenesis, evolution, and functions of plant microRNAs». Biochemistry. Biokhimiia. 78 (6): 627–37. doi:10.1134/S0006297913060084. PMID 23980889. S2CID 12025420.
- ^ a b Heimberg AM, Sempere LF, Moy VN, Donoghue PC, Peterson KJ (February 2008). «MicroRNAs and the advent of vertebrate morphological complexity». Proceedings of the National Academy of Sciences of the United States of America. 105 (8): 2946–50. Bibcode:2008PNAS..105.2946H. doi:10.1073/pnas.0712259105. PMC 2268565. PMID 18287013.
- ^ a b c Nozawa M, Miura S, Nei M (July 2010). «Origins and evolution of microRNA genes in Drosophila species». Genome Biology and Evolution. 2: 180–9. doi:10.1093/gbe/evq009. PMC 2942034. PMID 20624724.
- ^ Allen E, Xie Z, Gustafson AM, Sung GH, Spatafora JW, Carrington JC (December 2004). «Evolution of microRNA genes by inverted duplication of target gene sequences in Arabidopsis thaliana». Nature Genetics. 36 (12): 1282–90. doi:10.1038/ng1478. PMID 15565108. S2CID 11997028.
- ^ Warthmann N, Das S, Lanz C, Weigel D (May 2008). «Comparative analysis of the MIR319a microRNA locus in Arabidopsis and related Brassicaceae». Molecular Biology and Evolution. 25 (5): 892–902. doi:10.1093/molbev/msn029. PMID 18296705.
- ^ Fahlgren N, Jogdeo S, Kasschau KD, Sullivan CM, Chapman EJ, Laubinger S, Smith LM, Dasenko M, Givan SA, Weigel D, Carrington JC (April 2010). «MicroRNA gene evolution in Arabidopsis lyrata and Arabidopsis thaliana». The Plant Cell. 22 (4): 1074–89. doi:10.1105/tpc.110.073999. PMC 2879733. PMID 20407027.
- ^ Caravas J, Friedrich M (June 2010). «Of mites and millipedes: recent progress in resolving the base of the arthropod tree». BioEssays. 32 (6): 488–95. doi:10.1002/bies.201000005. PMID 20486135. S2CID 20548122.
- ^ Kenny NJ, Namigai EK, Marlétaz F, Hui JH, Shimeld SM (December 2015). «Draft genome assemblies and predicted microRNA complements of the intertidal lophotrochozoans Patella vulgata (Mollusca, Patellogastropoda) and Spirobranchus (Pomatoceros) lamarcki (Annelida, Serpulida)». Marine Genomics. 24 (2): 139–46. doi:10.1016/j.margen.2015.07.004. PMID 26319627.
- ^ Cock JM, Sterck L, Rouzé P, Scornet D, Allen AE, Amoutzias G, et al. (June 2010). «The Ectocarpus genome and the independent evolution of multicellularity in brown algae». Nature. 465 (7298): 617–21. Bibcode:2010Natur.465..617C. doi:10.1038/nature09016. PMID 20520714.
- ^ Cuperus JT, Fahlgren N, Carrington JC (February 2011). «Evolution and functional diversification of MIRNA genes». The Plant Cell. 23 (2): 431–42. doi:10.1105/tpc.110.082784. PMC 3077775. PMID 21317375.
- ^ Ryan JF, Pang K, Schnitzler CE, Nguyen AD, Moreland RT, Simmons DK, Koch BJ, Francis WR, Havlak P, Smith SA, Putnam NH, Haddock SH, Dunn CW, Wolfsberg TG, Mullikin JC, Martindale MQ, Baxevanis AD (December 2013). «The genome of the ctenophore Mnemiopsis leidyi and its implications for cell type evolution». Science. 342 (6164): 1242592. doi:10.1126/science.1242592. PMC 3920664. PMID 24337300.
- ^ Maxwell EK, Ryan JF, Schnitzler CE, Browne WE, Baxevanis AD (December 2012). «MicroRNAs and essential components of the microRNA processing machinery are not encoded in the genome of the ctenophore Mnemiopsis leidyi». BMC Genomics. 13 (1): 714. doi:10.1186/1471-2164-13-714. PMC 3563456. PMID 23256903.
- ^ Dimond PF (15 March 2010). «miRNAs’ Therapeutic Potential». Genetic Engineering & Biotechnology News. 30 (6): 1. Archived from the original on 19 July 2010. Retrieved 10 July 2010.
- ^ Tjaden B, Goodwin SS, Opdyke JA, Guillier M, Fu DX, Gottesman S, Storz G (2006). «Target prediction for small, noncoding RNAs in bacteria». Nucleic Acids Research. 34 (9): 2791–802. doi:10.1093/nar/gkl356. PMC 1464411. PMID 16717284.
- ^ Liu CG, Calin GA, Volinia S, Croce CM (2008). «MicroRNA expression profiling using microarrays». Nature Protocols. 3 (4): 563–78. doi:10.1038/nprot.2008.14. PMID 18388938. S2CID 2441105.
- ^ Chen C, Ridzon DA, Broomer AJ, Zhou Z, Lee DH, Nguyen JT, Barbisin M, Xu NL, Mahuvakar VR, Andersen MR, Lao KQ, Livak KJ, Guegler KJ (November 2005). «Real-time quantification of microRNAs by stem-loop RT-PCR». Nucleic Acids Research. 33 (20): e179. doi:10.1093/nar/gni178. PMC 1292995. PMID 16314309.
- ^ Shingara J, Keiger K, Shelton J, Laosinchai-Wolf W, Powers P, Conrad R, Brown D, Labourier E (September 2005). «An optimized isolation and labeling platform for accurate microRNA expression profiling». RNA. 11 (9): 1461–70. doi:10.1261/rna.2610405. PMC 1370829. PMID 16043497.
- ^ Buermans HP, Ariyurek Y, van Ommen G, den Dunnen JT, ‘t Hoen PA (December 2010). «New methods for next generation sequencing based microRNA expression profiling». BMC Genomics. 11: 716. doi:10.1186/1471-2164-11-716. PMC 3022920. PMID 21171994.
- ^ Kloosterman WP, Wienholds E, Ketting RF, Plasterk RH (2004). «Substrate requirements for let-7 function in the developing zebrafish embryo». Nucleic Acids Research. 32 (21): 6284–91. doi:10.1093/nar/gkh968. PMC 535676. PMID 15585662.
- ^ Flynt AS, Li N, Thatcher EJ, Solnica-Krezel L, Patton JG (February 2007). «Zebrafish miR-214 modulates Hedgehog signaling to specify muscle cell fate». Nature Genetics. 39 (2): 259–63. doi:10.1038/ng1953. PMC 3982799. PMID 17220889.
- ^ Meister G, Landthaler M, Dorsett Y, Tuschl T (March 2004). «Sequence-specific inhibition of microRNA- and siRNA-induced RNA silencing». RNA. 10 (3): 544–50. doi:10.1261/rna.5235104. PMC 1370948. PMID 14970398.
- ^ Kloosterman WP, Lagendijk AK, Ketting RF, Moulton JD, Plasterk RH (August 2007). «Targeted inhibition of miRNA maturation with morpholinos reveals a role for miR-375 in pancreatic islet development». PLOS Biology. 5 (8): e203. doi:10.1371/journal.pbio.0050203. PMC 1925136. PMID 17676975.
- ^ Gebert LF, Rebhan MA, Crivelli SE, Denzler R, Stoffel M, Hall J (January 2014). «Miravirsen (SPC3649) can inhibit the biogenesis of miR-122». Nucleic Acids Research. 42 (1): 609–21. doi:10.1093/nar/gkt852. PMC 3874169. PMID 24068553.
- ^ Choi WY, Giraldez AJ, Schier AF (October 2007). «Target protectors reveal dampening and balancing of Nodal agonist and antagonist by miR-430». Science. 318 (5848): 271–4. Bibcode:2007Sci…318..271C. doi:10.1126/science.1147535. PMID 17761850. S2CID 30461594.
- ^ You Y, Moreira BG, Behlke MA, Owczarzy R (May 2006). «Design of LNA probes that improve mismatch discrimination». Nucleic Acids Research. 34 (8): e60. doi:10.1093/nar/gkl175. PMC 1456327. PMID 16670427.
- ^ Lagendijk AK, Moulton JD, Bakkers J (June 2012). «Revealing details: whole mount microRNA in situ hybridization protocol for zebrafish embryos and adult tissues». Biology Open. 1 (6): 566–9. doi:10.1242/bio.2012810. PMC 3509442. PMID 23213449.
- ^ Kaur H, Arora A, Wengel J, Maiti S (June 2006). «Thermodynamic, counterion, and hydration effects for the incorporation of locked nucleic acid nucleotides into DNA duplexes». Biochemistry. 45 (23): 7347–55. doi:10.1021/bi060307w. PMID 16752924.
- ^ Nielsen JA, Lau P, Maric D, Barker JL, Hudson LD (August 2009). «Integrating microRNA and mRNA expression profiles of neuronal progenitors to identify regulatory networks underlying the onset of cortical neurogenesis». BMC Neuroscience. 10: 98. doi:10.1186/1471-2202-10-98. PMC 2736963. PMID 19689821.
- ^ Grimson A, Farh KK, Johnston WK, Garrett-Engele P, Lim LP, Bartel DP (July 2007). «MicroRNA targeting specificity in mammals: determinants beyond seed pairing». Molecular Cell. 27 (1): 91–105. doi:10.1016/j.molcel.2007.06.017. PMC 3800283. PMID 17612493.
- ^ Griffiths-Jones S, Saini HK, van Dongen S, Enright AJ (January 2008). «miRBase: tools for microRNA genomics». Nucleic Acids Research. 36 (Database issue): D154–8. doi:10.1093/nar/gkm952. PMC 2238936. PMID 17991681.
- ^ Nam S, Li M, Choi K, Balch C, Kim S, Nephew KP (July 2009). «MicroRNA and mRNA integrated analysis (MMIA): a web tool for examining biological functions of microRNA expression». Nucleic Acids Research. 37 (Web Server issue): W356–62. doi:10.1093/nar/gkp294. PMC 2703907. PMID 19420067.
- ^ Artmann S, Jung K, Bleckmann A, Beissbarth T (2012). Provero P (ed.). «Detection of simultaneous group effects in microRNA expression and related target gene sets». PLOS ONE. 7 (6): e38365. Bibcode:2012PLoSO…738365A. doi:10.1371/journal.pone.0038365. PMC 3378551. PMID 22723856.
- ^ Jiang Q, Wang Y, Hao Y, Juan L, Teng M, Zhang X, Li M, Wang G, Liu Y (January 2009). «miR2Disease: a manually curated database for microRNA deregulation in human disease». Nucleic Acids Research. 37. 37 (Database issue): D98–104. doi:10.1093/nar/gkn714. PMC 2686559. PMID 18927107.
- ^ Mencía A, Modamio-Høybjør S, Redshaw N, Morín M, Mayo-Merino F, Olavarrieta L, Aguirre LA, del Castillo I, Steel KP, Dalmay T, Moreno F, Moreno-Pelayo MA (May 2009). «Mutations in the seed region of human miR-96 are responsible for nonsyndromic progressive hearing loss». Nature Genetics. 41 (5): 609–13. doi:10.1038/ng.355. PMID 19363479. S2CID 11113852.
- ^ Hughes AE, Bradley DT, Campbell M, Lechner J, Dash DP, Simpson DA, Willoughby CE (November 2011). «Mutation altering the miR-184 seed region causes familial keratoconus with cataract». American Journal of Human Genetics. 89 (5): 628–33. doi:10.1016/j.ajhg.2011.09.014. PMC 3213395. PMID 21996275.
- ^ de Pontual L, Yao E, Callier P, Faivre L, Drouin V, Cariou S, Van Haeringen A, Geneviève D, Goldenberg A, Oufadem M, Manouvrier S, Munnich A, Vidigal JA, Vekemans M, Lyonnet S, Henrion-Caude A, Ventura A, Amiel J (September 2011). «Germline deletion of the miR-17∼92 cluster causes skeletal and growth defects in humans». Nature Genetics. 43 (10): 1026–30. doi:10.1038/ng.915. PMC 3184212. PMID 21892160.
- ^ Calin GA, Liu CG, Sevignani C, Ferracin M, Felli N, Dumitru CD, Shimizu M, Cimmino A, Zupo S, Dono M, Dell’Aquila ML, Alder H, Rassenti L, Kipps TJ, Bullrich F, Negrini M, Croce CM (August 2004). «MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias». Proc Natl Acad Sci U S A. 101 (32): 11755–60. Bibcode:2004PNAS..10111755C. doi:10.1073/pnas.0404432101. PMC 511048. PMID 15284443.
- ^ Velle, Angelo; Pesenti, Chiara; Grassi, Tommaso; Beltrame, Luca; Martini, Paolo; Jaconi, Marta; Agostinis, Federico; Calura, Enrica; Katsaros, Dionyssios; Borella, Fulvio; Fruscio, Robert; D’Incalci, Maurizio; Marchini, Sergio; Romualdi, Chiara (17 January 2023). «A comprehensive investigation of histotype‐specific microRNA and their variants in Stage I epithelial ovarian cancers». International Journal of Cancer: ijc.34408. doi:10.1002/ijc.34408. ISSN 0020-7136. PMID 36541726. S2CID 255034585.
- ^ Kyriakidis I, Kyriakidis K, Tsezou A (August 2022). «MicroRNAs and the Diagnosis of Childhood Acute Lymphoblastic Leukemia: Systematic Review, Meta-Analysis and Re-Analysis with Novel Small RNA-Seq Tools». Cancers. 14 (16): 3976. doi:10.3390/cancers14163976. PMC 9406077. PMID 36010971.
- ^ Võsa U, Vooder T, Kolde R, Fischer K, Välk K, Tõnisson N, Roosipuu R, Vilo J, Metspalu A, Annilo T (October 2011). «Identification of miR-374a as a prognostic marker for survival in patients with early-stage nonsmall cell lung cancer». Genes, Chromosomes & Cancer. 50 (10): 812–22. doi:10.1002/gcc.20902. PMID 21748820. S2CID 9746594.
- ^ Akçakaya P, Ekelund S, Kolosenko I, Caramuta S, Ozata DM, Xie H, Lindforss U, Olivecrona H, Lui WO (August 2011). «miR-185 and miR-133b deregulation is associated with overall survival and metastasis in colorectal cancer». International Journal of Oncology. 39 (2): 311–8. doi:10.3892/ijo.2011.1043. PMID 21573504.
- ^ a b Eyking A, Reis H, Frank M, Gerken G, Schmid KW, Cario E (2016). «MiR-205 and MiR-373 Are Associated with Aggressive Human Mucinous Colorectal Cancer». PLOS ONE. 11 (6): e0156871. Bibcode:2016PLoSO..1156871E. doi:10.1371/journal.pone.0156871. PMC 4894642. PMID 27271572.
- ^ MicroRNA-21 promotes hepatocellular carcinoma HepG2 cell proliferation through repression of mitogen-activated protein kinase-kinase 3. Guangxian Xu et al., 2013
- ^ Jones K, Nourse JP, Keane C, Bhatnagar A, Gandhi MK (January 2014). «Plasma microRNA are disease response biomarkers in classical Hodgkin lymphoma». Clinical Cancer Research. 20 (1): 253–64. doi:10.1158/1078-0432.CCR-13-1024. PMID 24222179.
- ^ Hosseinahli N, Aghapour M, Duijf PH, Baradaran B (August 2018). «Treating cancer with microRNA replacement therapy: A literature review». Journal of Cellular Physiology. 233 (8): 5574–5588. doi:10.1002/jcp.26514. PMID 29521426. S2CID 3766576.
- ^ Liu G, Sun Y, Ji P, Li X, Cogdell D, Yang D, Parker Kerrigan BC, Shmulevich I, Chen K, Sood AK, Xue F, Zhang W (July 2014). «MiR-506 suppresses proliferation and induces senescence by directly targeting the CDK4/6-FOXM1 axis in ovarian cancer». The Journal of Pathology. 233 (3): 308–18. doi:10.1002/path.4348. PMC 4144705. PMID 24604117.
- ^ Wen SY, Lin Y, Yu YQ, Cao SJ, Zhang R, Yang XM, Li J, Zhang YL, Wang YH, Ma MZ, Sun WW, Lou XL, Wang JH, Teng YC, Zhang ZG (February 2015). «miR-506 acts as a tumor suppressor by directly targeting the hedgehog pathway transcription factor Gli3 in human cervical cancer». Oncogene. 34 (6): 717–25. doi:10.1038/onc.2014.9. PMID 24608427. S2CID 20603801.
- ^ Peking University (13 September 2022). «Scientists Develop a New, Powerful Cancer-Fighting Weapon». Cell. SciTech Daily. 185 (11): 1888–1904.e24. doi:10.1016/j.cell.2022.04.030. PMID 35623329. S2CID 249070106. Retrieved 15 September 2022.
- ^ Loeb KR, Loeb LA (March 2000). «Significance of multiple mutations in cancer». Carcinogenesis. 21 (3): 379–385. doi:10.1093/carcin/21.3.379. PMID 10688858.
- ^ Lodish H, Berk A, Kaiser CA, Krieger M, Bretscher A, Ploegh H, Amon A, Martin KC (2016). Molecular Cell Biology (8th ed.). New York: W. H. Freeman and Company. p. 203. ISBN 978-1-4641-8339-3.
- ^ Hu H, Gatti RA (June 2011). «MicroRNAs: new players in the DNA damage response». Journal of Molecular Cell Biology. 3 (3): 151–158. doi:10.1093/jmcb/mjq042. PMC 3104011. PMID 21183529.
- ^ Jasperson KW, Tuohy TM, Neklason DW, Burt RW (June 2010). «Hereditary and familial colon cancer». Gastroenterology. 138 (6): 2044–58. doi:10.1053/j.gastro.2010.01.054. PMC 3057468. PMID 20420945.
- ^ Truninger K, Menigatti M, Luz J, Russell A, Haider R, Gebbers JO, Bannwart F, Yurtsever H, Neuweiler J, Riehle HM, Cattaruzza MS, Heinimann K, Schär P, Jiricny J, Marra G (May 2005). «Immunohistochemical analysis reveals high frequency of PMS2 defects in colorectal cancer». Gastroenterology. 128 (5): 1160–71. doi:10.1053/j.gastro.2005.01.056. PMID 15887099.
- ^ Valeri N, Gasparini P, Fabbri M, Braconi C, Veronese A, Lovat F, Adair B, Vannini I, Fanini F, Bottoni A, Costinean S, Sandhu SK, Nuovo GJ, Alder H, Gafa R, Calore F, Ferracin M, Lanza G, Volinia S, Negrini M, McIlhatton MA, Amadori D, Fishel R, Croce CM (April 2010). «Modulation of mismatch repair and genomic stability by miR-155». Proceedings of the National Academy of Sciences of the United States of America. 107 (15): 6982–7. Bibcode:2010PNAS..107.6982V. doi:10.1073/pnas.1002472107. PMC 2872463. PMID 20351277.
- ^ a b c Zhang W, Zhang J, Hoadley K, Kushwaha D, Ramakrishnan V, Li S, Kang C, You Y, Jiang C, Song SW, Jiang T, Chen CC (June 2012). «miR-181d: a predictive glioblastoma biomarker that downregulates MGMT expression». Neuro-Oncology. 14 (6): 712–9. doi:10.1093/neuonc/nos089. PMC 3367855. PMID 22570426.
- ^ Spiegl-Kreinecker S, Pirker C, Filipits M, Lötsch D, Buchroithner J, Pichler J, Silye R, Weis S, Micksche M, Fischer J, Berger W (January 2010). «O6-Methylguanine DNA methyltransferase protein expression in tumor cells predicts outcome of temozolomide therapy in glioblastoma patients». Neuro-Oncology. 12 (1): 28–36. doi:10.1093/neuonc/nop003. PMC 2940563. PMID 20150365.
- ^ Sgarra R, Rustighi A, Tessari MA, Di Bernardo J, Altamura S, Fusco A, Manfioletti G, Giancotti V (September 2004). «Nuclear phosphoproteins HMGA and their relationship with chromatin structure and cancer». FEBS Letters. 574 (1–3): 1–8. doi:10.1016/j.febslet.2004.08.013. PMID 15358530. S2CID 28903539.
- ^ Xu Y, Sumter TF, Bhattacharya R, Tesfaye A, Fuchs EJ, Wood LJ, Huso DL, Resar LM (May 2004). «The HMG-I oncogene causes highly penetrant, aggressive lymphoid malignancy in transgenic mice and is overexpressed in human leukemia». Cancer Research. 64 (10): 3371–5. doi:10.1158/0008-5472.CAN-04-0044. PMID 15150086.
- ^ Borrmann L, Schwanbeck R, Heyduk T, Seebeck B, Rogalla P, Bullerdiek J, Wisniewski JR (December 2003). «High mobility group A2 protein and its derivatives bind a specific region of the promoter of DNA repair gene ERCC1 and modulate its activity». Nucleic Acids Research. 31 (23): 6841–51. doi:10.1093/nar/gkg884. PMC 290254. PMID 14627817.
- ^ Facista A, Nguyen H, Lewis C, Prasad AR, Ramsey L, Zaitlin B, Nfonsam V, Krouse RS, Bernstein H, Payne CM, Stern S, Oatman N, Banerjee B, Bernstein C (April 2012). «Deficient expression of DNA repair enzymes in early progression to sporadic colon cancer». Genome Integrity. 3 (1): 3. doi:10.1186/2041-9414-3-3. PMC 3351028. PMID 22494821.
- ^ Gibert B, Delloye-Bourgeois C, Gattolliat CH, Meurette O, Le Guernevel S, Fombonne J, et al. (November 2014). «Regulation by miR181 family of the dependence receptor CDON tumor suppressive activity in neuroblastoma». Journal of the National Cancer Institute. 106 (11). doi:10.1093/jnci/dju318. PMID 25313246.
- ^ Chen JF, Murchison EP, Tang R, Callis TE, Tatsuguchi M, Deng Z, Rojas M, Hammond SM, Schneider MD, Selzman CH, Meissner G, Patterson C, Hannon GJ, Wang DZ (February 2008). «Targeted deletion of Dicer in the heart leads to dilated cardiomyopathy and heart failure». Proceedings of the National Academy of Sciences of the United States of America. 105 (6): 2111–6. Bibcode:2008PNAS..105.2111C. doi:10.1073/pnas.0710228105. PMC 2542870. PMID 18256189.
- ^ a b Zhao Y, Ransom JF, Li A, Vedantham V, von Drehle M, Muth AN, Tsuchihashi T, McManus MT, Schwartz RJ, Srivastava D (April 2007). «Dysregulation of cardiogenesis, cardiac conduction, and cell cycle in mice lacking miRNA-1-2». Cell. 129 (2): 303–17. doi:10.1016/j.cell.2007.03.030. PMID 17397913.
- ^ Thum T, Galuppo P, Wolf C, Fiedler J, Kneitz S, van Laake LW, Doevendans PA, Mummery CL, Borlak J, Haverich A, Gross C, Engelhardt S, Ertl G, Bauersachs J (July 2007). «MicroRNAs in the human heart: a clue to fetal gene reprogramming in heart failure». Circulation. 116 (3): 258–67. doi:10.1161/CIRCULATIONAHA.107.687947. PMID 17606841.
- ^ van Rooij E, Sutherland LB, Liu N, Williams AH, McAnally J, Gerard RD, Richardson JA, Olson EN (November 2006). «A signature pattern of stress-responsive microRNAs that can evoke cardiac hypertrophy and heart failure». Proceedings of the National Academy of Sciences of the United States of America. 103 (48): 18255–60. Bibcode:2006PNAS..10318255V. doi:10.1073/pnas.0608791103. PMC 1838739. PMID 17108080.
- ^ Tatsuguchi M, Seok HY, Callis TE, Thomson JM, Chen JF, Newman M, Rojas M, Hammond SM, Wang DZ (June 2007). «Expression of microRNAs is dynamically regulated during cardiomyocyte hypertrophy». Journal of Molecular and Cellular Cardiology. 42 (6): 1137–41. doi:10.1016/j.yjmcc.2007.04.004. PMC 1934409. PMID 17498736.
- ^ Zhao Y, Samal E, Srivastava D (July 2005). «Serum response factor regulates a muscle-specific microRNA that targets Hand2 during cardiogenesis». Nature. 436 (7048): 214–20. Bibcode:2005Natur.436..214Z. doi:10.1038/nature03817. PMID 15951802. S2CID 4340449.
- ^ Xiao J, Luo X, Lin H, Zhang Y, Lu Y, Wang N, Zhang Y, Yang B, Wang Z (April 2007). «MicroRNA miR-133 represses HERG K+ channel expression contributing to QT prolongation in diabetic hearts». The Journal of Biological Chemistry. 282 (17): 12363–7. doi:10.1074/jbc.C700015200. PMID 17344217.
- ^ Yang B, Lin H, Xiao J, Lu Y, Luo X, Li B, Zhang Y, Xu C, Bai Y, Wang H, Chen G, Wang Z (April 2007). «The muscle-specific microRNA miR-1 regulates cardiac arrhythmogenic potential by targeting GJA1 and KCNJ2». Nature Medicine. 13 (4): 486–91. doi:10.1038/nm1569. PMID 17401374. S2CID 1935811.
- ^ Carè A, Catalucci D, Felicetti F, Bonci D, Addario A, Gallo P, Bang ML, Segnalini P, Gu Y, Dalton ND, Elia L, Latronico MV, Høydal M, Autore C, Russo MA, Dorn GW, Ellingsen O, Ruiz-Lozano P, Peterson KL, Croce CM, Peschle C, Condorelli G (May 2007). «MicroRNA-133 controls cardiac hypertrophy». Nature Medicine. 13 (5): 613–8. doi:10.1038/nm1582. PMID 17468766. S2CID 10097893.
- ^ van Rooij E, Sutherland LB, Qi X, Richardson JA, Hill J, Olson EN (April 2007). «Control of stress-dependent cardiac growth and gene expression by a microRNA». Science. 316 (5824): 575–9. Bibcode:2007Sci…316..575V. doi:10.1126/science.1139089. PMID 17379774. S2CID 1927839.
- ^ Keller T, Boeckel JN, Groß S, Klotsche J, Palapies L, Leistner D, et al. (July 2017). «Improved risk stratification in prevention by use of a panel of selected circulating microRNAs». Scientific Reports. 7 (1): 4511. Bibcode:2017NatSR…7.4511K. doi:10.1038/s41598-017-04040-w. PMC 5495799. PMID 28674420.
- ^ Insull W (January 2009). «The pathology of atherosclerosis: plaque development and plaque responses to medical treatment». The American Journal of Medicine. 122 (1 Suppl): S3–S14. doi:10.1016/j.amjmed.2008.10.013. PMID 19110086.
- ^ a b c d e f g h i j k l m n o p q Son DJ, Kumar S, Takabe W, Kim CW, Ni CW, Alberts-Grill N, Jang IH, Kim S, Kim W, Won Kang S, Baker AH, Woong Seo J, Ferrara KW, Jo H (2013). «The atypical mechanosensitive microRNA-712 derived from pre-ribosomal RNA induces endothelial inflammation and atherosclerosis». Nature Communications. 4: 3000. Bibcode:2013NatCo…4.3000S. doi:10.1038/ncomms4000. PMC 3923891. PMID 24346612.
- ^ a b Basu R, Fan D, Kandalam V, Lee J, Das SK, Wang X, Baldwin TA, Oudit GY, Kassiri Z (December 2012). «Loss of Timp3 gene leads to abdominal aortic aneurysm formation in response to angiotensin II». The Journal of Biological Chemistry. 287 (53): 44083–96. doi:10.1074/jbc.M112.425652. PMC 3531724. PMID 23144462.
- ^ Libby P (2002). «Inflammation in atherosclerosis». Nature. 420 (6917): 868–74. Bibcode:2002Natur.420..868L. doi:10.1038/nature01323. PMID 12490960. S2CID 407449.
- ^ a b Phua YL, Chu JY, Marrone AK, Bodnar AJ, Sims-Lucas S, Ho J (October 2015). «Renal stromal miRNAs are required for normal nephrogenesis and glomerular mesangial survival». Physiological Reports. 3 (10): e12537. doi:10.14814/phy2.12537. PMC 4632944. PMID 26438731.
- ^ Maes OC, Chertkow HM, Wang E, Schipper HM (May 2009). «MicroRNA: Implications for Alzheimer Disease and other Human CNS Disorders». Current Genomics. 10 (3): 154–68. doi:10.2174/138920209788185252. PMC 2705849. PMID 19881909.
- ^ Amin ND, Bai G, Klug JR, Bonanomi D, Pankratz MT, Gifford WD, Hinckley CA, Sternfeld MJ, Driscoll SP, Dominguez B, Lee KF, Jin X, Pfaff SL (December 2015). «Loss of motoneuron-specific microRNA-218 causes systemic neuromuscular failure». Science. 350 (6267): 1525–9. Bibcode:2015Sci…350.1525A. doi:10.1126/science.aad2509. PMC 4913787. PMID 26680198.
- ^ Schratt G (December 2009). «microRNAs at the synapse». Nature Reviews. Neuroscience. 10 (12): 842–9. doi:10.1038/nrn2763. PMID 19888283. S2CID 3507952.
- ^ Cuellar TL, Davis TH, Nelson PT, Loeb GB, Harfe BD, Ullian E, McManus MT (April 2008). «Dicer loss in striatal neurons produces behavioral and neuroanatomical phenotypes in the absence of neurodegeneration». Proceedings of the National Academy of Sciences. 105 (14): 5614–19. Bibcode:2008PNAS..105.5614C. doi:10.1073/pnas.0801689105. PMC 2291142. PMID 18385371.
- ^ Hébert SS, Papadopoulou AS, Smith P, Galas M, Planel E, Silahtaroglu AN, Sergeant N, Buée L, De Strooper B (October 2010). «Genetic ablation of Dicer in adult forebrain neurons results in abnormal tau hyperphosphorylation and neurodegeneration». Human Molecular Genetics. 19 (20): 3959–69. doi:10.1093/hmg/ddq311. PMID 20660113.
- ^ Hosseinian S, Arefian A, Rakhsh-Khorshid H (2020). «A meta-analysis of gene expression data highlights synaptic dysfunction in the hippocampus of brains with Alzheimer’s disease». Scientific Reports. 10 (1): 8384. Bibcode:2020NatSR..10.8384H. doi:10.1038/s41598-020-64452-z. PMC 7239885. PMID 32433480.
- ^ Hommers LG, Domschke K, Deckert J (January 2015). «Heterogeneity and individuality: microRNAs in mental disorders». Journal of Neural Transmission. 122 (1): 79–97. doi:10.1007/s00702-014-1338-4. PMID 25395183. S2CID 25088900.
- ^ Feng J, Sun G, Yan J, Noltner K, Li W, Buzin CH, Longmate J, Heston LL, Rossi J, Sommer SS (July 2009). Reif A (ed.). «Evidence for X-chromosomal schizophrenia associated with microRNA alterations». PLOS ONE. 4 (7): e6121. Bibcode:2009PLoSO…4.6121F. doi:10.1371/journal.pone.0006121. PMC 2699475. PMID 19568434.
- ^ Beveridge NJ, Gardiner E, Carroll AP, Tooney PA, Cairns MJ (December 2010). «Schizophrenia is associated with an increase in cortical microRNA biogenesis». Molecular Psychiatry. 15 (12): 1176–89. doi:10.1038/mp.2009.84. PMC 2990188. PMID 19721432.
- ^ «Stroke Facts | cdc.gov». www.cdc.gov. 15 March 2019. Retrieved 5 December 2019.
- ^ Rink C, Khanna S (May 2011). «MicroRNA in ischemic stroke etiology and pathology». Physiological Genomics. 43 (10): 521–528. doi:10.1152/physiolgenomics.00158.2010. PMC 3110894. PMID 20841499.
- ^ Ouyang YB, Stary CM, Yang GY, Giffard R (January 2013). «microRNAs: innovative targets for cerebral ischemia and stroke». Current Drug Targets. 14 (1): 90–101. doi:10.2174/138945013804806424. PMC 3673881. PMID 23170800.
- ^ a b c Lewohl JM, Nunez YO, Dodd PR, Tiwari GR, Harris RA, Mayfield RD (November 2011). «Up-regulation of microRNAs in brain of human alcoholics». Alcoholism: Clinical and Experimental Research. 35 (11): 1928–37. doi:10.1111/j.1530-0277.2011.01544.x. PMC 3170679. PMID 21651580.
- ^ Tapocik JD, Solomon M, Flanigan M, Meinhardt M, Barbier E, Schank JR, Schwandt M, Sommer WH, Heilig M (June 2013). «Coordinated dysregulation of mRNAs and microRNAs in the rat medial prefrontal cortex following a history of alcohol dependence». The Pharmacogenomics Journal. 13 (3): 286–96. doi:10.1038/tpj.2012.17. PMC 3546132. PMID 22614244.
- ^ Gorini G, Nunez YO, Mayfield RD (2013). «Integration of miRNA and protein profiling reveals coordinated neuroadaptations in the alcohol-dependent mouse brain». PLOS ONE. 8 (12): e82565. Bibcode:2013PLoSO…882565G. doi:10.1371/journal.pone.0082565. PMC 3865091. PMID 24358208.
- ^ Tapocik JD, Barbier E, Flanigan M, Solomon M, Pincus A, Pilling A, Sun H, Schank JR, King C, Heilig M (March 2014). «microRNA-206 in rat medial prefrontal cortex regulates BDNF expression and alcohol drinking». The Journal of Neuroscience. 34 (13): 4581–8. doi:10.1523/JNEUROSCI.0445-14.2014. PMC 3965783. PMID 24672003.
- ^ Lippai D, Bala S, Csak T, Kurt-Jones EA, Szabo G (2013). «Chronic alcohol-induced microRNA-155 contributes to neuroinflammation in a TLR4-dependent manner in mice». PLOS ONE. 8 (8): e70945. Bibcode:2013PLoSO…870945L. doi:10.1371/journal.pone.0070945. PMC 3739772. PMID 23951048.
- ^ a b Li J, Li J, Liu X, Qin S, Guan Y, Liu Y, Cheng Y, Chen X, Li W, Wang S, Xiong M, Kuzhikandathil EV, Ye JH, Zhang C (September 2013). «MicroRNA expression profile and functional analysis reveal that miR-382 is a critical novel gene of alcohol addiction». EMBO Molecular Medicine. 5 (9): 1402–14. doi:10.1002/emmm.201201900. PMC 3799494. PMID 23873704.
- ^ Romao JM, Jin W, Dodson MV, Hausman GJ, Moore SS, Guan LL (September 2011). «MicroRNA regulation in mammalian adipogenesis». Experimental Biology and Medicine. 236 (9): 997–1004. doi:10.1258/ebm.2011.011101. PMID 21844119. S2CID 30646787.
- ^ Skårn M, Namløs HM, Noordhuis P, Wang MY, Meza-Zepeda LA, Myklebost O (April 2012). «Adipocyte differentiation of human bone marrow-derived stromal cells is modulated by microRNA-155, microRNA-221, and microRNA-222». Stem Cells and Development. 21 (6): 873–83. doi:10.1089/scd.2010.0503. hdl:10852/40423. PMID 21756067.
- ^ Zuo Y, Qiang L, Farmer SR (March 2006). «Activation of CCAAT/enhancer-binding protein (C/EBP) alpha expression by C/EBP beta during adipogenesis requires a peroxisome proliferator-activated receptor-gamma-associated repression of HDAC1 at the C/ebp alpha gene promoter». The Journal of Biological Chemistry. 281 (12): 7960–7. doi:10.1074/jbc.M510682200. PMID 16431920.
- ^ Jun-Hao ET, Gupta RR, Shyh-Chang N (March 2016). «Lin28 and let-7 in the Metabolic Physiology of Aging». Trends in Endocrinology and Metabolism. 27 (3): 132–141. doi:10.1016/j.tem.2015.12.006. PMID 26811207. S2CID 3614126.
- ^ Zhu H, Shyh-Chang N, Segrè AV, Shinoda G, Shah SP, Einhorn WS, Takeuchi A, Engreitz JM, Hagan JP, Kharas MG, Urbach A, Thornton JE, Triboulet R, Gregory RI, Altshuler D, Daley GQ (September 2011). «The Lin28/let-7 axis regulates glucose metabolism». Cell. 147 (1): 81–94. doi:10.1016/j.cell.2011.08.033. PMC 3353524. PMID 21962509.
- ^ Frost RJ, Olson EN (December 2011). «Control of glucose homeostasis and insulin sensitivity by the Let-7 family of microRNAs». Proceedings of the National Academy of Sciences of the United States of America. 108 (52): 21075–80. Bibcode:2011PNAS..10821075F. doi:10.1073/pnas.1118922109. PMC 3248488. PMID 22160727.
- ^ Teruel-Montoya R, Rosendaal FR, Martínez C (February 2015). «MicroRNAs in hemostasis». Journal of Thrombosis and Haemostasis. 13 (2): 170–181. doi:10.1111/jth.12788. PMID 25400249.
- ^ a b Nourse J, Braun J, Lackner K, Hüttelmaier S, Danckwardt S (November 2018). «Large-scale identification of functional microRNA targeting reveals cooperative regulation of the hemostatic system». Journal of Thrombosis and Haemostasis. 16 (11): 2233–2245. doi:10.1111/jth.14290. PMID 30207063.
- ^ Nourse J, Danckwardt S (February 2021). «A novel rationale for targeting FXI: Insights from the hemostatic microRNA targetome for emerging anticoagulant strategies». Pharmacology & Therapeutics. 218: 107676. doi:10.1016/j.pharmthera.2020.107676. PMID 32898547.
- ^ Berardi E, Pues M, Thorrez L, Sampaolesi M (October 2012). «miRNAs in ESC differentiation». American Journal of Physiology. Heart and Circulatory Physiology. 303 (8): H931–H939. doi:10.1152/ajpheart.00338.2012. PMID 22886416. S2CID 6402014.
- ^ a b Borchers A, Pieler T (November 2010). «Programming pluripotent precursor cells derived from Xenopus embryos to generate specific tissues and organs». Genes. 1 (3): 413–426. doi:10.3390/ijms232314755. PMC 9740008. PMID 36499082.
- ^ Curaba J, Spriggs A, Taylor J, Li Z, Helliwell C (July 2012). «miRNA regulation in the early development of barley seed». BMC Plant Biology. 12 (1): 120. doi:10.1186/1471-2229-12-120. PMC 3443071. PMID 22838835.
- ^ Choudhary A, Kumar A, Kaur H, Kaur N (May 2021). «MiRNA: the taskmaster of plant world». Biologia. 76 (5): 1551–1567. doi:10.1007/s11756-021-00720-1. ISSN 1336-9563. S2CID 233685660.
- ^ Waheed S, Zeng L (March 2020). «The Critical Role of miRNAs in Regulation of Flowering Time and Flower Development». Genes. 11 (3): 319. doi:10.3390/genes11030319. PMC 7140873. PMID 32192095.
- ^ Wong CE, Zhao YT, Wang XJ, Croft L, Wang ZH, Haerizadeh F, et al. (May 2011). «MicroRNAs in the shoot apical meristem of soybean». Journal of Experimental Botany. 62 (8): 2495–2506. doi:10.1093/jxb/erq437. hdl:10536/DRO/DU:30106047. PMID 21504877.
- ^ Qureshi A, Thakur N, Monga I, Thakur A, Kumar M (1 January 2014). «VIRmiRNA: a comprehensive resource for experimentally validated viral miRNAs and their targets». Database. 2014: bau103. doi:10.1093/database/bau103. PMC 4224276. PMID 25380780.
- ^ Kumar M. «VIRmiRNA». Resource for experimental viral miRNA and their targets. Bioinformatics center, CSIR-IMTECH.
- ^ Tuddenham L, Jung JS, Chane-Woon-Ming B, Dölken L, Pfeffer S (February 2012). «Small RNA deep sequencing identifies microRNAs and other small noncoding RNAs from human herpesvirus 6B». Journal of Virology. 86 (3): 1638–49. doi:10.1128/JVI.05911-11. PMC 3264354. PMID 22114334.
- ^ Zheng H, Fu R, Wang JT, Liu Q, Chen H, Jiang SW (April 2013). «Advances in the techniques for the prediction of microRNA targets». International Journal of Molecular Sciences. 14 (4): 8179–87. doi:10.3390/ijms14048179. PMC 3645737. PMID 23591837.
- ^ Agarwal V, Bell GW, Nam JW, Bartel DP (August 2015). «Predicting effective microRNA target sites in mammalian mRNAs». eLife. 4: e05005. doi:10.7554/eLife.05005. PMC 4532895. PMID 26267216.
Further reading[edit]
- miRNA definition and classification: Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T (March 2003). «A uniform system for microRNA annotation». RNA. 9 (3): 277–9. doi:10.1261/rna.2183803. PMC 1370393. PMID 12592000.
- Science review of small RNA: Baulcombe D (September 2002). «DNA events. An RNA microcosm». Science. 297 (5589): 2002–3. doi:10.1126/science.1077906. PMID 12242426. S2CID 82531727.
- Discovery of lin-4, the first miRNA to be discovered: Lee RC, Feinbaum RL, Ambros V (December 1993). «The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14». Cell. 75 (5): 843–54. doi:10.1016/0092-8674(93)90529-Y. PMID 8252621.
External links[edit]
- The miRBase database
- miRTarBase, the experimentally validated microRNA-target interactions database.
- semirna, Web application to search for microRNAs in a plant genome.
- ONCO.IO: Integrative resource for microRNA and transcription factors analysis in cancer.
- MirOB: MicroRNA targets database and data analysis and dataviz tool.
- ChIPBase database: An open access database for decoding the transcription factors that were involved in or affected the transcription of microRNAs from ChIP-seq data.
- An animated video of the microRNA biogenesis process.
- miRNA modulation reagents to enable up-regulation or suppression of endogenous mature microRNA function
МикроРНК (англ. microRNA, miRNA) — класс некодирующих РНК, которые имеют длину около 22 нуклеотидов. Эти РНК играют важную роль в регуляции трансляции и деградации мРНК. Регуляция осуществляется путем комплементарного связывания микроРНК с частично комплементарными сайтами в нетранслируемых участках (UTRs) мРНК (мишенями). Также имеются указания на возможность взаимодействия микроРНК непосредственно с ДНК генов в процессе РНК-зависимого метилирования ДНК, которое является одним из ключевых механизмов репрессии генов, аллельного исключения и предотвращения активности транспозонов[1].
МикроРНК кодируются генами, первые из которых были обнаружены в 1993 году у Caenorhabditis elegans группой исследователей во главе с Виктором Амбросом. Эволюционно гены микроРНК являются производными мобильных элементов класса MITE (англ. miniature inverted-repeat transposable elements) и возникли из отдельных копий транспозонов. Транскрипты MITE благодаря наличию инвертированных повторов и формированию шпилек клетка распознавала и использовала для подавления активности остальных копий транспозонов. В настоящее время микроРНК обнаружены у животных и растений. Мишенями микроРНК является внушительное число генов — по меньшей мере треть генов генома. Ранее считалось, что микроРНК присущи лишь многоклеточным организмам, однако, наличие этой группы молекул обнаружено и у одноклеточных эукариот, а именно — у вида зелёных водорослей Chlamydomonas reinhardtii. Это может свидетельствовать о большем эволюционном возрасте микроРНК, чем ранее предполагалось.
Содержание
- 1 Процессинг микроРНК
- 2 Механизм действия микроРНК
- 3 Клиническое значение
- 4 Примечания
- 5 Внешние ссылки
Процессинг микроРНК
Показано, что у животных экспрессия микроРНК происходит в два этапа. Сначала с гена микроРНК транскрибируется протяженный первичный транскрипт, который может содержать в себе несколько копий микроРНК. Процессинг первичного транскрипта осуществляется в ядре, а продукт процессинга (зашпиленный предшественник) экспортируется в цитоплазму. В цитоплазме фермент Dicer (имеет свойства РНКазы III) вырезает из предшественника зрелую микроРНК.
Механизм действия микроРНК
Клиническое значение
Изменения в генах микроРНК могут играть роль в развитии ряда заболеваний. В некоторых исследованиях отмечается возможная роль микроРНК при шизофрении.[2]
МикроРНК miR-140 участвует в патогенезе остеоартрита, регулируя, экспрессию гена ADAMTS5. У мышей, не экспрессирующих miR-140, нарушена пролиферация хондроцитов, мыши имеют карликовый фенотип.
Примечания
- ↑ Галицкий В.А. (2008). «Гипотеза о механизме инициации малыми РНК метилирования ДНК de novo и аллельного исключения» (русский). Цитология 50(4): 277–286.
- ↑ Evidence for X-chromosomal schizophrenia associated with microRNA alterations. Feng J, Sun G, Yan J, Noltner K, Li W, Buzin CH, Longmate J, Heston LL, Rossi J, Sommer SS. PLoS One. 2009 Jul 1;4(7):e6121. PMID 19568434
Внешние ссылки
- Коллекция статей, посвященных микроРНК на сайте журнала Nature
- Обо всех РНК на свете, больших и малых (подробный популярный обзор РНК-интерференции) — Старокадомский П.
- Нейробиологи нашли «включатель» человеческой памяти. Происходящий в нейронах синтез белков, необходимый для формирования памяти, контролируются с помощью микроРНК
| |
|
|---|---|
| Азотистые основания |
Пурины (Аденин • Гуанин) • Пиримидины (Урацил • Тимин • Цитозин) |
| Нуклеозиды |
Аденозин • Гуанозин • Уридин • Тимидин • Цитидин |
| Нуклеотиды |
монофосфаты (АМФ • ГМФ • UMP • ЦМФ) • дифосфаты (АДФ • ГДФ • УДФ • ЦДФ) • трифосфаты (АТФ • ГТФ • УТФ • ЦТФ) • циклические (цАМФ • цГМФ • cADPR) |
| РНК |
мРНК • тРНК • рРНК • антисмысловые • gRNA • микро • некодирующие • piwi-interacting • shRNA • малые интерферирующие • малые ядерные • малые ядрышковые • тмРНК |
| ДНК |
кДНК • Геном • msDNA • Митохондриальная |
| Аналоги |
Гликоль-нуклеиновая кислота[ru] • Замкнутая нуклеиновая кислота[ru] • ПНК • ТНК • Морфолино |
| Типы векторов |
Фазмиды[ru] • Плазмиды • Фаг лямбда • Космиды[ru] • Фаг P1[ru] • Фосмиды[ru] • Искусственная бактериальная хромосома[ru] • Искусственная дрожжевая хромосома[ru] • Искусственная человеческая хромосома[ru] |
|
Основные группы биохимических молекул Аминокислоты · Пептиды · Белки · Углеводы · Нуклеотиды · Нуклеиновая кислота · Липиды · Терпены · Каротиноиды · Стероиды · Флавоноиды · Алкалоиды · Гликозиды · Иридоиды |
Схема действия микроРНК (миРНК) с мРНК
Примеры стволовых петель миРНК, зрелые миРНК показаны красным
МикроРНК (сокращенно микроРНК ) представляет собой небольшой одноцепочечный некодирующие РНК молекулы (содержащий около 22 нуклеотидов ) содержится в растениях, животных и некоторых вирусов, который функционирует в РНК глушителей и пост-транскрипционной регуляции экспрессии генов . miRNAs функционируют посредством спаривания оснований с комплементарными последовательностями в молекулах мРНК . В результате эти молекулы мРНК заглушаются одним или несколькими из следующих процессов: (1) расщепление цепи мРНК на две части, (2) дестабилизация мРНК за счет укорочения ее поли (A) хвоста и ( 3) менее эффективная трансляция мРНК в белки рибосомами .
miRNA напоминают малые интерферирующие РНК (siRNA) пути РНК-интерференции (RNAi) , за исключением того, что miRNA происходят из областей транскриптов РНК, которые складываются сами по себе, образуя короткие шпильки, тогда как siRNA происходят из более длинных областей двухцепочечной РНК . Геном человека может кодировать более 1900 микроРНК, хотя недавний анализ показывает , что число ближе к 2,300. Однако только около 500 человеческих микроРНК представляют настоящую miRNA в вручную созданной базе данных генов miRNA MirGeneDB .
miRNAs в изобилии во многих типах клеток млекопитающих и в виде циркулирующих вне клетки miRNA . Циркулирующие миРНК выделяются в жидкости организма, включая кровь и спинномозговую жидкость, и потенциально могут быть доступны в качестве биомаркеров при ряде заболеваний. MiRNA, по-видимому, нацелены на около 60% генов человека и других млекопитающих. Многие miRNA эволюционно консервативны, что означает, что они выполняют важные биологические функции. Например, 90 семейств miRNA были законсервированы, по крайней мере, со времен общего предка млекопитающих и рыб, и большинство из этих консервативных miRNAs имеют важные функции, как показали исследования, в которых были выбиты гены для одного или нескольких членов семейства. у мышей.
История
Первая миРНК была открыта в начале 1990-х годов. Однако miRNA не были признаны отдельным классом биологических регуляторов до начала 2000-х годов. Исследования miRNA выявили различные наборы miRNA, экспрессируемые в разных типах клеток и тканях, и множественные роли miRNA в развитии растений и животных, а также во многих других биологических процессах. Аберрантная экспрессия miRNA вовлечена в болезненные состояния. Терапия на основе MiRNA находится на стадии исследования.
Первая miRNA была открыта в 1993 году группой под руководством Амброса, в которую входили Ли и Фейнбаум. Однако для дополнительного понимания его способа действий потребовались одновременно опубликованные работы команды Рувкуна , включая Вайтмана и Ха. Эти группы опубликовали несколько статей о гене lin-4 , который, как известно, контролирует время личиночного развития C. elegans путем репрессии гена lin-14 . Когда Ли и др. изолировали miRNA lin-4 , они обнаружили, что вместо получения мРНК, кодирующей белок, она продуцировала короткие некодирующие РНК , одна из которых была ~ 22-нуклеотидной РНК, которая содержала последовательности, частично комплементарные множественным последовательностям в 3 ‘UTR от лин-14 мРНК. Эта комплементарность была предложена для ингибирования трансляции мРНК lin-14 в белок LIN-14. В то время считалось, что малая РНК lin-4 является идиосинкразией нематод .
В 2000 году была охарактеризована вторая малая РНК: let-7 RNA, которая репрессирует lin-41, чтобы способствовать более позднему переходу в развитии у C. elegans . Было обнаружено, что РНК let-7 консервативна у многих видов, что привело к предположению, что РНК let-7 и дополнительные «малые временные РНК» могут регулировать время развития у различных животных, включая человека.
Годом позже было обнаружено , что lin-4 и let-7 РНК являются частью большого класса малых РНК, присутствующих в клетках C. elegans , дрозофилы и человека. Многие РНК этого класса напоминают РНК lin-4 и let-7 , за исключением того, что паттерны их экспрессии обычно несовместимы с ролью в регуляции времени развития. Это предполагает, что большинство из них может функционировать в других типах регуляторных путей. На этом этапе исследователи начали использовать термин «микроРНК» для обозначения этого класса малых регуляторных РНК.
Первым заболеванием человека, связанным с нарушением регуляции miRNA, был хронический лимфолейкоз . При этом заболевании miRNA играют двойную роль, работая как супрессоры опухолей, так и онкогены.
Номенклатура
В соответствии со стандартной системой номенклатуры имена присваиваются экспериментально подтвержденным miRNA перед публикацией. За префиксом miR следует тире и число, последнее часто указывает порядок именования. Например, miR-124 был назван и, вероятно, обнаружен до miR-456. «MiR-» с заглавной буквы относится к зрелой форме miRNA, а «mir-» без заглавной буквы относится к pre-miRNA и pri-miRNA. Гены, кодирующие миРНК, также называются с использованием того же трехбуквенного префикса в соответствии с соглашениями номенклатуры генов организма. Например, официальные названия генов miRNA у некоторых организмов: « mir-1 у C. elegans и Drosophila, Mir-1 у Rattus norvegicus и MIR-25 у человека».
miRNA с почти идентичными последовательностями, за исключением одного или двух нуклеотидов, помечаются дополнительной строчной буквой. Например, miR-124a тесно связана с miR-124b. Например:
-
hsa-miR-181a :aacauucaACgcugucggugAgu
- hsa-miR-181b :aacauucaUUgcugucggugGgu
Пре-миРНК, при-миРНК и гены, которые приводят к 100% идентичным зрелым миРНК, но которые расположены в разных местах генома, обозначаются дополнительным суффиксом в виде тире. Например, пре-миРНК hsa-mir-194-1 и hsa-mir-194-2 приводят к идентичной зрелой миРНК (hsa-miR-194), но происходят из генов, расположенных в разных областях генома.
Виды происхождения обозначены трехбуквенным префиксом, например, hsa-miR-124 — это миРНК человека ( Homo sapiens ), а oar-miR-124 — миРНК овцы ( Ovis aries ). Другие распространенные префиксы включают «v» для вируса (miRNA, кодируемая вирусным геномом) и «d» для miRNA дрозофилы (плодовая муха, обычно изучаемая в генетических исследованиях).
Когда две зрелые микроРНК происходят из противоположных ветвей одной и той же пре-миРНК и обнаруживаются в примерно одинаковых количествах, они обозначаются суффиксом -3p или -5p. (В прошлом это различие также проводилось с помощью «s» ( смысл ) и «as» (антисмысловой)). Однако зрелых микроРНК, обнаруженных в одном плече шпильки, обычно гораздо больше, чем в другом плече, и в этом случае звездочка после названия указывает на зрелые виды, обнаруженные на низких уровнях в противоположном плече шпильки. Например, miR-124 и miR-124 * имеют общую шпильку пре-miRNA, но в клетке обнаруживается гораздо больше miR-124.
Цели
MiRNA растений обычно имеют почти идеальное спаривание со своими мишенями мРНК, что вызывает репрессию генов через расщепление транскриптов-мишеней. Напротив, miRNA животных способны распознавать свои мРНК-мишени, используя всего 6-8 нуклеотидов (область затравки) на 5′-конце miRNA, что недостаточно для спаривания, чтобы вызвать расщепление мРНК-мишени. Комбинаторная регуляция — это особенность регуляции miRNA у животных. Данная миРНК может иметь сотни различных мишеней мРНК, и данная мишень может регулироваться множеством миРНК.
Оценки среднего числа уникальных матричных РНК, которые являются мишенями для репрессии с помощью типичной miRNA, варьируются в зависимости от метода оценки, но несколько подходов показывают, что miRNA млекопитающих могут иметь множество уникальных мишеней. Напр., Анализ miRNAs, высококонсервативных у позвоночных, показывает, что каждая имеет в среднем примерно 400 консервативных мишеней. Точно так же эксперименты показывают, что один вид miRNA может снижать стабильность сотен уникальных информационных РНК. Другие эксперименты показывают, что один вид miRNA может подавлять продукцию сотен белков, но эта репрессия часто бывает относительно мягкой (гораздо меньше, чем в 2 раза). Первым заболеванием человека, связанным с нарушением регуляции miRNA, был хронический лимфолейкоз . Последовали и другие В-клеточные злокачественные новообразования.
Биогенез
До 40% генов miRNA могут находиться в интронах или даже экзонах других генов. Они обычно, хотя и не исключительно, находятся в определенной смысловой ориентации и, таким образом, обычно регулируются вместе с их генами-хозяевами.
ДНК-матрица — не последнее слово в вопросе продукции зрелой miRNA: 6% человеческих miRNA демонстрируют редактирование РНК ( IsomiRs ), сайт-специфическую модификацию последовательностей РНК для получения продуктов, отличных от тех, которые кодируются их ДНК. Это увеличивает разнообразие и объем действия miRNA, выходящий за рамки того, что связано только с геномом.
Транскрипция
Гены miRNA обычно транскрибируются РНК-полимеразой II (Pol II). Полимераза часто связывается с промотором, обнаруженным рядом с последовательностью ДНК, кодируя то, что станет петлей шпильки пре-миРНК. Полученный транскрипт кэпируется специально модифицированным нуклеотидом на 5′-конце, полиаденилируется множеством аденозинов (поли (A) хвост) и сплайсируется . МикроРНК животных первоначально транскрибируется как часть одного плеча стволовой петли РНК из 80 нуклеотидов, которая, в свою очередь, образует часть предшественника миРНК длиной в несколько сотен нуклеотидов, называемого при-миРНК. Когда предшественник «стебель-петля» обнаруживается в 3′-UTR, транскрипт может служить в качестве при-миРНК и мРНК. РНК-полимераза III (Pol III) транскрибирует некоторые miRNA, особенно с последовательностями Alu выше , транспортными РНК (тРНК) и промоторными единицами с широким вкраплением повторов (MWIR) млекопитающих.
Ядерная обработка
Одна при-миРНК может содержать от одного до шести предшественников миРНК. Эти структуры петли шпильки состоят примерно из 70 нуклеотидов каждая. Каждая шпилька окружена последовательностями, необходимыми для эффективной обработки.
Структура двухцепочечной РНК (дцРНК) шпилек в при-миРНК распознается ядерным белком, известным как критическая область 8 синдрома ДиДжорджи (DGCR8 или «Паша» у беспозвоночных), названная в честь его ассоциации с синдромом ДиДжорджи . DGCR8 связывается с ферментом Drosha , белком, который разрезает РНК, с образованием микропроцессорного комплекса . В этом комплексе DGCR8 ориентирует каталитический домен РНКазы III Drosha для освобождения шпилек от pri-miRNAs путем расщепления РНК примерно на одиннадцать нуклеотидов от основания шпильки (одна спиральная дцРНК превращается в стебель). Полученный продукт имеет двухнуклеотидный выступ на его 3′-конце; он имеет 3 ‘гидроксильные и 5’ фосфатные группы. Ее часто называют пре-миРНК (миРНК-предшественница). Были идентифицированы мотивы последовательностей ниже пре-miRNA, которые важны для эффективного процессинга.
Пре-миРНК, которые сплайсируются непосредственно из интронов, минуя микропроцессорный комплекс, известны как « миртроны ». Первоначально считалось , что миртроны существуют только у Drosophila и C. elegans , теперь же миртроны обнаружены у млекопитающих.
До 16% пре-miRNAs могут быть изменены посредством редактирования ядерной РНК . Чаще всего ферменты, известные как аденозиндезаминазы, действующие на РНК (ADAR), катализируют переходы аденозина в инозин (от A к I). Редактирование РНК может останавливать ядерный процессинг (например, pri-miR-142, что приводит к деградации рибонуклеазой Tudor-SN) и изменять последующие процессы, включая процессинг цитоплазматической miRNA и специфичность мишени (например, путем изменения затравочной области miR-376 в центральной нервной системе).
Ядерный экспорт
Человек Exportin-5 белки (красная) в комплексе с Ran-ГТФ (желтым) и предварительно микроРНКом (зеленый), показывая двух- нуклеотидного элемент распознавания свеса (оранжевый). Из PDB : 3A6P .
Шпильки пре-миРНК экспортируются из ядра в процессе с участием ядерно — цитоплазматического челночного транспортера Exportin-5 . Этот белок, член семейства кариоферинов , распознает двухнуклеотидный выступ, оставленный ферментом РНКазы III Drosha на 3′-конце шпильки пре-миРНК. Опосредованный экспортином-5 транспорт в цитоплазму зависит от энергии с использованием гуанозинтрифосфата (GTP), связанного с белком Ran .
Цитоплазматическая обработка
В цитоплазме шпилька пре-миРНК расщепляется ферментом РНКазы III Dicer . Эта эндорибонуклеаза взаимодействует с 5 ‘и 3’ концами шпильки и отрезает петлю, соединяющую 3 ‘и 5’ ветви, давая несовершенный дуплекс miRNA: miRNA * длиной около 22 нуклеотидов. Общая длина шпильки и размер петли влияют на эффективность обработки дайсером. Несовершенная природа спаривания miRNA: miRNA * также влияет на расщепление. Некоторые из G-богатых пре-miRNAs потенциально могут принимать структуру G-квадруплекса в качестве альтернативы канонической структуре стержень-петля. Например, человеческая пре-миРНК 92b принимает структуру G-квадруплекса, которая устойчива к опосредованному Дайсером расщеплению в цитоплазме . Хотя любая цепь дуплекса потенциально может действовать как функциональная miRNA, только одна цепь обычно включается в РНК-индуцированный комплекс сайленсинга (RISC), где miRNA и ее мишень мРНК взаимодействуют.
Хотя большинство miRNA расположены внутри клетки, некоторые miRNA, обычно известные как циркулирующие miRNA или внеклеточные miRNA, также были обнаружены во внеклеточной среде, включая различные биологические жидкости и среды для культивирования клеток.
Биогенез в растениях
Биогенез miRNA в растениях отличается от биогенеза животных, главным образом, стадиями ядерной обработки и экспорта. Вместо того, чтобы расщепляться двумя разными ферментами, один раз внутри и один раз за пределами ядра, оба расщепления миРНК растения выполняются гомологом Dicer, называемым Dicer-like1 (DL1). DL1 экспрессируется только в ядре растительных клеток, что указывает на то, что обе реакции происходят внутри ядра. До того, как дуплексы miRNA: miRNA * переносятся из ядра, его 3′-выступы метилируются с помощью РНК-метилтрансферазного белка, называемого Hua-Enhancer1 (HEN1). Затем дуплекс транспортируется из ядра в цитоплазму с помощью белка, называемого Hasty (HST), гомолога Exportin 5, где они разбираются, и зрелая миРНК включается в RISC.
РНК-индуцированный комплекс сайленсинга
Зрелая miRNA является частью активного комплекса RNA-индуцированного сайленсинга (RISC), содержащего Dicer и многие связанные с ним белки. RISC также известен как комплекс рибонуклеопротеидов микроРНК (miRNP); RISC со встроенной miRNA иногда называют miRISC.
Считается, что процессинг пре-миРНК дайсером связан с раскручиванием дуплекса. Обычно в miRISC включается только одна цепь, выбранная на основе ее термодинамической нестабильности и более слабого спаривания оснований на 5′-конце по сравнению с другой цепью. Положение стержня-петли также может влиять на выбор пряди. Другая ветвь, называемая пассажирской цепью из-за ее более низких уровней в установившемся состоянии, обозначена звездочкой (*) и обычно деградирует. В некоторых случаях обе цепи дуплекса жизнеспособны и становятся функциональной miRNA, нацеленной на разные популяции мРНК.
Члены семейства белков Argonaute (Ago) играют центральную роль в функции RISC. Аргонавты необходимы для индуцированного miRNA сайленсинга и содержат два консервативных домена связывания РНК: домен PAZ, который может связывать одноцепочечный 3′-конец зрелой miRNA, и домен PIWI, который структурно напоминает рибонуклеазу-H и функционирует для взаимодействия с 5′- концом. конец направляющей пряди. Они связывают зрелую миРНК и ориентируют ее для взаимодействия с целевой мРНК. Некоторые аргонавты, например человеческий Ago2, непосредственно расщепляют транскрипты-мишени; аргонавты могут также привлекать дополнительные белки для достижения репрессии трансляции. Геном человека кодирует восемь белков аргонаута, разделенных по сходству последовательностей на два семейства: AGO (четыре члена присутствуют во всех клетках млекопитающих и называются E1F2C / hAgo у людей) и PIWI (обнаруживаются в зародышевой линии и гемопоэтических стволовых клетках).
Дополнительные компоненты RISC включают TRBP [белок, связывающий РНК (TAR), трансактивирующую ответную РНК вируса иммунодефицита человека (ВИЧ)], PACT (белковый активатор интерферон- индуцированной протеинкиназы ), комплекс SMN, ломкий белок X-умственной отсталости (FMRP), стафилококковый тудор. белок, содержащий домен нуклеазы (Tudor-SN), предполагаемая ДНК- геликаза MOV10 и мотив узнавания РНК, содержащий белок TNRC6B .
Режим молчания и регуляторные петли
Молчание генов может происходить либо за счет деградации мРНК, либо за счет предотвращения трансляции мРНК. Например, miR16 содержит последовательность, комплементарную AU-богатому элементу, обнаруженному в 3’UTR многих нестабильных мРНК, таких как TNF-альфа или GM-CSF . Было продемонстрировано, что при полной комплементарности между miRNA и последовательностью мРНК-мишени Ago2 может расщеплять мРНК и приводить к прямой деградации мРНК. В отсутствие комплементарности молчание достигается за счет предотвращения трансляции. Взаимосвязь miRNA и ее целевой мРНК может быть основана на простой отрицательной регуляции целевой мРНК, но кажется, что распространенным сценарием является использование «когерентной петли прямой связи », «петли взаимной отрицательной обратной связи» (также называемой двойной отрицательный контур) и «контур положительной обратной связи / прямой связи». Некоторые miRNA работают как буферы случайных изменений экспрессии генов, возникающих из-за случайных событий транскрипции, трансляции и стабильности белка. Такая регуляция обычно достигается за счет петель отрицательной обратной связи или некогерентной петли прямой связи, разобщающей выход белка с транскрипцией мРНК.
Оборот
Оборот зрелой miRNA необходим для быстрых изменений в профилях экспрессии miRNA. Считается, что во время созревания miRNA в цитоплазме поглощение белком Argonaute стабилизирует направляющую цепь, в то время как противоположная (* или «пассажирская») цепь предпочтительно разрушается. В том, что было названо стратегией «Используй или потеряй», Argonaute может предпочтительно сохранять miRNAs со многими мишенями, чем miRNAs с небольшим количеством мишеней или без них, что приводит к деградации нецелевых молекул.
Распад зрелых миРНК у Caenorhabditis elegans опосредуется экзорибонуклеазой XRN2 с 5′-к- 3’ом , также известной как Rat1p. У растений члены семейства SDN (малые РНК-деградирующие нуклеазы) разрушают miRNA в противоположном (3′-к-5 ‘) направлении. Подобные ферменты кодируются в геномах животных, но их роль не описана.
Некоторые модификации miRNA влияют на стабильность miRNA. Как показала работа с модельным организмом Arabidopsis thaliana (кресс-салат), миРНК зрелых растений, по-видимому, стабилизируются добавлением метильных групп на 3′-конце. 2′-O-конъюгированные метильные группы блокируют добавление остатков урацила (U) ферментами уридилтрансферазы , модификация, которая может быть связана с деградацией miRNA. Однако уридилирование может также защищать некоторые miRNA; последствия этой модификации до конца не изучены. Сообщалось об уридилировании некоторых микроРНК животных. Как растения, так и животные miRNA могут быть изменены путем добавления остатков аденина (A) к 3′-концу miRNA. Дополнительный A, добавленный к концу miR-122 млекопитающих , обогащенной печенью miRNA, важной при гепатите C , стабилизирует молекулу, а миРНК растений, заканчивающиеся остатком аденина, имеют более медленную скорость распада.
Клеточные функции
Взаимодействие микроРНК с процессом трансляции белков. Показаны несколько механизмов репрессии трансляции: M1) в процессе инициации, предотвращая сборку инициирующего комплекса или рекрутируя 40S рибосомную субъединицу; M2) на сборке рибосом; M3) о процессе перевода; M7, M8) на деградацию мРНК. 40S и 60S — это легкий и тяжелый компоненты рибосомы, 80S — это собранная рибосома, связанная с мРНК, eIF4F — фактор инициации трансляции, PABC1 — связывающий белок Poly-A, а «cap» — структура кэпа мРНК, необходимая для циркуляризации мРНК. (который может быть обычным m7G-cap или модифицированным A-cap). Инициирование мРНК может происходить независимым от кэпа образом посредством рекрутирования 40S в IRES (сайт внутреннего входа рибосомы ), расположенный в области 5’UTR. Фактическая работа по подавлению молчания РНК выполняется RISC, в котором основной каталитической субъединицей является один из белков Argonaute (AGO), а miRNA служит матрицей для распознавания специфических последовательностей мРНК.
Функция miRNA, по-видимому, заключается в регуляции генов. Для этой цели miRNA является комплементарной частью одной или нескольких информационных РНК (мРНК). MiRNA животных обычно комплементарны сайту в 3 ‘UTR, тогда как miRNA растений обычно комплементарны кодирующим областям мРНК. Идеальное или почти идеальное спаривание оснований с целевой РНК способствует расщеплению РНК. Это основной вид миРНК растений. У животных совпадения несовершенные.
Для частично комплементарных микроРНК, чтобы распознавать свои мишени, нуклеотиды 2-7 miRNA (ее «затравочная область») должны быть совершенно комплементарными. MiRNA животных ингибируют трансляцию белка целевой мРНК (это присутствует, но реже у растений). Частично комплементарные микроРНК также могут ускорять деаденилирование , вызывая более раннюю деградацию мРНК. В то время как деградация мРНК, нацеленная на miRNA, хорошо задокументирована, вопрос о том, достигается ли репрессия трансляции посредством деградации мРНК, ингибирования трансляции или их комбинации, горячо обсуждается. Недавняя работа с miR-430 у рыбок данио, а также с bantam-miRNA и miR-9 в культивируемых клетках дрозофилы , показывает, что репрессия трансляции вызывается нарушением инициации трансляции , независимо от деаденилирования мРНК.
микроРНК иногда также причина модификации гистонов и метилирование ДНК из промотора сайтов, что влияет на экспрессию генов — мишеней.
Девять механизмов действия miRNA описаны и собраны в единую математическую модель:
- Ингибирование инициации Cap-40S;
- 60S ингибирование присоединения рибосомных единиц;
- Замедление удлинения;
- Выпадение рибосомы (преждевременное прекращение);
- Ко-трансляционная деградация растущего белка;
- Секвестр в П-телах;
- распад мРНК (дестабилизация);
- расщепление мРНК;
- Ингибирование транскрипции посредством реорганизации хроматина, опосредованной микроРНК, с последующим подавлением генов.
Эти механизмы часто невозможно распознать, используя экспериментальные данные о стационарных скоростях реакций. Тем не менее они различаются по динамике и имеют разные кинетические сигнатуры .
В отличие от растительных микроРНК, животные микроРНК нацелены на различные гены. Однако гены, участвующие в функциях, общих для всех клеток, таких как экспрессия генов, имеют относительно меньше участков-мишеней для микроРНК и, по-видимому, подвергаются отбору, чтобы избежать нацеливания микроРНК.
dsRNA может также активировать экспрессию гена , механизм, который был назван «активацией гена, индуцированной малой РНК» или RNAa . дцРНК, нацеленные на промоторы генов, могут вызывать сильную активацию транскрипции ассоциированных генов. Это было продемонстрировано на клетках человека с использованием синтетических дцРНК, называемых малыми активирующими РНК ( саРНК ), но также было продемонстрировано для эндогенных микроРНК.
Считается, что взаимодействия между микроРНК и комплементарными последовательностями генов и даже псевдогенами, которые имеют общую гомологию последовательностей, являются обратным каналом связи, регулирующим уровни экспрессии между паралогичными генами (генами, имеющими аналогичную структуру, указывающую на дивергенцию от общего предкового гена). Получив название «конкурирующие эндогенные РНК» ( цРНК ), эти микроРНК связываются с «элементами ответа микроРНК» на генах и псевдогенах и могут служить другим объяснением устойчивости некодирующей ДНК.
Некоторые исследования показывают, что груз мРНК экзосом может играть роль в имплантации, они могут нарушать адгезию между трофобластом и эндометрием или поддерживать адгезию путем понижающей или повышающей регуляции экспрессии генов, участвующих в адгезии / инвазии.
Более того, miRNA как miR-183/96/182, по- видимому, играет ключевую роль в циркадном ритме .
Эволюция
miRNAs хорошо сохраняются как у растений, так и у животных и считаются жизненно важным и эволюционно древним компонентом регуляции генов. В то время как основные компоненты пути микроРНК сохраняются у растений и животных , репертуары миРНК в двух царствах, по-видимому, возникли независимо с разными первичными способами действия.
микроРНК являются полезными филогенетическими маркерами из-за их явно низкой скорости эволюции. Происхождение микроРНК как регуляторный механизм развился из предыдущего механизма РНКи, который первоначально использовался в качестве защиты от экзогенного генетического материала, такого как вирусы. Их происхождение, возможно, способствовало развитию морфологических инноваций и, сделав экспрессию генов более специфичной и «тонко настраиваемой», позволило генезису сложных органов и, возможно, в конечном итоге, сложной жизни. Быстрые всплески морфологических инноваций обычно связаны с высокой скоростью накопления микроРНК.
Новые микроРНК создаются несколькими способами. Новые микроРНК могут возникать в результате случайного образования шпилек в «некодирующих» участках ДНК (то есть в интронах или межгенных областях), но также в результате дупликации и модификации существующих микроРНК. микроРНК могут также образовываться из перевернутых дубликатов последовательностей, кодирующих белок, что позволяет создавать складную структуру шпильки. Скорость эволюции (т. Е. Нуклеотидного замещения) в недавно возникших микроРНК сравнима с таковой в других частях некодирующей ДНК, что подразумевает эволюцию за счет нейтрального дрейфа; однако более старые микроРНК имеют гораздо более низкую скорость изменения (часто менее одной замены за сто миллионов лет), предполагая, что, как только микроРНК приобретает функцию, она подвергается очищающей селекции. Отдельные области в гене miRNA сталкиваются с разными эволюционными давлениями, где области, которые жизненно важны для процессинга и функционирования, имеют более высокие уровни консервации. На этом этапе микроРНК редко теряется из генома животного, хотя более новые микроРНК (таким образом, предположительно нефункциональные) часто теряются. У Arabidopsis thaliana чистый поток генов miRNA, по прогнозам, составляет от 1,2 до 3,3 генов на миллион лет. Это делает их ценным филогенетическим маркером, и они рассматриваются как возможное решение выдающихся филогенетических проблем, таких как взаимоотношения членистоногих . С другой стороны, во многих случаях микроРНК плохо коррелируют с филогенезом, и возможно, что их филогенетическое соответствие в значительной степени отражает ограниченный выбор микроРНК.
МикроРНК присутствуют в геномах большинства эукариотических организмов, от бурых водорослей до животных. Однако разница в том, как эти микроРНК функционируют и как они обрабатываются, предполагает, что микроРНК возникли независимо у растений и животных.
Сосредоточившись на животных, в геноме Mnemiopsis leidyi, по- видимому, отсутствуют узнаваемые микроРНК, а также ядерные белки Drosha и Pasha , которые имеют решающее значение для биогенеза канонических микроРНК. На данный момент это единственное животное, пропавшее без вести Дроша. МикроРНК играют жизненно важную роль в регуляции экспрессии генов у всех исследованных животных без гребневиков, за исключением Trichoplax adhaerens , единственного известного представителя типа Placozoa .
У всех видов к марту 2010 года было идентифицировано более 5000 различных miRNA. В то время как короткие последовательности РНК (50 — сотни пар оснований) с широко сопоставимой функцией встречаются у бактерий, у бактерий отсутствуют настоящие микроРНК.
Экспериментальное обнаружение и манипуляции
В то время как исследователи сосредоточились на экспрессии miRNA в физиологических и патологических процессах, возникли различные технические переменные, связанные с выделением микроРНК. Стабильность хранимых образцов miRNA была поставлена под сомнение. микроРНК разлагаются намного легче, чем мРНК, отчасти из-за их длины, но также из-за повсеместного присутствия РНКаз . Это требует охлаждения образцов на льду и использования оборудования, не содержащего РНКазы .
Экспрессия микроРНК может быть определена количественно в процессе двухэтапной полимеразной цепной реакции модифицированной ОТ-ПЦР с последующей количественной ПЦР . Варианты этого метода позволяют получить абсолютную или относительную количественную оценку. miRNA также можно гибридизировать с микрочипами , слайдами или чипами с зондами для сотен или тысяч мишеней miRNA, так что относительные уровни miRNA могут быть определены в различных образцах. микроРНК могут быть обнаружены и профилированы методами высокопроизводительного секвенирования ( секвенирование микроРНК ). Активность в микроРНК может быть экспериментально ингибируется с использованием заперта нуклеиновой кислоты (LNA) олиго , а морфолино олиго или РНК олиго 2′-О-метил. Конкретная miRNA может подавляться комплементарным антагомиром . Созревание микроРНК может ингибироваться в нескольких точках стерически-блокирующими олигонуклеотидами. Сайт-мишень miRNA транскрипта мРНК также может быть заблокирован стерически-блокирующим олиго. Для обнаружения «in situ» miRNA могут использоваться зонды LNA или Morpholino. Закрытая конформация LNA приводит к улучшенным свойствам гибридизации и увеличивает чувствительность и селективность, что делает ее идеальной для обнаружения коротких miRNA.
Высокопроизводительная количественная оценка miRNA подвержена ошибкам из-за большей дисперсии (по сравнению с мРНК ), которая сопряжена с методологическими проблемами. Поэтому экспрессию мРНК часто анализируют, чтобы проверить эффекты miRNA на их уровнях (например, in). Базы данных могут использоваться для объединения данных мРНК и миРНК, которые предсказывают цели миРНК на основе их базовой последовательности. Хотя это обычно делается после обнаружения представляющих интерес miRNA (например, из-за высоких уровней экспрессии), были предложены идеи инструментов анализа, которые интегрируют информацию об экспрессии мРНК и miRNA.
Болезнь
Подобно тому, как miRNA участвует в нормальном функционировании эукариотических клеток, нарушение регуляции miRNA связано с заболеванием. Созданная вручную общедоступная база данных miR2Disease документирует известную взаимосвязь между нарушением регуляции miRNA и заболеванием человека.
Наследственные болезни
Мутация в семенной области miR-96 вызывает наследственную прогрессирующую потерю слуха.
Мутация в семенной области miR-184 вызывает наследственный кератоконус с передней полярной катарактой.
Делеция кластера miR-17 ~ 92 вызывает дефекты скелета и роста.
Рак
Роль miRNA в раковой клетке
Первым заболеванием человека, связанным с нарушением регуляции miRNA, был хронический лимфолейкоз. Многие другие miRNA также связаны с раком и, соответственно, иногда называются « онкомирами ». В злокачественных В-клетках миРНК участвуют в путях, фундаментальных для развития В-клеток, таких как передача сигналов рецептора В-клеток (BCR), миграция / адгезия В-клеток, межклеточные взаимодействия в иммунных нишах и производство и переключение классов иммуноглобулинов. MiRNA влияют на созревание В-клеток, образование пре-, маргинальной зоны, фолликулярных, В1, плазменных В-клеток и В-клеток памяти.
Другая роль miRNA при раке заключается в использовании уровня их экспрессии для прогноза. В образцах NSCLC низкие уровни miR-324a могут служить индикатором плохой выживаемости. Высокие уровни miR-185 или низкие уровни miR-133b могут коррелировать с метастазированием и плохой выживаемостью при колоректальном раке .
Кроме того, определенные miRNA могут быть связаны с определенными гистологическими подтипами колоректального рака. Например, было показано, что уровни экспрессии miR-205 и miR-373 увеличиваются при муцинозном колоректальном раке и связанном с муцином язвенном колите раке толстой кишки, но не при спорадической аденокарциноме толстой кишки, в которой отсутствуют муцинозные компоненты. Исследования in vitro показали, что miR-205 и miR-373 могут функционально индуцировать различные признаки опухолевого прогрессирования, связанного с муцином, в эпителиальных клетках кишечника.
Пролиферация клеток гепатоцеллюлярной карциномы может возникать в результате взаимодействия miR-21 с MAP2K3, геном-репрессором опухоли. Оптимальное лечение рака включает точное определение пациентов для терапии со стратификацией риска. Те, у кого есть быстрый ответ на начальное лечение, могут получить пользу от сокращенных схем лечения, что свидетельствует о ценности точных показателей реакции на заболевание. Внеклеточные циркулирующие миРНК (кимиРНК) очень стабильны в крови, сверхэкспрессируются при раке и поддаются количественной оценке в диагностической лаборатории. При классической лимфоме Ходжкина плазменные miR-21, miR-494 и miR-1973 являются многообещающими биомаркерами ответа на заболевание. Циркулирующие миРНК могут помочь в принятии клинических решений и в интерпретации позитронно-эмиссионной томографии в сочетании с компьютерной томографией . Их можно проводить на каждой консультации для оценки реакции на заболевание и выявления рецидива.
МикроРНК могут быть использованы в качестве инструментов или мишеней для лечения различных видов рака. В нескольких исследованиях было обнаружено, что специфическая микроРНК, miR-506, действует как антагонист опухоли. Было обнаружено, что в значительном количестве образцов рака шейки матки экспрессия miR-506 подавлена. Кроме того, miR-506 способствует апоптозу клеток рака шейки матки посредством своего прямого транскрипционного фактора пути хэджхог, Gli3.
Ремонт ДНК и рак
Рак вызывается накоплением мутаций либо из-за повреждения ДНК, либо из-за неисправленных ошибок репликации ДНК . Дефекты репарации ДНК вызывают накопление мутаций, которые могут привести к раку. Некоторые гены, участвующие в репарации ДНК, регулируются микроРНК.
Мутации зародышевой линии в генах репарации ДНК вызывают только 2–5% случаев рака толстой кишки . Однако измененная экспрессия микроРНК, вызывающая дефицит репарации ДНК, часто связана с раком и может быть важным причинным фактором. Среди 68 спорадического рака толстой кишки с пониженной экспрессией ДНК — репарация ошибочно спаренной белок MLH1 , большинство из них были признаны недостаточными из — за эпигенетический метилирования на CpG острове MLH1 гена. Однако до 15% дефицита MLH1 при спорадическом раке толстой кишки, по-видимому, происходит из-за сверхэкспрессии микроРНК miR-155, которая подавляет экспрессию MLH1.
В 29–66% глиобластом репарация ДНК недостаточна из-за эпигенетического метилирования гена MGMT , которое снижает экспрессию белка MGMT. Однако для 28% глиобластом белок MGMT недостаточен, но промотор MGMT не метилирован. В глиобластомах без метилированных промоторов MGMT уровень микроРНК miR-181d обратно коррелирует с экспрессией белка MGMT, а прямой мишенью miR-181d является мРНК 3’UTR MGMT ( три основных нетранслируемых участка мРНК MGMT). Таким образом, в 28% глиобластом повышенная экспрессия miR-181d и пониженная экспрессия фермента репарации ДНК MGMT могут быть причинным фактором.
Белки HMGA (HMGA1a, HMGA1b и HMGA2) участвуют в развитии рака, и экспрессия этих белков регулируется микроРНК. Экспрессия HMGA почти не обнаруживается в дифференцированных тканях взрослого человека, но повышена во многих случаях рака. Белки HMGA представляют собой полипептиды из ~ 100 аминокислотных остатков, характеризующиеся модульной организацией последовательностей. Эти белки имеют три высоко положительно заряженных области, называемых крючками АТ , которые связывают малую бороздку богатых АТ участков ДНК в определенных областях ДНК. Новообразования человека, в том числе карциномы щитовидной железы, предстательной железы, шейки матки, колоректального рака, поджелудочной железы и яичников, демонстрируют сильное увеличение белков HMGA1a и HMGA1b. У трансгенных мышей с HMGA1, нацеленным на лимфоидные клетки, развивается агрессивная лимфома, что показывает, что высокая экспрессия HMGA1 связана с раком и что HMGA1 может действовать как онкоген. Белок HMGA2 специфически нацелен на промотор ERCC1 , тем самым снижая экспрессию этого гена репарации ДНК. Экспрессия белка ERCC1 была недостаточной в 100% из 47 оцененных случаев рака толстой кишки (хотя степень участия HGMA2 неизвестна).
Однонуклеотидные полиморфизмы (SNP) могут изменять связывание miRNA с 3’UTR, например, в случае hsa-mir181a и hsa-mir181b с геном-супрессором опухоли CDON.
Сердечное заболевание
Глобальная роль функции miRNA в сердце решается путем условного ингибирования созревания miRNA в сердце мыши . Это показало, что miRNAs играют важную роль во время его развития. Исследования профилей экспрессии miRNA демонстрируют, что уровни экспрессии определенных miRNA изменяются в пораженном сердце человека, указывая на их участие в кардиомиопатиях . Более того, исследования специфических miRNA на животных выявили различные роли miRNA как во время развития сердца, так и при патологических состояниях, включая регуляцию ключевых факторов, важных для кардиогенеза, реакции гипертрофического роста и сердечной проводимости. Другая роль miRNA при сердечно-сосудистых заболеваниях — использование уровней их экспрессии для диагностики, прогноза или стратификации риска. miRNA в моделях на животных также были связаны с метаболизмом и регуляцией холестерина.
miRNA-712
МикроРНК-712 мыши является потенциальным биомаркером (то есть предиктором) атеросклероза , сердечно-сосудистого заболевания артериальной стенки, связанного с задержкой липидов и воспалением. Неламинарный кровоток также коррелирует с развитием атеросклероза, поскольку механосеноры эндотелиальных клеток реагируют на сдвигающую силу нарушенного кровотока (d-поток). Ряд проатерогенных генов, включая матриксные металлопротеиназы (ММР), активируются с помощью d-потока, опосредуя провоспалительные и проангиогенные сигналы. Эти результаты наблюдались в перевязанных сонных артериях мышей, чтобы имитировать эффекты d-потока. В течение 24 часов ранее существовавшая незрелая miR-712 образовывала зрелую miR-712, что позволяет предположить, что miR-712 является чувствительной к потоку. Соответствуя этим результатам, miR-712 также активируется в эндотелиальных клетках, подверженных естественному d-потоку в большей кривизне дуги аорты.
Источник
Последовательность пре-мРНК miR-712 генерируется из мышиного гена рибосомных RN45s во внутренней транскрибируемой спейсерной области 2 (ITS2). XRN1 представляет собой экзонуклеазу, которая разрушает область ITS2 во время процессинга RN45. Таким образом, уменьшение XRN1 в условиях d-потока ведет к накоплению miR-712.
Механизм
MiR-712 нацелен на тканевой ингибитор металлопротеиназы 3 (TIMP3). ТИМП обычно регулируют активность матриксных металлопротеиназ (ММП), которые разрушают внеклеточный матрикс (ЕСМ). Артериальный ECM в основном состоит из волокон коллагена и эластина , обеспечивающих структурную поддержку и свойства отдачи артерий. Эти волокна играют решающую роль в регуляции воспаления и проницаемости сосудов, которые важны в развитии атеросклероза. Экспрессируемый эндотелиальными клетками, TIMP3 является единственным TIMP, связанным с ECM. Снижение экспрессии TIMP3 приводит к увеличению деградации ECM в присутствии d-потока. В соответствии с этими находками, ингибирование pre-miR712 увеличивает экспрессию TIMP3 в клетках даже при воздействии турбулентного потока.
TIMP3 также снижает экспрессию TNFα (провоспалительного регулятора) во время турбулентного потока. Активность TNFα в турбулентном потоке измеряли по экспрессии TNFα-конвертирующего фермента (TACE) в крови. TNFα снижается, если miR-712 ингибируется или TIMP3 сверхэкспрессируется, что позволяет предположить, что miR-712 и TIMP3 регулируют активность TACE в условиях турбулентного потока.
Анти-miR-712 эффективно подавляет экспрессию miR-712, индуцированную d-потоком, и увеличивает экспрессию TIMP3. Анти-miR-712 также ингибирует повышенную проницаемость сосудов, тем самым значительно снижая развитие атеросклероза и инфильтрацию иммунных клеток.
Человеческий гомолог микроРНК-205
Человеческий гомолог miR-712 был обнаружен в гене гомолога RN45s, который поддерживает miRNA, сходные с мышами. MiR-205 человека имеет сходные последовательности с miR-712 мышей и сохраняется у большинства позвоночных. MiR-205 и miR-712 также разделяют более 50% мишеней передачи сигналов клеток, включая TIMP3.
При тестировании d-поток снижал экспрессию XRN1 у людей, как и в эндотелиальных клетках мышей, что указывает на потенциально общую роль XRN1 у людей.
Болезнь почек
Нацеленная делеция Dicer в производных от FoxD1 почечных клетках-предшественниках на мышиной модели привела к сложному почечному фенотипу, включающему распространение предшественников нефронов , меньшее количество рениновых клеток, артериол гладких мышц , прогрессирующую мезангиальную потерю и клубочковые аневризмы. Высокопроизводительное профилирование полного транскриптома модели мышей с нокаутом FoxD1-Dicer выявило эктопическую активацию проапоптотического гена, Bcl2L11 (Bim) и нарушение регуляции пути p53 с увеличением эффекторных генов p53, включая Bax , Trp53inp1 , Jun, Cdkn1a , Mmp2 и Arid3a . Уровни белка p53 остались неизменными, что позволяет предположить, что стромальные miRNA FoxD1 непосредственно репрессируют гены-эффекторы p53. Используя метод отслеживания клонов с последующей сортировкой флуоресцентно-активируемых клеток , профилирование миРНК клеток, полученных из FoxD1, не только всесторонне определили транскрипционный ландшафт miРНК, которые имеют решающее значение для развития сосудов, но также идентифицировали ключевые миРНК, которые могут модулировать почечный фенотип. в его отсутствие. Эти miRNA включают miRs-10a, 18a, 19b, 24, 30c, 92a, 106a, 130a, 152, 181a, 214, 222, 302a, 370 и 381, которые регулируют Bcl2L11 (Bim) и miRs-15b, 18a, 21, 30c, 92a, 106a, 125b-5p, 145, 214, 222, 296-5p и 302a, которые регулируют гены эффекторов р53. В соответствии с результатами профилирования, эктопический апоптоз наблюдался в клеточных производных линии предков, происходящих от FoxD1, и подтверждает важность почечных стромальных miRNAs в клеточном гомеостазе.
Нервная система
miRNAs, по-видимому, регулируют развитие и функцию нервной системы . Нейронные микроРНК вовлечены на различных стадиях развития синаптической, в том числе dendritogenesis ( с участием микроРНК-132 , MIR-134 и MIR-124 ), синапсов формирования и созревания синапсов (где микроРНК-134 и микроРНК-138 , как полагают, участвует). Устранение образования miRNA у мышей путем экспериментального подавления Dicer привело к патологическим результатам, таким как уменьшение размера нейронов и двигательные аномалии при подавлении молчания в нейронах полосатого тела и нейродегенерация при подавлении молчания в нейронах переднего мозга . Некоторые исследования обнаруживают измененную экспрессию miRNA при болезни Альцгеймера , а также при шизофрении , биполярном расстройстве , большой депрессии и тревожных расстройствах .
Инсульт
По данным Центра по контролю и профилактике заболеваний, инсульт является одной из основных причин смерти и длительной инвалидности в Америке. 87% случаев связаны с ишемическим инсультом, который возникает в результате закупорки артерии головного мозга, по которой течет богатая кислородом кровь. Нарушение кровотока означает, что мозг не может получать необходимые питательные вещества, такие как кислород и глюкоза, и удалять отходы, такие как углекислый газ. miRNA играют роль в посттрансляционном молчании генов, воздействуя на гены в патогенезе церебральной ишемии, такие как воспалительный, ангиогенез и апоптотический путь.
Алкоголизм
Жизненно важная роль miRNA в экспрессии генов важна для зависимости , в частности, от алкоголизма . Хроническое злоупотребление алкоголем приводит к стойким изменениям функции мозга, частично опосредованным изменениями экспрессии генов . miRNA глобальная регуляция многих нижестоящих генов считается значимой в отношении реорганизации или синаптических связей или долгосрочных нейронных адаптаций, включающих изменение поведения от потребления алкоголя к абстиненции и / или зависимости. Было обнаружено, что в посмертном мозге алкоголика изменено до 35 различных miRNA, все из которых нацелены на гены, которые включают регуляцию клеточного цикла , апоптоз , клеточную адгезию , развитие нервной системы и передачу сигналов клеток . Измененные уровни miRNA были обнаружены в медиальной префронтальной коре головного мозга алкоголезависимых мышей, что указывает на роль miRNA в управлении трансляционным дисбалансом и создании дифференциально экспрессируемых белков в той области мозга, где вероятно возникает сложное когнитивное поведение и принятие решений .
miRNA могут быть активированы или подавлены в ответ на хроническое употребление алкоголя. Экспрессия miR-206 увеличивалась в префронтальной коре головного мозга алкогольных крыс, воздействуя на нейротрофический фактор мозга ( BDNF ) фактора транскрипции и в конечном итоге снижая его экспрессию. BDNF играет критическую роль в формировании и созревании новых нейронов и синапсов, предполагая возможное участие в росте синапсов / синаптической пластичности у лиц, злоупотребляющих алкоголем. Было обнаружено, что miR-155, важный в регуляции вызванных алкоголем реакций нейровоспаления , активируется, что предполагает роль микроглии и воспалительных цитокинов в патофизиологии алкоголя. Подавление miR-382 было обнаружено в прилежащем ядре , структуре базального переднего мозга, которая играет важную роль в регулировании чувства вознаграждения, которое питает мотивационные привычки. miR-382 является мишенью для дофаминового рецептора D1 (DRD1), и его сверхэкспрессия приводит к усилению регуляции DRD1 и delta fosB , фактора транскрипции, который активирует серию событий транскрипции в прилежащем ядре, что в конечном итоге приводит к аддиктивному поведению. С другой стороны , с гиперэкспрессией микроРНК-382 в результате ослабленных питьевой и ингибирования DRD1 и дельта fosB повышающей регуляции в крысиных моделях алкоголизма, демонстрируя возможность использования микроРНКа-целевые лекарственных препаратов в лечении.
Ожирение
miRNAs играют решающую роль в регуляции дифференциации клеток- предшественников стволовых клеток в адипоциты . Исследования, чтобы определить, какую роль плюрипотентные стволовые клетки играют в адипогенезе , были изучены на иммортализованной линии стромальных клеток человеческого костного мозга hMSC-Tert20. Снижение экспрессии miR-155 , miR-221 и miR-222 было обнаружено во время адипогенного программирования как иммортализованных, так и первичных hMSC, что позволяет предположить, что они действуют как негативные регуляторы дифференцировки. Напротив, эктопическая экспрессия miRNA 155 , 221 и 222 значительно ингибировала адипогенез и подавляла индукцию главных регуляторов PPARγ и CCAAT / связывающего энхансера белка альфа ( CEBPA ). Это открывает путь к возможному лечению генетического ожирения.
Другой класс миРНК, регулирующих инсулинорезистентность , ожирение и диабет , — это семейство let-7 . Let-7 накапливается в тканях человека в процессе старения . Когда let-7 была эктопически сверхэкспрессирована, чтобы имитировать ускоренное старение, мыши становились инсулинорезистентными и, следовательно, более склонными к ожирению и диабету, вызванному диетой с высоким содержанием жиров . Напротив, когда let-7 ингибировалась инъекциями let-7-специфических антагомиров , мыши становятся более чувствительными к инсулину и заметно устойчивы к ожирению и диабету, вызванному диетой с высоким содержанием жиров. Подавление let-7 может не только предотвратить ожирение и диабет, но и обратить вспять и вылечить это состояние. Эти экспериментальные данные предполагают, что ингибирование let-7 может представлять собой новую терапию ожирения и диабета 2 типа.
Гемостаз
miRNA также играют решающую роль в регуляции сложных ферментативных каскадов, включая систему свертывания крови. Крупномасштабные исследования функционального нацеливания miRNA недавно обнаружили рациональные терапевтические мишени в системе гемостаза.
Некодирующие РНК
Когда в 1999 году в рамках проекта генома человека была нанесена на карту его первая хромосома , было предсказано, что геном будет содержать более 100 000 генов, кодирующих белок. Однако в конечном итоге было идентифицировано только около 20 000 человек. С тех пор появление биоинформатических подходов в сочетании с исследованиями разбиения генома, изучающими транскриптом, систематическим секвенированием библиотек полноразмерных кДНК и экспериментальной проверкой (включая создание антисмысловых олигонуклеотидов, полученных из микроРНК, называемых антагомирами ), показало, что многие транскрипты не кодируют белок. РНК, включая несколько snoRNA и miRNA.
Вирусы
Вирусные микроРНК играют важную роль в регуляции экспрессии генов вируса и / или генов хозяина, принося пользу вирусу. Следовательно, miRNAs играют ключевую роль во взаимодействиях вирус-хозяин и в патогенезе вирусных заболеваний. Считается, что экспрессия активаторов транскрипции ДНК вируса герпеса-6 человека регулируется вирусной miRNA.
Прогнозирование цели
miRNA могут связываться с транскриптами целевой матричной РНК (мРНК) генов, кодирующих белок, и отрицательно контролировать их трансляцию или вызывать деградацию мРНК. Ключевое значение имеет точное определение мишеней miRNA. Доступно сравнение прогнозирующей производительности восемнадцати алгоритмов in silico . Крупномасштабные исследования функционального нацеливания miRNA предполагают, что многие функциональные miRNA могут быть упущены с помощью алгоритмов прогнозирования мишеней.
Смотрите также
- Олигонуклеотиды против miRNA
- Экспрессия гена
- Список инструментов для прогнозирования генов miRNA
- Список инструментов для прогнозирования мишеней miRNA
- МикроДНК
- miR-324-5p
- РНК-интерференция
- Малая интерферирующая РНК
- МикроРНК, происходящая из малой ядрышковой РНК
использованная литература
дальнейшее чтение
- Определение и классификация miRNA: Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T (март 2003 г.). «Единая система аннотации микроРНК» . РНК . 9 (3): 277–9. DOI : 10,1261 / rna.2183803 . PMC 1370393 . PMID 12592000 .
- Научный обзор малых РНК: Baulcombe D (сентябрь 2002 г.). «События ДНК. Микромир РНК». Наука . 297 (5589): 2002–3. DOI : 10.1126 / science.1077906 . PMID 12242426 . S2CID 82531727 .
- Открытие lin-4 , первой обнаруженной миРНК: Lee RC, Feinbaum RL, Ambros V (декабрь 1993). «Гетерохронный ген lin-4 C. elegans кодирует малые РНК с антисмысловой комплементарностью lin-14» . Cell . 75 (5): 843–54. DOI : 10.1016 / 0092-8674 (93) 90529-Y . PMID 8252621 .
внешние ссылки
- База данных miRBase
- miRTarBase , экспериментально подтвержденная база данных взаимодействий микроРНК-мишень.
- semirna , Веб-приложение для поиска микроРНК в геноме растений.
- ONCO.IO : Интегративный ресурс по анализу микроРНК и факторов транскрипции при раке.
- MirOB : MicroRNA нацелена на базу данных и инструмент для анализа и обработки данных.
- База данных ChIPBase : база данных с открытым доступом для декодирования факторов транскрипции , которые участвовали в транскрипции микроРНК или влияли на нее, из данных ChIP-seq.
- Анимационный ролик о процессе биогенеза микроРНК .
- Реагенты для модуляции miRNA для активации или подавления функции эндогенной зрелой микроРНК
Синтетическое представление биосинтеза и функционирования микроРНК.
МикроРНК (аббревиатура: микроРНК или μ РНК ) представляют собой класс небольших (ен) рибонуклеиновых кислот (РНК) , одноцепочечной , некодирующей и клеточно-специфической эукариотических . Они имеют среднюю длину 22 нуклеотида (от 21 до 24 в целом), что меньше, чем у других РНК .
MiRNA являются посттранскрипционными регуляторами, способными подавлять экспрессию гена : их спаривание с целевой информационной РНК (мРНК) может приводить к ингибированию ее трансляции или к ее деградации в зависимости от степени комплементарности между последовательностью miRNA и этой последовательностью. его целевой мРНК. Поскольку они влияют на экспрессию многих генов, miRNA участвуют в большинстве биологических процессов. MiRNA участвуют во многих процессах, от развития до образования опухоли .
Человеческого генома будет содержать примерно 2000 генов в происхождении транскрипции микроРНК , которые будут ориентированы примерно 60% генов. Было продемонстрировано, что miRNA могут по-разному экспрессироваться от одной ткани или от одного типа клеток к другому; они в изобилии присутствуют в нескольких типах клеток человека .
Аберрантная экспрессия определенных miRNA связана с многочисленными патологиями; методы лечения, основанные на коррекции их обилия, в настоящее время изучаются.
Исторический
Первая miRNA, lin-4 (en) , была охарактеризована в начале 1990-х годов у нематоды Caenorhabditis elegans . Авторы этого исследования обнаружили, что эта малая РНК экспрессируется в конце первой стадии личиночного развития . Затем он связывается с мРНК белка LIN-14, ингибирует его синтез и тем самым индуцирует переход между первыми двумя личиночными фазами. lin-4 поэтому играет фундаментальную роль в контроле последовательности фаз развития.
Однако только в начале 2000-х и открытии let-7 (en) у C. elegans , а также др. MiRNAs у разных видов, посттранскрипционный регуляторный механизм с помощью miRNAs может быть установлен.
Биосинтез
Рисунок 1. Биосинтез miRNA у животных. MiRNA получают в ядре путем транскрипции или сплайсинга из интрона .
Происхождение и эволюция
MiRNA хорошо законсервированы у эукариотических организмов и считаются предковыми и существенными компонентами регуляции экспрессии генов . Путь miRNA у животных и растений может иметь различное происхождение и возник независимо. Считается, что путь miRNA происходит от более древней системы — РНК-интерференции . Ферменты Dicer особенно ответственны за производство малых интерферирующих РНК (пРНК) из длинных двухцепочечных РНК. Этот процесс, описанный в растении, действует как противовирусная защита.
Ядерная обработка
MiRNA канонически транскрибируются с помощью РНК-полимеразы типа II в виде длинных предшественников петли, называемых первичными miRNA ( pri -miRNA) (Рисунок 1, слева) . Эти при-миРНК затем расщепляются гетеротримерным белковым комплексом, называемым «микропроцессором» (en) , образованным, в частности, из молекулы рибонуклеазы Дроша III (en) и двух молекул ее партнера DGCR8 (en) ( Di George синдром Критической области 8 — называется Паша у дрозофилы и нематод). Таким образом образуется промежуточный продукт, называемый предшественником миРНК ( пре- миРНК).
Некоторые miRNA не подвергаются лечению Drosha. В некоторых случаях пре-миРНК продуцируются сплайсосомой из интронов во время сплайсинга и затем называются «миРтронами» (рис. 1, справа) . В других случаях пре-миРНК продуцируются из тРНК или из малых ядрышковых РНК (мяРНК для малых ядрышковых РНК ).
Пре-миРНК имеет длину около 60 нуклеотидов и свернута в несовершенную стебель-петлю за счет комплементарности оснований между первой половиной и второй половиной ее последовательности.
Пре-миРНК активно транспортируются, в зависимости от GTP , из ядра в цитозоль с помощью экспортина 5 (en) .
Цитоплазматическое лечение
Пре-миРНК расщепляется ферментным комплексом, состоящим из члена семейства рибонуклеаз III Dicer и его белкового партнера TRBP (называемого у Drosophila локвативным). Белок Dicer распознает два нуклеотида 3′- когезионного конца, используя свои платформенные и белковые домены PAZ. Дайсер действует как линейка, измеряя 22 нуклеотида от 5 ‘или 3’ конца, тем самым определяя сайт расщепления пре-миРНК. Пре-миРНК с относительно нестабильным 5 ‘концом (несовпадения, пары G — U или A -U) помещаются в 5’ карман платформенного домена Дайсера. Затем сайт расщепления определяется с 5 ‘конца, это называется правилом подсчета 5′ . Когда 5′-конец стабилен (пара G- C ), это 3′-конец пре-miRNA, который взаимодействует с 3′-карманом Dicer, расположенным в домене PAZ; в этом случае сайт расщепления расположен в 22 нуклеотидах от 3 ‘конца (правило счета 3’). Дайсер позволяет гидролиз петлевой структуры пре-миРНК и высвобождает зрелую миРНК с 3 ‘когезионными концами, называемую «дуплексная миРНК / миРНК * (читай звездчатая миРНК )» — или миРНК (дб для двухцепочечной) — с направляющая прядь и проходная прядь (рисунок 1) .
Существует по крайней мере одно исключение из созревания miRNAs с помощью Dicer, такое как miR-451 (en) , специфическая miRNA позвоночных, участвующая в эритропоэзе . В данном случае это белок AGO2 (из семейства Argonaute), который расщепляет структуру «стебель-петля» в середине 3′-плеча. Затем рибонуклеаза ( PARN ) разрезает в направлении от 3 ‘до 5’ с образованием направляющей цепи miR-451.
Способ действия
Рисунок 2. Схематическое изображение канонического взаимодействия между miRNA и ее мишенью, в данном случае мРНК. MRNA обычно имеет кэп , открытую рамку считывания (ORF), область 3′-UTR и поли (A) хвост . MiRNA взаимодействует с доменом области 3’UTR мРНК, называемым элементом ответа miRNA, в первую очередь через «затравочную» область.
Одна нить дуплекса миРНК / микроРНК * взаимодействует с белком семейства Argonaute (AGO1 4) для формирования комплекса «RISC» ( R NA- я nduced с ilencing с омплекс ) иногда называют «miRISC» (рисунок 3) . Белки семейства Argonaute имеют домен PAZ и домен PIWI. У млекопитающих miRNAs, по-видимому, загружаются безразлично одним из четырех членов семейства Ago. Это не так у мух, где есть только два Ago и где miRNAs преимущественно ориентированы в сторону AGO1.
Комплекс RISC, имеющий приблизительно 160 кДа, был описан как достаточный для подавления активности генов miRNA, однако другие белки, такие как геминин, могут объединяться с ним, образуя комплексы до 550 кДа . Комплекс RISC связывается с другими партнерами, такими как белки семейства GW182, которые, в свою очередь, привлекают другие эффекторы, участвующие, в частности, в разрушении поли (A) -хвоста, устранении 5′-кэпа или вмешательстве в инициацию трансляции. целевой мРНК.
Рисунок 3. Схематическое изображение двух посттранскрипционных регуляторных путей экспрессии генов с помощью miRNA. В зависимости от степени комплементарности между miRNA и ее мишенью miRISC индуцирует: либо ингибирование трансляции, либо деградацию мРНК.
Во время образования miRISC происходит изменение ds miRNA на miRNA (sb = одноцепочечный). Выбор цепи, связанной с miRISC, осуществляется в соответствии с двумя основными правилами, при этом обычно основной путь может сосуществовать с второстепенным путем. Внутри белка Ago домен MID играет центральную роль во взаимодействии с 5′-концом направляющей цепи. Выбор цепи Ago основан на нестабильности 5′-конца miRNA, что делает ее более доступной для домена MID белка. С другой стороны, Ago способствует присутствию нуклеотида A или U на 5′-конце. Как только miRNA загружена, белок AGO представляет нуклеотиды 2-8 направляющей цепи по направлению к внешней стороне молекулы.
Затем целевая мРНК загружается в комплекс miRISC в соответствии с правилом комплементарности оснований с miRNA. Однако гибридизация miRNA с их мишенью предпочтительно определяется ограниченной областью miRNA, простирающейся от нуклеотидов 2 до 7 и называемой «затравкой» (фиг. 2) . МикроРНК может быть нацелена на несколько сайтов на одной и той же мРНК или на несколько целевых мРНК, имеющих сходную последовательность распознавания, называемую элементом ответа на miРНК (MRE, для м- IRNA R esponse E Elements e ) (Рисунок 2) . У многоклеточных животных сайт гибридизации преимущественно расположен в 3′-нетранслируемой области (3’UTR) .
Тогда возможны два пути « сайленсинга » : либо эндонуклеолитическое расщепление целевой мРНК, если важна комплементарность с миРНК, либо репрессия трансляции, связанная с экзонуклеолитической деградацией мРНК, в случае, когда комплементарность между миРНК и мРНК несовершенный (рисунок 3) . Однако деградация мРНК, по-видимому, доминирует над ингибированием трансляции.
В растениях
Есть некоторые различия между биосинтезом и механизмом действия miRNAs у растений и у многоклеточных . В растениях количество miRNA было бы более ограниченным, и не было бы промежуточных продуктов pri-miRNA (рисунок 4) . Образование дуплекса miRNA / miRNA * с помощью DCL1 (en) ( Dicer-like 1 , гомолог Dicer) происходит в ядре ; зрелый дуплекс затем транспортируется в цитоплазму с помощью экспортина HASTY. Кроме того, посттранскрипционная репрессия обычно включает идеальную или почти идеальную гибридизацию между miRNA и ее целевой мРНК, она вызывает расщепление мРНК в середине сайта гибридизации miRNA. Наконец, у растений, если сайт гибридизации с miRNA можно найти в области 3’UTR мРНК, он чаще всего находится в кодирующей последовательности .
В некоторых случаях, как и в моховых Physcomitrella patens , накопление микроРНК дуплекс: РНК — мишени , могут участвовать в образовании комплекса под названием «РИЦ» (В) ( R NA-индуцированной я nitiation из T ranscriptional гена сек ilencing ) , который, связывание непосредственно с ДНК должно вызывать ее метилирование, приводящее к «исчезновению» мишеней miRNA. Не исключено, что такой процесс существует у животных .
Нормативно-правовые акты
MiRNA регулируются транскрипцией, как гены, кодирующие белки. Миртроны, в свою очередь, регулируются транскрипцией так же, как их гены-хозяева.
Функции
MiRNA участвуют в большом количестве важных физиологических функций, таких как:
- рост и дифференциация клеток ,
- апоптоз ,
- обмен веществ ,
Обучение и память
Контекст
Долговременная память зависит от экспрессии гена , белка и структурных модификаций нейронов . Кроме того, в его основе лежат механизмы синаптической пластичности на уровне небольшого числа синапсов, специально участвующих в модификации запоминаемого поведения . Экспрессия гена вызвана формированием долговременной памяти, происходящей на ядерном уровне , возникает вопрос, транспортируется ли продукт экспрессии гена в несколько синапсов, участвующих в запоминаемом поведении, или он распределяется однородно. в нейроне, а затем переводится локально. Это основные вопросы в изучении биологии памяти. Локальная трансляция мРНК, расположенных на уровне аксонов и дендритов , и зависящая от нейрональной активности, позволила бы локально усилить синаптическую эффективность и, таким образом, создать «след» обучения и сохранить его в памяти.
Считается, что miRNA играют центральную роль в этом процессе. МРНК будут экспрессироваться и распределяться однородным образом во всех нейронах, но «заблокированы» ( заблокированы ) miRNAs и комплексом RISC . Во время определенной активности на уровне синапса комплекс RISC будет выделять мРНК, которые затем транслируются локально. Транслируемые таким образом белки будут участвовать в модуляции синаптической эффективности и позволят формировать локальный след памяти. Некоторые процессы могут приводить к локальной модуляции синтеза белка: созревание определенных miRNA, высвобождение заблокированных мРНК или даже деградация определенных miRNA.
Вовлечение комплекса RISC
Первая команда , которая задействована RISC комплекс в формировании долговременной памяти используется классический кондиционер в уксусе лета . В этой модели запоминания мухи учатся ассоциировать запах (условный раздражитель) с легким электрошоком (безусловный раздражитель). Когда мухи подвергаются воздействию этого запаха, они будут стремиться уйти от него, и можно будет оценить их память, проанализировав их поведение . Одним из преимуществ обонятельной системы является то, что каждый запах активирует определенные области антеннальных долей в них (что эквивалентно обонятельным луковицам у млекопитающих).
Авторы исследования продемонстрировали, что протокол, индуцирующий долговременную память, стимулировал транспорт мРНК от CaMKII , белка, важного для памяти, к дендритам и индуцировал локальный синтез белка . Область 3′-UTR CaMKII, а также компоненты (тяжелая цепь кинезина , Staufen ) механизма транспорта мРНК содержат сайты-мишени miRNA (miR-280, miR-289 и miR-305 (in) ). Обонятельное обучение вызывает деградацию компонента, связанного с комплексом RISC — Armitage, ортолога Mov10l1 (en) у позвоночных — протеасомой , особенно в областях, активируемых изученным запахом. Эта деградация Armitage дестабилизирует RISC-комплекс, высвобождая мРНК, которые он блокирует, обеспечивая их трансляцию и участвуя в формировании долгосрочного следа обучения в сетях антенной доли в форме модификации синаптической эффективности.
В результате этого исследования остается ряд вопросов. В самом деле, авторы не вмешиваются (путем инактивации или сверхэкспрессии) в идентифицированные miRNAs, которые, если они теоретически могут связываться с областью 3’UTR CaMKII, не предполагают, что они действительно обладают репрессивным действием в физиологических условиях. Более того, белок Armitage участвует в физиологии другого типа РНК, РНК, взаимодействующих с Piwi ( piRNA ), предполагая, что другой тип РНК может участвовать в процессе обучения и запоминания.
Вовлечение комплекса transline / trax
Совсем недавно был описан другой механизм. Комплекс transline (en) / trax (en) может быть задействован активированными синапсами и участвовать в деградации miRNA, тем самым локально высвобождая экспрессию белка. Связывания РНК сродство к translin модулируется с помощью фосфорилирования, что позволяет ему связываться с мРНК мишеней, кроме того, он связывается с актином позволяя ему перемещаться вдоль цитоскелета . В гиппокампе после обучения транслинация перемещается в синаптическое пространство (собственно синаптосомы (in) ). Комплекс transline / trax, обладающий эндонуклеолитической активностью, благодаря trax, будет разрушать несколько miRNA, включая let-7c (en) , miR-125b и miR-128. Все эти miRNA участвуют в ингибировании экспрессии рецептора активина ( ACVR1C (en) ), их деградация высвобождает трансляцию этого рецептора и, таким образом, способствует усилению синаптической эффективности в связи с долгосрочным запоминанием.
Специфические миРНК
Несколько miRNA были более специфически вовлечены в хранение, такие как miR-124 (en) , miR-128b, miR-132 (en) , miR-134 (en) , miR-181a (en) или miR-182.
МиР-124
MiR-124 — первая miRNA, которая участвует в процессе синаптической пластичности, связанной с обучением и запоминанием. Это эволюционно высококонсервативная миРНК , наиболее распространенная в центральной нервной системе и изначально известная своей ролью в дифференцировке нейронов. Команда P Dr. Eric Кандель изучает то форму синаптической пластичности , известной как сенсибилизации в моллюска, на морского зайца . Во время этого явления, то рефлекс отвод из жабр , во время нейтрального сенсорного стимула на сифоне , усиливают с помощью предыдущего ноцицептивного стимула на другую часть тела. Этот стимул приводит к высвобождению серотонина , нейромедиатора , на сенсорных нейронах, тем самым усиливая их реакцию.
Команда на Р д — р Кандель показала , что повторное применение серотонина вызывало быстрое уменьшение обилия MIR-124 в сенсорных нейронах. Это MAPK- зависимое снижение высвобождает экспрессию белка CREB1 , фактора транскрипции, который играет центральную роль в формировании долговременной памяти. Затем CREB1 индуцирует экспрессию непосредственных ранних генов (en), участвующих в нейрональных изменениях, поддерживающих запоминание. Авт. Пришли к выводу, что miR-124 является одним из многих «замков», которые необходимо снять при формировании долговременной памяти, и что это очень регулируемый механизм.
MiR-181a, местное созревание и зависит от активности нейронов
В 2017 г.Команда D г Erin Шуман (в) — нейробиологе специализирующаяся на местном синтезе белков и в настоящее время директор Института Макса Планка по изучению мозга — опубликовано в журнале Science исследование показывает , что микроРНК может контролировать экспрессию белка в одном синапсе.
Авт. Впервые продемонстрировали, что предшественник miR-181a (en) (pre-miR-181a) сильно экспрессируется в дендритах нейронов в области CA1 гиппокампа , области мозга, играющей центральную роль в формировании памяти . Используя флуоресцентный зонд, позволяющий визуализировать расщепление пре-miRNA на зрелую miRNA, они показали, что нейрональная активность в форме потенциалов действия индуцирует созревание miR-181a. Это созревание осуществлялось Дайсером, активность которого стимулировалась увеличением внутриклеточной концентрации через глутаматные рецепторы типа NMDA . Используя другую технику, называемую нейротрансмиттерами «в клетке», авторы высвобождали глутамат путем фотостимуляции в непосредственной близости от одного синапса, чтобы активировать только этот. Таким образом, они продемонстрировали, что miR-181a созревает только в этом синапсе и что она ингибирует экспрессию CaMKII только в этом синапсе. Этот механизм, таким образом, позволит контролировать экспрессию генов на уровне каждого синапса индивидуально и, таким образом, участвовать в механизмах локальной синаптической пластичности , связанных с формированием памяти.

Спорный
Было высказано предположение, что миРНК, полученные из пищи, такой как рис, могут быть обнаружены в организме хозяина и влияют на его метаболизм, поэтому их можно рассматривать как новый класс питательных микроэлементов, таких как витамины , фитогормоны и другие фитонутриенты . Однако это может показаться неожиданным, поскольку нуклеиновые кислоты обычно гидролизуются до нуклеотидов (моно, ди и три) нуклеазами поджелудочной железы . Ферменты щеточной каймы (нуклеозидазы и фосфатазы ), в свою очередь, гидролизуют нуклеотиды до азотистых оснований и пентоз, прежде чем последние проникнут в энтероциты, подавляя таким образом последовательность miRNA и тем самым их информативный характер. Гипотеза впоследствии была опровергнута, первое исследование сначала оспаривало воспроизводимость результатов, а затем второе продемонстрировало, что использование инструментов секвенирования нового поколения , чрезвычайно чувствительных, обнаружило только заражение образцов от хозяина миРНК растений.
Это противоречие могло бы показаться анекдотическим, если бы оно не отражало основные проблемы научных исследований в настоящее время. Существует , прежде всего, необходимость воспроизводимости из экспериментов , то необходимо установить соответствующие контрольные группы . Более того, это подпитывает дискуссию, которая долгое время волновала биологию эволюции и которая может быть сведена к «противостоянию» теорий Чарльза Дарвина и Жана-Батиста де Ламарка . Короче говоря, одна из проблем заключается в том, чтобы определить влияние, которое окружающая среда («внешние обстоятельства») может оказывать на генетическое наследие, для регистрации в нем и в какой степени это влияние может передаваться от одного поколения к другому ». Другие. Дискуссия возобновляется неоднократно из-за идеологий, таких как Lysenkism , или по случаю новых открытий, таких как недавнее развитие эпигенетики , в которой участвуют miRNAs.
Другие роли
Митохондриальный
Центральная роль митохондрий в метаболизме клеток означает, что они подвержены тонкой и реактивной регуляции. Гены, важные для функции митохондрий и расположенные в ядре , являются мишенью регуляции miRNA, в частности miR-210 ( fr ) . Другие связи между miRNA и митохондриями были предложены открытием miRNA, расположенных в митохондриях и названных «mitomiR», а также присутствием белка AGO2. Неожиданно было продемонстрировано, что miR-1 (en) во время дифференцировки мышечных клеток проникает в митохондрии, где стимулирует трансляцию митохондриальных генов, вместо того, чтобы репрессировать ее, как мы ожидали от miRNA. Несколько других исследований подтвердили присутствие miRNA в митохондриях, а также присутствие компонентов комплекса RISC, и, таким образом, подтверждают идею о том, что mitomiR влияют на метаболизм митохондрий и имеют потенциальное патофизиологическое воздействие. Поэтому была предложена возможность использования регуляции экспрессии miRNA для прямой модуляции гомеостаза и функции митохондрий, и в этом направлении был подан патент.
Популярный
Несколько команд изучали влияние miRNA на функционирование вирусов . В самом деле, определенные miRNA клеток-хозяев могли бы быть способны нацеливаться на вирусные РНК, придавая miRNA антивирусную роль, или, наоборот, позволяли бы вирусу накапливаться в клетке. Эта ситуация была описана , в частности , для MIR-122 , который специфически экспрессируется в печени , это будет способствовать репликации РНК из гепатита В и С вирусами .
Патологии
Bilharzia
В шистосомозных являются червь паразит вызывает заболевание под названием шистосомоз или шистосомоз . Шистосомные инфекции носят хронический характер и могут длиться десятилетиями, что свидетельствует о том, что паразит изменяет иммунную систему своего хозяина, так что он не уничтожается.
Недавнее исследование показывает, что Schistosoma japonicum , один из пяти патогенных видов , будет использовать miRNA для отклонения иммунного ответа своего хозяина и, таким образом, облегчения его выживания в своем хозяине. Авторы этого исследования показали, что S. japonicum высвобождает внеклеточные везикулы (en), содержащие miRNA, в основном miR-125b и bantam. Их результаты предполагают, что miRNA интернализуются периферическими иммунными клетками, макрофагами . После интернализации эти miRNA будут модифицировать экспрессию многих генов в организме хозяина и тем самым увеличивать пролиферацию макрофагов, а также продукцию TNF-α . Авторы предполагают, что эти механизмы позволяют S. japonicum изменять иммунную систему хозяина, и демонстрируют, что уменьшение количества моноцитов, а также TNF-α — путем инъекции клодроната — снижает бремя шистосом и образование гранулем печени.
Хотя гипотеза авторов привлекательна, до недавнего времени сохранялись споры относительно функциональной роли передачи miRNA внеклеточными пузырьками. Действительно, количество передаваемых miRNA настолько низкое, что они не могут иметь физиологического действия, хотя они обнаруживаются чрезвычайно чувствительными методами секвенирования нового поколения .
Сердечный
Некоторые микро-РНК многочисленны ( miR-1 (en) , miR-133a , miR-499) или экспрессируются специфически в мышцах и, в частности, в сердце ( miR-208a (en) ). Возникновение инфаркта миокарда приведет к их выбросу в кровоток во время гибели клеток ; поэтому их количественная оценка может использоваться в качестве раннего плазменного маркера начала инфаркта.
Нервной системы
Несколько нейродегенеративных заболеваний ( болезнь Альцгеймера , Паркинсона , Хантингтона болезнь ) связаны с изменениями в численности некоторых специфических микроРНК ( микроРНК-34 (ен) , микроРНК-124 (ен) и микроРНК-132 (ен) ).
Болезнь Альцгеймера
Болезнь Альцгеймера — самая частая причина деменции у пожилых людей. Это прогрессирующее нейродегенеративное заболевание, характеризующееся, в частности, на ранней стадии потерей памяти , когнитивным и интеллектуальным дефицитом. На клеточном уровне заболевание характеризуется потерей нейронов , внеклеточным накоплением β-амилоидного пептида и внутриклеточным накоплением тау-белка, который может нарушать нормальное функционирование нейронов, в частности синаптическую пластичность .
Изобилие miR-132 сильно снижено в головном мозге пациентов с болезнью Альцгеймера, а точнее в структурах, участвующих в запоминании, таких как гиппокамп , префронтальная и височная кора и мозжечок . MiR-132 будет регулировать многие гены ( MeCP2 (en) , SIRT1 ), участвующие в молекулярных механизмах обучения и запоминания , поэтому его дерегуляция может объяснить часть дефицита, связанного с болезнью Альцгеймера.
MiR-124 — еще одна миРНК, количество которой снижено у пациентов с этим заболеванием. Эта miRNA д. Поддерживать идентичность нейронов и синаптическую пластичность, поддерживающую долгосрочную память (см. Выше), ее дерегуляция будет иметь вредные последствия для этих механизмов.
Экспрессия miR-181a значительно увеличена в гиппокампе мышей «3xTg-AD», мышиной модели болезни Альцгеймера. Это увеличение связанно с уменьшением экспрессии белков , ассоциированных с синаптической пластичностью ( GluA1 (о) и GluA2 (о) субъединицей из АМРА , PSD95 (о) и Transline (о) рецепторов ). Однако ингибирование miR-181a способно восстановить экспрессию GluA1 и GluA2 и улучшить производительность пространственной памяти в тесте местоположения объекта.
| миРНК | Регулирование | Причины | Цели | Функции | Рекомендации |
|---|---|---|---|---|---|
| miR-132 | ↓ | ? (CREB, ОТДЫХ) | MeCP2, SIRT1 | Выживание нейронов, синаптическая пластичность, память и обучение | |
| miR-124 | ↓ | ? (КАРТА, ОТДЫХ) | PTBT1, Cdc42, Rac1, Zif268, Glu2A | Поддержание нейрональной идентичности, синаптической пластичности | |
| miR-34 | ↑ | p53, SIRT1 | VAMP2, Notch1, mGluR7, ARC, синаптотагмин, синтаксин | Нейронная активность, память и обучение | |
| miR-181a | ↑ | Transline | GluA1 и GluA2 | Синаптическая пластичность, память и обучение |
Некоторые циркулирующие miRNA ( miR-9 (en) , miR-125b, miR-146 (en) , miR-181c (en) , let-7g-5p (en) и miR-191-5p (en) ) могут быть используется в качестве ранних биомаркеров болезни Альцгеймера. Эти миРНК находятся в свободной форме в жидкостях или инкапсулированы в экзосомы .
болезнь Паркинсона
Циркулирующие миРНК также могут использоваться в качестве биомаркеров болезни Паркинсона .
Опухоли
Мутация или нарушение регуляции определенных miRNA, по-видимому, прямо или косвенно является причиной большого количества опухолей . Те, кто непосредственно вызывает механизм инициации рака , называются «онкомириями» (in) . Та же самая miRNA, такая как miR-22 , по-видимому, иногда может быть онкогенной, а иногда, наоборот, способствовать подавлению опухоли. Многие miRNA постоянно нарушают регуляцию в солидных опухолях. Например, в опухолях печени часто наблюдается избыточная экспрессия miR-21 и недостаточная экспрессия miR-122 . Кроме того, существует нарушение регуляции экспрессии miRNA, связанное с факторами риска гепатоцеллюлярной карциномы .
Определенные типы микроРНК также присутствуют в плазме крови , такие как MIR-92а (ен) , и может быть хорошими биомаркеры для лейкемии .
Следовательно, по-видимому, существуют специфические пути канцерогенеза для miRNA, которые затем будут действовать как факторы риска; это указывает на весь интерес miRNA как диагностических, прогностических или даже терапевтических маркеров.
Исследование показывает, что рак центральной нервной системы , в частности глиобластомы , можно диагностировать на ранней стадии путем анализа микроРНК, содержащихся в моче пациентов. Команда исследователей разработала легко промышленное устройство, состоящее из сотен миллионов нановолокон оксида цинка , которое собирает микроРНК в достаточных количествах.
Вирусный онкогенез
Похоже, что некоторые ретровирусы, обладающие онкогенными свойствами, имеют в своем геноме миРНК. Эти миРНК после их транскрипции могут участвовать в репрессии вирусных РНК и / или РНК клетки-хозяина и, таким образом, участвовать в прогрессии опухоли. Таким образом, проблема будет заключаться в том, чтобы лучше идентифицировать гены, на которые нацелены эти miRNA, чтобы лучше понять их роль и последствия их дерегуляции в процессе онкогенеза.
Терапевтические мишени
Определенное количество ингибиторов миРНК находится в стадии разработки, они по существу представляют собой короткие цепи РНК, антисмысловые олигонуклеотиды, называемые « антагомирами », присоединяющиеся посредством комплементарности к части микроРНК-мишени. Вектор, который позволил бы их введение, был бы непатогенным вирусом, который вводил бы нуклеотидную последовательность в клетку.
Введение miRNA в терапевтических целях непросто, потому что они разлагаются до того, как достигают своей цели. Использование блокированных нуклеиновых кислот — конформационных аналогов нуклеотидов, но относительно более устойчивых к нуклеазам — может быть использовано при разработке терапевтических решений на основе миРНК.
Таким образом, проводятся испытания для лечения гепатита С и синдрома Альпорта .
Примечания и ссылки
-
↑ a b и c (en) Дэвид П. Бартель, « МикроРНК многоклеточных животных » , Cell , vol. 173, п о 1,2018 г., стр. 20-51 ( PMID 29570994 , DOI 10.1016 / j.cell.2018.03.006 , читать онлайн [PDF] , по состоянию на 15 марта 2020 г. ) редактировать
-
↑ (in) Д. Бартель , » МикроРНК: распознавание мишеней и регуляторные функции « , Cell , vol. 136, п о 22009 г., стр. 215–233 ( PMID 19167326 , PMCID 3794896 , DOI 10.1016 / j.cell.2009.01.002 , читать онлайн , по состоянию на 15 марта 2020 г. ) редактировать
-
↑ (in) Б. Кусенда, г-н Мраз , Дж. Майер и С. Посписилова , « Биогенез микроРНК, функциональность и актуальность рака » , Биомедицинские документы , вып. 150, п о 22006 г., стр. 205–215 ( PMID 17426780 , DOI 10.5507 / bp.2006.029 , читать онлайн [PDF] , по состоянию на 28 апреля 2020 г. ) редактировать
- ↑ (in) Huntzinger Izaurralde E and E, « Генное молчание с помощью микроРНК: вклад репрессии трансляции и распада мРНК » , Nat Rev Genet. , т. 12, п о 22011 г., стр. 99-110 ( PMID 21245828 , DOI 10.1038 / nrg2936 , читать онлайн , по состоянию на 16 марта 2020 г. ) редактировать
-
↑ а и б (в) Клоостерман и Пластерк WP HR. « Разнообразные функции микроРНК в развитии и болезнях животных. » , Dev Cell. , т. 11, п о 4,2006 г., стр. 441-50 ( PMID 17011485 , DOI 10.1016 / j.devcel.2006.09.009 , читать онлайн [EPUB] , по состоянию на 16 марта 2020 г. ) редактировать
-
↑ (in) Адамс Б.Д., А.Л. Касински и Слэк Ф.Д., « Аберрантная регуляция и функция микроРНК при раке. « , Curr Biol. , т. 24, п о 16,2014 г., стр. 762-76 ( PMID 25137592 , DOI 10.1016 / j.cub.2014.06.043 , читать онлайн [PDF] , по состоянию на 16 марта 2020 г. ) редактировать
- ↑ miRNAs Homo sapiens в miRBase в Манчестерском университете
- ↑ (ru) Бентвич I, Авниэль А., Каров Ю., Ааронов Р., Гилад С., Барад О., Барзилай А., Эйнат П., Эйнав Ю., Мейри Е., Шарон Е., Спектор И. и Бентвич З., « Идентификация сотен сохраненных и несохраняемых человеческие микроРНК. » , Нат Генет. , т. 37, п о 7,2005 г., стр. 766-70 ( PMID 15965474 , DOI 10.1038 / ng1590 , читать онлайн , по состоянию на 15 марта 2020 г. ) редактировать
-
↑ (ru) Lewis BP, Burge CB и Bartel DP., « Консервативное спаривание семян, часто фланкированное аденозинами, указывает на то, что тысячи человеческих генов являются мишенями для микроРНК. ” , Cell , vol. 120, п о 1,2005 г., стр. 15-20 ( PMID 15652477 , DOI 10.1016 / j.cell.2004.12.035 , читать онлайн [PDF] , по состоянию на 15 марта 2020 г. ) редактировать
-
↑ (ru) Фридман Р.К., Фарх К.К., Бердж С.Б. и Бартел Д.П., « Большинство мРНК млекопитающих являются консервативными мишенями для микроРНК. ” , Genome Res. , т. 19, п о 1,2009 г., стр. 92-105 ( PMID 18955434 , DOI 10.1101 / gr.082701.108 , читать онлайн [PDF] , по состоянию на 16 марта 2020 г. ) редактировать
- ↑ a и b (in) Хе-Ин Им и Пол Дж. Кенни, « МикроРНК в функции и дисфункции нейронов » , Trends Neurosci , vol. 35, п о 5,2012 г., стр. 325-34 ( PMID 22436491 , DOI 10.1016 / j.tins.2012.01.004 , читать онлайн [PDF] , по состоянию на 14 марта 2020 г. ) редактировать
- ↑ Эта публикация описывает открытие lin-4 , первой miRNA, обнаруженной у C.elegans . (ru) Ли Р.К., Фейнбаум Р.Л. и Амброс В. « Гетерохронный ген lin-4 C. elegans кодирует малые РНК с антисмысловой комплементарностью lin-14. ” , Cell , vol. 75, п о 5,1993 г., стр. 843-54 ( PMID 8252621 , DOI 10.1016 / 0092-8674 (93) 90529-y , читать онлайн [PDF] , по состоянию на 17 марта 2020 г. ) редактировать
- ↑ (ru) Lagos-Quintana M, R Rauhut, Lendeckel W., Tuschl T. « Идентификация новых генов, кодирующих малые экспрессируемые РНК. » , Наука , т. 294, п о 5543,2001 г., стр. 853-8 ( PMID 11679670 , DOI 10.1126 / science.1064921 , читать онлайн , по состоянию на 17 марта 2020 г. ) редактировать
- ↑ (in) Axtell MJ, OJ Westholm and Lai EC. « Vive la разница: биогенез и эволюция микроРНК у растений и животных. « , Genome Biol. , т. 12, п о 4,2011 г., стр. 221 ( PMID 21554756 , PMCID PMC3218855 , DOI 10.1186 / gb-2011-12-4-221 , читать онлайн [PDF] , по состоянию на 13 апреля 2020 г. ) редактировать
- ↑ (in) Шабалина С.А., Кунин Е.В., « Происхождение и эволюция эукариотической РНК-интерференции. » , Trends Ecol Evol. , т. 23, п о 10,2008 г., стр. 578-87 ( PMID 18715673 , PMCID PMC2695246 , DOI 10.1016 / j.tree.2008.06.005 , читать онлайн [PDF] , по состоянию на 25 марта 2020 г. ) редактировать
- ↑ В этой публикации обсуждаются процессы, в которых участвуют миРНК и микроРНК, в контексте двух статей, опубликованных в журнале Science . (ru) Баулкомб Д., « События ДНК. Микромир РНК. » , Наука. , т. 297, п о 5589,2002 г., стр. 2002-3 ( PMID 12242426 , DOI 10.1126 / science.1077906 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ (in) Ван М.Б. и г-н Метцлафф, « Молчание РНК и противовирусная защита растений. « , Curr Opin Plant Biol. , т. 8, п о 22005 г., стр. 216-22 ( PMID 15753004 , DOI 10.1016 / j.pbi.2005.01.006 , читать онлайн , по состоянию на 22 марта 2020 г. ) редактировать
- ↑ (in) Денли AM, BB Tops, Plasterk RH, Ketting RF и Hannon GJ, « Обработка первичных микроРНК с помощью микропроцессорного комплекса. » , Природа , т. 432, п о 7014,2004 г., стр. 231-5 ( PMID 15531879 , DOI 10.1038 / nature03049 , читать онлайн , по состоянию на 16 марта 2020 г. ) редактировать
- ↑ (in) Okamura K, Hagen JW, Duan H, Tyler DM и Lai EC, « Путь миртрон генерирует регуляторные РНК класса микроРНК у дрозофилы. ” , Cell , vol. 130, п о 1,2007 г., стр. 89-100 ( PMID 17599402 , DOI 10.1016 / j.cell.2007.06.028 , читать онлайн , по состоянию на 17 марта 2020 г. ) редактировать
- ↑ (in) Haussecker D, Huang Y, Lau A, P Parameswaran, AZ Fire и Kay MA., « Небольшие РНК, полученные из тРНК человека, в общей регуляции молчания РНК. » , РНК , т. 16, п о 4,2010 г., стр. 673-95 ( PMID 20181738 , PMCID PMC2844617 , DOI 10.1261 / rna.2000810 , читать онлайн [PDF] , по состоянию на 7 апреля 2020 г. ) редактировать
- ↑ (en) Эндер С., Крек А., Фридлендер М.Р., М. Бейтцингер, Вайнманн Л., Чен В., С. Пфеффер, Раевски Н. и Г. Мейстер, « snoRNA человека с функциями, подобными микроРНК. « , Mol Cell. , т. 32, п о 4,2008 г., стр. 519-28 ( PMID 19026782 , DOI 10.1016 / j.molcel.2008.10.017 , читать онлайн [PDF] , по состоянию на 7 апреля 2020 г. ) редактировать
-
↑ (in) Лю З., Ван Дж., Ченг Х., Ке Х, Сунь Л., Чжан и Ван Х.В. КК., « Крио-ЭМ структура человеческого дайсера и его комплексов с субстратом пре-миРНК. ” , Cell , vol. 173, п о 5,2018 г., стр. 1191-1203 ( PMID 29706542 , DOI 10.1016 / j.cell.2018.03.080 , читать онлайн [PDF] , по состоянию на 16 июня 2020 г. ) редактировать
-
↑ a и b (in) Park I, Heo I, Tian Y, Simanshu DK, Chang H, Jee D. Patel DJ и Kim VN., « Дайсер распознает 5 ‘конец РНК для эффективной и точной обработки. » , Природа , т. 475, п о 73552011 г., стр. 201-5 ( PMID 21753850 , PMCID 4693635 , DOI 10.1038 / nature10198 , читать онлайн [PDF] , по состоянию на 17 июня 2020 г. ) редактировать
- ↑ a и b (in) MacRae IJ, Zhou K, Li F, Repic A YEAR Brooks, WZ Cande, Adams PD и Doudna JA., « Структурная основа для процессинга двухцепочечной РНК с помощью Dicer. » , Наука , т. 311, п о 5758,2006 г., стр. 195-8 ( PMID 16410517 , DOI 10.1126 / science.1121638 , читать онлайн , по состоянию на 17 июня 2020 г. ) редактировать
-
↑ (ru) Cheloufi S, CO Dos Santos, Chong MM и Hannon GJ., » Дайсер-независимый путь биогенеза miRNA, который требует катализа Ago. » , Природа , т. 465, п о 7298,2010 г., стр. 584-9 ( PMID 20424607 , PMCID 2995450 , DOI 10.1038 / nature09092 , читать онлайн [PDF] , по состоянию на 11 апреля 2020 г. ) редактировать
-
↑ (in) Cifuentes D. Xue H, Taylor DW, Patnode H, Mishima Y, Cheloufi S, My E Mane S, Hannon GJ Lawson ND, Wolfe SA и Giraldez AJ., « Новый путь процессинга miRNA, независимый от Dicer, требует каталитического действия Argonaute2. Мероприятия. » , Наука , т. 328, п о 5986,2010 г., стр. 1694-8 ( PMID 20448148 , PMCID 3093307 , DOI 10.1126 / science.1190809 , читать онлайн [PDF] , по состоянию на 13 апреля 2020 г. ) редактировать
-
↑ (ru) Йода М., Сифуэнтес Д., Изуми Н., Сакагучи Ю., Сузуки Т., Гиралдез А. Дж. И Ю. Томари, « Поли (А) -специфическая рибонуклеаза опосредует обрезание 3′-концов микроРНК предшественников, расщепленных аргонавтом-2. » , Cell Rep. , т. 5, п о 3,2013, стр. 715-26 ( PMID 24209750 , PMCID 3856240 , DOI 10.1016 / j.celrep.2013.09.029 , читать онлайн [PDF] , по состоянию на 7 апреля 2020 г. ) редактировать
- ↑ (in) Okamura K, Phillips MD, DM Tyler, Duan H, Chou and YT Lai EC, « Активность регуляторных видов микроРНК * HAS оказывает существенное влияние на бизнес микроРНК и 3 ‘UTR Evolution. » , Nat Struct Mol Biol. , т. 15, п о 4,2008 г., стр. 354-63 ( PMID 18376413 , DOI 10.1038 / nsmb.1409 , читать онлайн , по состоянию на 17 марта 2020 г. ) редактировать
-
↑ (in) Хворова А., А. и Рейнольдс Джаясена С.Д., « Функциональные миРНК и миРНК демонстрируют смещение цепи. ” , Cell , vol. 115, п о 22003 г., стр. 209-16 ( PMID 14567918 , DOI 10.1016 / s0092-8674 (03) 00801-8 , читать онлайн [PDF] , по состоянию на 23 июня 2020 г. ) редактировать
-
↑ (in) Ghildiyal M, Xu J, Seitz M, Weng Z и Zamore PD., « Сортировка малых РНК сайленсинга дрозофилы оценивает * нити микроРНК в пути интерференции РНК. » , РНК , т. 16, п о 1,2010 г., стр. 43-56 ( PMID 19917635 , PMCID 2802036 , DOI 10.1261 / rna.1972910 , читать онлайн [PDF] , по состоянию на 23 июня 2020 г. ) редактировать
- ↑ (ru) Грубер А.Р., Лоренц Р., Бернхарт С.Х. и Р. Нойбек Хофакер И.Л., « Веб-сайт Венской РНК. ” , Nucleic Acids Res. , т. 36, п о выпуске веб — сервера,2008 г., W70-4 ( PMID 18424795 , PMCID PMC2447809 , DOI 10.1093 / nar / gkn188 , читать онлайн [PDF] , по состоянию на 2 апреля 2020 г. ) редактировать
- ↑ (ru) Бродерсен П., Сакварелидзе-Ачард Л., Бруун-Расмуссен М., П. Дюнуайер, Ямамото Ю.Ю., Зибурт Л. и Воиннет О. « Широко распространенное ингибирование трансляции миРНК и миРНК растений. » , Наука , т. 320, п о 5880,2008 г., стр. 1185-90 ( PMID 18483398 , DOI 10.1126 / science.1159151 , читать онлайн , по состоянию на 18 марта 2020 г. ) редактировать
- ↑ (in) Khraiwesh B. MA Arif, Seumel IM, Ossowski S, D Weigel, Reski R и W. Frank, « Транскрипционный контроль экспрессии генов с помощью микроРНК. ” , Cell , vol. 140, п о 1,2010 г., стр. 111-22 ( PMID 20085706 , DOI 10.1016 / j.cell.2009.12.023 , читать онлайн , по состоянию на 18 марта 2020 г. ) редактировать
- ↑ (in) Баскервиль и Бартель Д.П. С., « Профилирование микромассивов микроРНК выявляет частую коэкспрессию с соседними миРНК и генами хозяина. » , РНК. , т. 11, п о 3,2005 г., стр. 241-7 ( PMID 15701730 , PMCID PMC1370713 , DOI 10.1261 / rna.7240905 , читать онлайн [PDF] , по состоянию на 25 марта 2020 г. ) редактировать
- ↑ (in) Сайед Д. и г-н Абделлатиф, « МикроРНК в развитии и болезни. » , Physiol Rev. , т. 91, п о 3,2011 г., стр. 827-87 ( PMID 21742789 , DOI 10.1152 / Physrev.00006.2010 , читать онлайн , по состоянию на 20 марта 2020 г. ) редактировать
- ↑ (in) Буэно Малумбрес Дж. И М., « МикроРНК и клеточный цикл. » , Biochim Biophys Acta. , т. 1812 п о 5,2011 г., стр. 592-601 ( PMID 21315819 , DOI 10.1016 / j.bbadis.2011.02.002 , читать онлайн , по состоянию на 20 марта 2020 г. ) редактировать
- ↑ (in) Айви К. Н. Шривастава и Д. « МикроРНК как регуляторы дифференцировки и решений клеточной судьбы. « , Cell Stem Cell , vol. 7, п о 1,2010 г., стр. 36-41 ( PMID 20621048 , DOI 10.1016 / j.stem.2010.06.012 , читать онлайн , по состоянию на 18 марта 2020 г. ) редактировать
- ↑ (in) Йованович М. и Хенгартнер М.О. « MiRNAs и апоптоз: РНК, за которые стоит умереть. » , Онкоген. , т. 25, п о 46,2006 г., стр. 6176-87 ( PMID 17028597 , DOI 10.1038 / sj.onc.1209912 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ Для обзора см .: (en) Rottiers V и Näär AM., « МикроРНК в метаболизме и метаболических нарушениях. « , Nat Rev Mol Cell Biol. , т. 13, п о 4,2012 г., стр. 239-50 ( PMID 22436747 , PMCID PMC4021399 , DOI 10.1038 / nrm3313 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ Дейл Первес, Джордж Дж. Августин, Дэвид Фицпатрик, Уильям С. Холл, Энтони-Самуэль Ламантия, Джеймс О. Макнамара и С. Марк Уильямс, Неврология , Брюссель, Университет Де Бока , колл. «Неврология и познание»,2005 г., 3 е изд. , 811 с. ( ISBN 978-2-8041-4797-6 , читать онлайн ) , гл. 24 («Пластичность синапсов и цепей взрослых»), стр. 575-610.
- ↑ (in) Кандел Э. Р. « Молекулярная биология хранения памяти: диалог энтре генов и синапсов. » , Наука. , т. 294, п о 5544,2001 г., стр. 1030-8 ( PMID 11691980 , DOI 10.1126 / science.1067020 , читать онлайн , по состоянию на 24 марта 2020 г. ) редактировать
-
↑ a b и c (ru) Саттон М.А. и Шуман Е.М., « Синтез дендритных белков, синаптическая пластичность и память. ” , Cell , vol. 127, п о 1,2006 г., стр. 49-58 ( PMID 17018276 , DOI 10.1016 / j.cell.2006.09.014 , читать онлайн [PDF] , по состоянию на 12 апреля 2020 г. ) редактировать
-
↑ (in) Martin KC Casadio A, Zhu H, Yaping E, Rose JC, Chen M, Bailey CH и Kandel ER. « Синапс-специфичное, долгосрочное облегчение сенсорной аплизии к моторным синапсам: функция локального синтеза белка в хранение в памяти. ” , Cell , vol. 91, п о 7,1997 г., стр. 927-38 ( PMID 9428516 , DOI 10.1016 / s0092-8674 (00) 80484-5 , читать онлайн [PDF] , по состоянию на 12 апреля 2020 г. ) редактировать
-
↑ (in) Casadio A, Martin KC, Giustetto M, Zhu M, Chen M, Bartsch D, Bailey CH и Kandel ER., « Временная, охватывающая все нейроны форма CREB-опосредованного длительного облегчения может быть стабилизирована в определенных синапсах. путем местного синтеза белка. ” , Cell , vol. 99, п о 21999 г., стр. 221-37 ( PMID 10535740 , DOI 10.1016 / s0092-8674 (00) 81653-0 , читать онлайн [PDF] , по состоянию на 12 апреля 2020 г. ) редактировать
- ↑ (in) Бивер А., Донлин П.Г.-Асп и Шуман Е.М., « Предпосылки трансляции в нейрональных процессах. « , Curr Opin Neurobiol. , т. 57,2019 г., стр. 141-148 ( PMID 30861464 , DOI 10.1016 / j.conb.2019.02.008 , читать онлайн , по состоянию на 12 апреля 2020 г. ) редактировать
- ↑ (in) Шратт Г., « МикроРНК в синапсе. » , Nat Rev Neurosci. , т. 10, п о 12,2009 г., стр. 842-9 ( PMID 19888283 , DOI 10.1038 / nrn2763 , читать онлайн , по состоянию на 12 апреля 2020 г. ) редактировать
-
↑ a и b (ru) Ашраф С.И., МакЛун А.Л., Sclarsic SM и Kunes S., « Синтез синаптического белка, связанный с памятью, регулируется путем RISC у дрозофилы. ” , Cell , vol. 124, п о 1,2006 г., стр. 191-205 ( PMID 16413491 , DOI 10.1016 / j.cell.2005.12.017 , читать онлайн [PDF] , по состоянию на 12 апреля 2020 г. ) редактировать
-
↑ a и b (ru) Пак Эй Джей, Хавекес Р., Фу Х, Хансен Р., Тюдор Дж. К., Пейшото Л., Ли З., Ву Ю. К., Поплавски С. Г., Барабан Дж. М. и Абель Т., « Обучение индуцирует комплекс транслин / тракс РНКазы для экспрессии рецепторов активина для постоянной памяти. » , Элиф , т. 6,2017 г., e27872 ( PMID 28927503 , PMCID PMC5606845 , DOI 10.7554 / eLife.27872 , читать онлайн [EPUB] , по состоянию на 14 апреля 2020 г. ) редактировать
- ↑ (in) Хегде А. Н. Смит и С. Г., « Последние разработки в области регуляции транскрипции и трансляции, лежащие в основе долговременной синаптической пластичности и памяти. » , Learn Mem. , т. 26, п о 9,2019 г., стр. 307-317 ( PMID 31416904 , PMCID PMC6699410 , DOI 10.1101 / lm.048769.118 , читать онлайн , по состоянию на 12 апреля 2020 г. ) редактировать
- ↑ (in) Ашраф И.Ф. и Кунес С., « След молчания: память и микроРНК в синапсе. « , Curr Opin Neurobiol. , т. 16, п о 5,2006 г., стр. 535-9 ( PMID 16962314 , DOI 10.1016 / j.conb.2006.08.007 , читать онлайн , по состоянию на 12 апреля 2020 г. ) редактировать
- ↑ (ru) White-Grindley If E and K. « RISC there Memories. ” , Cell , vol. 124, п о 1,2006 г., стр. 23-6 ( PMID 16413478 , DOI 10.1016 / j.cell.2005.12.027 , читать онлайн [PDF] , по состоянию на 15 апреля 2020 г. ) редактировать
- ↑ (in) Талли и Куинн Т. В., « Классическое кондиционирование и удержание у нормальных и мутантных Drosophila melanogaster. » , J. Comp Physiol A. , т. 157, п о 21985 г., стр. 263-77 ( PMID 3939242 , DOI 10.1007 / bf01350033 , читать онлайн , по состоянию на 15 апреля 2020 г. ) редактировать
- ↑ (en) Job C и Eberwine J., « Локализация и трансляция мРНК в дендритах и аксонах. ” , Nat Rev Neurosci. , т. 2, п о 12,2001 г., стр. 889-98 ( PMID 11733796 , DOI 10.1038 / 35104069 , читать онлайн , по состоянию на 15 апреля 2020 г. ) редактировать
- ↑ (in) Черн Й Дог Т., Фу Х, А. П. Шах, Абель Т. и Барабан Дж. М. « Трэкс: универсальный сигнальный белок играет ключевую роль в синаптической пластичности и репарации ДНК. » , Neurobiol Learn Mem. , т. 159,2019 г., стр. 46-51 ( PMID 30017897 , PMCID PMC6329659 , DOI 10.1016 / j.nlm.2018.07.003 , читать онлайн [PDF] , по состоянию на 12 апреля 2020 г. ) редактировать
- ↑ Для обзора см .: (ru) Сааб Б.Дж. и Мансуй И.М., « Нейроэпигенетика формирования и нарушения памяти: роль микроРНК. » , Нейрофармакология , т. 80,2014 г., стр. 61-9 ( PMID 24486712 , DOI 10.1016 / j.neuropharm.2014.01.026 , читать онлайн , по состоянию на 16 марта 2020 г. ) редактировать
-
↑ a b и c (en) Rajasethupathy P, Fiumara F, Sheridan R, Betel D, Puthanveettil SV, Russo JJ, Sander C, Tuschl T. и Kandel E., « Характеристика малых РНК в Aplysia выявляет роль miR-124. в ограничении синаптической пластичности посредством CREB. » , Нейрон , т. 63, п о 6,2009 г., стр. 803-17 ( PMID 19778509 , PMCID PMC2875683 , DOI 10.1016 / j.neuron.2009.05.029 , читать онлайн [PDF] , по состоянию на 12 апреля 2020 г. ) редактировать
- ↑ (ен) Лин Q, Вэй Вт, Коэльо СМ, Ли Х, Бейкер-Андрезен Д, Дадли К, Ratnu В.С., Босковича Z Kobor М.С., вс Ю.Е. и Бреды TW., » Мозг специфические микроРНК микроРНК-128b Регулирует формирование памяти о страхе-угасании. » , Nat Neurosci. , т. 14, п о 9,2011 г., стр. 1115-7 ( PMID 21841775 , DOI 10.1038 / nn.2891 , читать онлайн , по состоянию на 12 апреля 2020 г. ) редактировать
- ↑ (ru) Эдбауэр Д., Нейлсон Дж. Р., Фостер К. А., Ван К. Ф., Зеебург Д. П. Баттертон М. Н., Тада Т., Долан Б. М., Шарп П. А. и Шенг М., « Регулирование синаптической структуры и функции с помощью FMRP-ассоциированных микроРНК miR-125b. и miR-132. » , Нейрон , т. 65, п о 3,2010 г., стр. 373-84 ( PMID 20159450 , PMCID PMC5018398 , DOI 0.1016 / j.neuron.2010.01.005 , читать онлайн [PDF] , по состоянию на 12 апреля 2020 г. ) редактировать
- ↑ (in) Гао Дж., Ван В.Й., Мао Ю.В., Графф Дж. Гуан Дж.С., Пан Л., Мак Дж., Ким Д., Су СК и Цай Л.Х., « Новый путь регулирует память и пластичность с помощью SIRT1 и miR-134. » , Природа , т. 466, п о 7310,2010 г., стр. 1105-9 ( PMID 20622856 , PMCID PMC2928875 , DOI 10.1038 / nature09271 , читать онлайн [PDF] , по состоянию на 12 апреля 2020 г. ) редактировать
- ↑ a b и c (ru) Родригес-Ортис CJ, Прието GA, Martini AC, Forner S, Trujillo-Estrada L, LaFerla FM, Baglietto-Vargas D, Cotman CW и Kitazawa M., « miR-181a отрицательно модулирует синаптическую пластичность. в культурах гиппокампа и его ингибирование восстанавливает дефицит памяти на мышиной модели болезни Альцгеймера. » , Ячейка старения , т. 19, п о 3,2020 г., e13118 ( PMID 32087004 , PMCID PMC7059142 , DOI 10.1111 / acel.13118 , читать онлайн , по состоянию на 12 апреля 2020 г. ) редактировать
- ↑ (in) Григгс EM, EJ Young, Rumbaugh G и Miller CA., « MicroRNA-182 регулирует формирование памяти, зависящей от миндалины. » , J. Neurosci. , т. 33, п о 4,2013, стр. 1734-40 ( PMID 23345246 , PMCID PMC3711533 , DOI 10.1523 / JNEUROSCI.2873-12.2013 , читать онлайн [PDF] , по состоянию на 12 апреля 2020 г. ) редактировать
-
↑ (in) Фишбах и С.Дж. Кэрью Т.Дж., « МикроРНК в обработке памяти. » , Нейрон , т. 63, п о 6,2009 г., стр. 714-6 ( PMID 19778498 , DOI 10.1016 / j.neuron.2009.09.007 , читать онлайн [PDF] , по состоянию на 30 апреля 2020 г. ) редактировать
- ↑ (ru) Самбандан С., Акбалык Г., Кочен Л., Ринне Дж., Дж. Кальштатт, Глок С. Тушев Г., Альварес-Кастелао Б., А. и Хекель Шуман Е. М., « Зависимое от активности созревание пространственно локализованной миРНК в нейрональных дендритах. » , Наука , т. 355, п о 6325,2017 г., стр. 634-637 ( PMID 28183980 , DOI 10.1126 / science.aaf8995 , читать онлайн [PDF] , по состоянию на 23 мая 2020 г. ) редактировать
- ↑ Александр Муро, « Плащ-невидимка для глутамата » , на insb.cnrs.fr ,12 марта 2020 г.(просмотрено 23 мая 2020 г. ) .
- ↑ (in) Энн-Мари С. Ходж, « Пища, которую мы едим, может контролировать наши гены — ученые находят микроРНК риса внутри клеток человека » , Scientific American,2011 г.(по состоянию на 14 марта 2020 г. )
- ↑ (ru) Zhang L et al. , « Экзогенное растение MIR168a специфически нацелено на LDLRAP1 млекопитающих: доказательство перекрестной регуляции царства с помощью микроРНК. « , Cell Res. , т. 22, п о 1,2012 г., стр. 107-26 ( PMID 21931358 , DOI 10.1038 / cr.2011.158 , читать онлайн , по состоянию на 20 марта 2020 г. ) редактировать
- ↑ Элейн Н. Мариеб и Катя Хоэн ( пер. С англ.), Анатомия и физиология человека , Монреаль, Пирсон Прентис Холл,2014 г., 9- е изд. , 1291 с. ( ISBN 978-2-7613-6932-9 , читать онлайн ) , стр. 1052
- ↑ (in) Брент Дикинсон, Юаньцзи Чжан, Джей Петрик С. Грегори Хек, Сергей Ивашута и Уильям С. Маршалл, « Недостаток детектируемой пероральной биодоступности растительных микроРНК после кормления у мышей » , Nature Biotechnology , Vol. 31, п о 11,2013, стр. 965-7 ( PMID 24213763 , DOI 10.1038 / nbt.2737 , читать онлайн , по состоянию на 14 марта 2020 г. ) редактировать
- ↑ (in) Тосар Дж. П., Ровира С., Х. и Найя Кайота А., « Анализ данных секвенирования общедоступных баз данных подтверждает недиетическое происхождение иностранных предполагаемых миРНК: недооцененные эффекты контаминации в Новом Южном Уэльсе. » , РНК , т. 20, п о 6,2014 г., стр. 754-7 ( PMID 24729469 , PMCID PMC4024629 , DOI 10.1261 / rna.044263.114 , читать онлайн [PDF] , по состоянию на 3 апреля 2020 г. ) редактировать
- ↑ Обзорная статья о различиях между теориями Дарвина и Ламарка, а также о влиянии эпигенетики: Жан-Клод БРЕГЛИАНО, « Ламарк и Дарвин: два разных видения живого мира » , в Энциклопедии окружающей среды ,4 марта 2020 г.(по состоянию на 4 апреля 2020 г. ) .
- ↑ (in) Яо Q, Чен Y и X. Чжоу, » Роль микроРНК в эпигенетической регуляции. « , Curr Opin Chem Biol. , т. 51,2019 г., стр. 11-17 ( PMID 30825741 , DOI 10.1016 / j.cbpa.2019.01.024 , читать онлайн , по состоянию на 4 апреля 2020 г. ) редактировать
- ↑ (in) Чан С.Ю., Чжан Ю.Й., Хеманн К. Махони, Э. К. Цвейер Дж. Л. и Дж. Лоскальцо, « МикроРНК-210 контролирует митохондриальный метаболизм во время гипоксии путем подавления белков сборки кластера железа и серы ISCU1 / 2. « , Cell Metab. , т. 10, п о 4,2009 г., стр. 273-84 ( PMID 19808020 , DOI 10.1016 / j.cmet.2009.08.015 , читать онлайн , по состоянию на 18 марта 2020 г. ) редактировать
- ↑ (in) Шринивасан Х. и С. Дас, « Митохондриальная миРНК (MitomiR): новый игрок в сердечно-сосудистой системе. « , Can J Physiol Pharmacol. , т. 93, п о 10,2015 г., стр. 855-61 ( PMID 26030833 , DOI 10.1139 / cjpp-2014-0500 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ (in) Bandiera S Rüberg S, M Girard, Dodger N, Hanein S, D Christian, Munnich A, Lyonnet S и A. Henrion-Caude, » Ядерный аутсорсинг компонентов РНК-интерференции в митохондриях человека. » , PLoS One. , т. 6, п о 6,2011 г., e20746 ( PMID 21695135 , DOI 10.1371 / journal.pone.0020746 , читать онлайн , по состоянию на 18 марта 2020 г. ) редактировать
- ↑ (ru) Чжан X, Цзо X, Ян Би, Ли З, Сюэ Y, Чжоу Y, Хуан Дж, Чжао X, Чжоу Дж, Ян И, Чжан Х, Го П, Сунь Х, Го Л., Чжан И и Фу XD., « МикроРНК напрямую усиливает митохондриальную трансляцию во время дифференцировки мышц. « , Cell. , т. 158, п о 3,2014 г., стр. 607-19 ( PMID 25083871 , DOI 10.1016 / j.cell.2014.05.047 , читать онлайн , по состоянию на 18 марта 2020 г. ) редактировать
- ↑ Для обзора см .: (в) Гейгер Дж. И Далгаард LT. « Взаимодействие митохондриального метаболизма и микроРНК. « , Cell Mol Life Sci. , т. 74, п о 4,2017 г., стр. 631-646 ( PMID 27563705 , DOI 10.1007 / s00018-016-2342-7 , читать онлайн , по состоянию на 18 марта 2020 г. ) редактировать
- ↑ (in) Bandiera S Matégot R, Girard M, J и Demongeot Henrion-Caude A., « MitomiRs, определяющие внутриклеточную локализацию микроРНК, делали митохондрии. » , Free Radic Biol Med. , т. 64,2013, стр. 12-9 ( PMID 23792138 , DOI 10.1016 / j.freeradbiomed.2013.06.013 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ Александра Хенрион-Код , Симонетта Бандиера и Станислас Лионнет , Методы регулировки экспрессии митохондриального генома с помощью Микрорны ,7 декабря 2012 г.( читать онлайн )
- ↑ (in) Пфеффер С. и О. Воиннет, « Вирусы, микроРНК и рак. » , Онкоген. , т. 25, п о 46,2006 г., стр. 6211-9 ( PMID 17028601 , DOI 10.1038 / sj.onc.1209915 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ (in) Жирар М., Жакемин Э, Мюнхен А., Лионне С. и А. Генрион-Код, « miR-122, парадигма роли микроРНК в печени. » , J Hepatol. , т. 48, п о 4,2008 г., стр. 648-56 ( PMID 18291553 , DOI 10.1016 / j.jhep.2008.01.019 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
-
↑ (in) Колли Д.Г., Бастиндуй А.Л., Секор В.Е. Кинг и Ч., « Человеческий шистосомоз. » , Ланцет , т. 383, п о 9936,2014 г., стр. 2253-64 ( PMID 24698483 , PMCID PMC4672382 , DOI 10.1016 / S0140-6736 (13) 61949-2 , читать онлайн [PDF] , по состоянию на 28 апреля 2020 г. ) редактировать
-
↑ (ru) Лю Дж., Чжу Л., Ван Дж., Цю Л., Чен Й, Дэвис Р. Э. и Г. Ченг, « Груз внеклеточных пузырьков Schistosoma japonicum Регулирует функции миРНК-хозяина макрофагов, облегчая паразитизм. » , PLoS Pathog. , т. 15, п о 6,2019 г., e1007817 ( PMID 31163079 , PMCID PMC6548406 , DOI 10.1371 / journal.ppat.1007817 , читать онлайн [PDF] , по состоянию на 28 апреля 2020 г. ) редактировать
-
↑ (in) Ткач М. и К. Тери, « Связь внеклеточных пузырьков. Где мы находимся и куда нам нужно идти » , Cell , vol. 164, п о 6,2016 г., стр. 1226-1232 ( PMID 26967288 , DOI 10.1016 / j.cell.2016.01.043 , читать онлайн [PDF] , по состоянию на 29 апреля 2020 г. ) редактировать
- ↑ (ru) Ван Г.К., Дж.К. Чжу, Чжан Дж. Т., Ли Кью, Ли Й, Хэ Дж, Цинь Ю. В. и Цзин К. « Циркулирующая микроРНК: новый потенциальный биомаркер для ранней диагностики острого инфаркта миокарда у людей. ” , Eur Heart J. , vol. 31, п о 6,2010 г., стр. 659-66 ( PMID 20159880 , DOI 10.1093 / eurheartj / ehq013 , читать онлайн , по состоянию на 18 марта 2020 г. ) редактировать
- ↑ a b c и d (ru) Эрнандес-Рапп Дж., Райноне С. и Эбер С. С., « МикроРНК, лежащие в основе дефицита памяти при нейродегенеративных расстройствах. » , Prog Neuropsychopharmacol Biol Psychiatry. , т. 73,2017 г., стр. 79-86 ( PMID 27117821 , DOI 10.1016 / j.pnpbp.2016.04.011 , читать онлайн , по состоянию на 13 апреля 2020 г. ) редактировать
- ↑ (in) Субодх Кумар Редди и П. Хемачандра, « Являются ли циркулирующие микроРНК периферическими биомаркерами болезни Альцгеймера? » , Biochim Biophys Acta , т. +1862 п о 9,2016 г., стр. 1617-27 ( PMID 27264337 , DOI 10.1016 / j.bbadis.2016.06.001 , читать онлайн , консультация 13 марта 2020 г. ) редактировать
- ↑ (in) Валади Х., К. Экстрём, Боссиос А. Шёстранд М., Ли Дж. Дж. И Лётвалл О. Дж. « Экзосомный перенос мРНК и микроРНК — это новый механизм генетического обмена в клетках. « , Nat Cell Biol. , т. 9, п о 6,2007 г., стр. 654-9 ( PMID 17486113 , DOI 10.1038 / ncb1596 , читать онлайн , по состоянию на 13 апреля 2020 г. ) редактировать
- ↑ (в) Ahmad R Аршад, Siti А Сулейману, Амалия А Saperi Рахман Джамала, Norlinah Мохамед Ибрагим и Нор Azian Абдул Мурад, « MicroRNAs и генов — мишеней в качестве биомаркеров для диагностики раннего начала болезни Паркинсона » , передний Mol Neurosci , полет . 10,2017 г., стр. 352 ( PMID 29163029 , DOI 10.3389 / fnmol.2017.00352 , читать онлайн , по состоянию на 13 марта 2020 г. ) редактировать
- ↑ (in) esquela A-Kerscher and Slack FJ. « Онкомиры — микроРНК, играющие роль в развитии рака. « , Nat Rev Cancer. , т. 6, п о 4,2006 г., стр. 259-69 ( PMID 16557279 , DOI 10.1038 / nrc1840 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ (in) Xiong J, « Новые роли микроРНК-22 в заболеваниях человека и нормальной физиологии. « , Curr Mol Med. , т. 12, п о 3,2012 г., стр. 247-58 ( PMID 22300138 , DOI 10.2174 / 156652412799218886 , читать онлайн , по состоянию на 2 апреля 2020 г. ) редактировать
- ↑ Ладейро Ю. и Цукман-Росси Дж. « [МиРНК в раке: случай опухолей печени]. », Мед. Наука (Париж). , т. 25, п о 5,2009 г., стр. 467-72 ( PMID 19480827 , DOI 10.1051 / medsci / 2009255467 , читать онлайн [PDF] , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ (ru) Ладейро Ю., Куши Дж., Балабауд С. Биулак-П Шалфей, Пеллетье Л., С. и Ребуиссу Зукман-Росси Дж. « Профилирование микроРНК в гепатоцеллюлярных опухолях связано с клиническими особенностями и мутациями гена онкогена / супрессора опухоли. » , Гепатология. , т. 47, п о 6,2008 г., стр. 1955-63 ( PMID 18433021 , DOI 10.1002 / hep.22256 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ (in) Танака М., Оикава К., Таканаши М., Кудо М., Охьяшики Дж., К. и Охьяшики, г-н Курода, « Снижение регуляции miR-92 в плазме человека является новым маркером для пациентов с острым лейкозом. » , PLoS One. , т. 4, п о 5,2009 г., e5532 ( PMID 19440243 , DOI 10.1371 / journal.pone.0005532 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ https://www.futura-sciences.com/sante/actualites/cancer-cerveau-simple-test-urine-permettra-detection-tumeurs-cerebrales-88186/
- ↑ Йотаро Китано и др. , Диагностическая модель на основе микроРНК в моче для опухолей центральной нервной системы с использованием нанопроволочных каркасов , 2021. DOI : 10.1021 / acsami.1c01754
- ↑ (ru) Розвик Н., Момонт М. Дуркин К., Такеда Х., Ф. Каймент, Клютер Й, Вернин С., Мортре Ф., Ваттель Э, Берни А., М. Жорж и Ван ден Брук А., « Глубокое секвенирование выявляет многочисленные неканонические ретровирусные микроРНК. при В-клеточном лейкозе / лимфоме. « , Proc Natl Acad Sci US A. , vol. 110, п о 6,2013, стр. 2306-11 ( PMID 23345446 , DOI 10.1073 / pnas.1213842110 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ (ru) Крюцфельдт Дж., Раевски Н., Брайх Р., Раджив К.Г., Тушл Т., Манохаран М. и Стоффель М., « Отключение микроРНК in vivo с помощью антагомиров. » , Природа. , т. 438, п о 7068,2005 г., стр. 685-9 ( PMID 16258535 , DOI 10.1038 / nature04303 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ (in) Борел Ф., Кей М.А. и К. Мюллер, « Рекомбинантный AAV как платформа для трансляции терапевтического потенциала РНК-интерференции. » , Mol Ther. , т. 22, п о 4,2014 г., стр. 692-701 ( PMID 24352214 , DOI 10.1038 / mt.2013.285 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ (in) Янссен Х.Л., Рисинк Х.В., Лавиц Э.Дж., Зеузем С., Родригес-Торрес М., Патель К., ван дер Меер А. Дж. Патик А. К., Чен А., Чжоу Й., Перссон Р., King Comics, Кауппинен С., Левин А. А. Ходжес М. Р. ., « Лечение инфекции HCV с помощью нацеливания на микроРНК. » , N Engl J Med. , т. 368, п о 18,2013, стр. 1685-94 ( PMID 23534542 , DOI 10.1056 / NEJMoa1209026 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
- ↑ (in) Gomez IG et al. , « Олигонуклеотиды против микроРНК-21 предотвращают прогрессирование нефропатии Альпорта, стимулируя метаболические пути. » , J Clin Invest. , т. 125, п о 1,2015 г., стр. 141-56 ( PMID 25415439 , PMCID PMC4382246 , DOI 10.1172 / JCI75852 , читать онлайн , по состоянию на 19 марта 2020 г. ) редактировать
Смотрите также
Библиография
- Кэролайн Хартманн, Фабьен Корре-Менгуи, Аднан Буалем, Мариана Йованович и Кристин Леландайс-Бриер, «МикроРНК — новый класс регуляторов экспрессии генов», Med Sci (Paris) , 2004, 20, 894–898 ( читать онлайн , доступно 16 марта 2020 г.).
- (en) Альбертс Б., Джонсон А., Льюис Дж., Рафф М., Робертс К. и Уолтер П., Молекулярная биология клетки , 5-е издание, Garland Science, Нью-Йорк, 2007, стр. 493-495 ( ISBN 978-0815341055 ) .
Статьи по Теме
- Рибонуклеиновая кислота
- Список типов РНК
- РНК-интерференция
- Мессенджер рибонуклеиновая кислота (мРНК)
- Передача рибонуклеиновой кислоты (тРНК)
- Посттранскрипционная регуляция
- Эпигенетика
- Список сокращений клеточной и молекулярной биологии
- Виктор Амброс (ru)
- Дэвид Бартель (en)
- Томас Тушль (ru)
Внешние ссылки
- Веб-сайт исследовательской группы CNRS «Системное влияние малых регуляторных РНК» в IGH — UMR9002
- (in) Анимация, объясняющая, как вмешательство РНК
- (ru) Сайт лаборатории Виктора Амброса в Медицинской школе УМАСС
- (ru) Сайт лаборатории Дэвида Бартеля в Массачусетском технологическом институте
- (ru) Сайт лаборатории Томаса Тушля в Рокфеллеровском университете
- (ru) База данных miRBase
|
в · м Нуклеиновые кислоты |
|||||||
|---|---|---|---|---|---|---|---|
| Нуклеиновые основания |
|
||||||
| Нуклеозиды |
|
||||||
| Нуклеотиды |
|
||||||
| Подобные не рибозиды |
|
||||||
| Клонирование векторов |
|
||||||
|